+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-7939 | |||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Integrin alpha-v beta-8 in complex with the Fabs 8B8 and 68 | |||||||||||||||||||||
 マップデータ マップデータ | Headpiece map, 4.8 A resolution, sharpened with a B-factor of -144 | |||||||||||||||||||||
 試料 試料 |
| |||||||||||||||||||||
 キーワード キーワード | GLYCOPROTEIN / MEMBRANE PROTEIN / ADHESION / FAB | |||||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報ganglioside metabolic process / Langerhans cell differentiation / integrin alphav-beta8 complex / integrin alphav-beta6 complex / transforming growth factor beta production / negative regulation of entry of bacterium into host cell / integrin alphav-beta5 complex / opsonin binding / integrin alphav-beta1 complex / Cross-presentation of particulate exogenous antigens (phagosomes) ...ganglioside metabolic process / Langerhans cell differentiation / integrin alphav-beta8 complex / integrin alphav-beta6 complex / transforming growth factor beta production / negative regulation of entry of bacterium into host cell / integrin alphav-beta5 complex / opsonin binding / integrin alphav-beta1 complex / Cross-presentation of particulate exogenous antigens (phagosomes) / extracellular matrix protein binding / placenta blood vessel development / Laminin interactions / integrin alphav-beta3 complex / negative regulation of lipoprotein metabolic process / alphav-beta3 integrin-PKCalpha complex / entry into host cell by a symbiont-containing vacuole / alphav-beta3 integrin-HMGB1 complex / negative regulation of lipid transport / hard palate development / regulation of phagocytosis / Elastic fibre formation / alphav-beta3 integrin-IGF-1-IGF1R complex / transforming growth factor beta binding / positive regulation of small GTPase mediated signal transduction / filopodium membrane / extracellular matrix binding / cartilage development / negative regulation of low-density lipoprotein particle clearance / apolipoprotein A-I-mediated signaling pathway / apoptotic cell clearance / wound healing, spreading of epidermal cells / integrin complex / heterotypic cell-cell adhesion / Molecules associated with elastic fibres / negative chemotaxis / Mechanical load activates signaling by PIEZO1 and integrins in osteocytes / Syndecan interactions / cell adhesion mediated by integrin / positive regulation of osteoblast proliferation / microvillus membrane / cell-substrate adhesion / endodermal cell differentiation / PECAM1 interactions / TGF-beta receptor signaling activates SMADs / positive regulation of intracellular signal transduction / fibronectin binding / lamellipodium membrane / negative regulation of macrophage derived foam cell differentiation / negative regulation of lipid storage / ECM proteoglycans / Integrin cell surface interactions / vasculogenesis / voltage-gated calcium channel activity / specific granule membrane / coreceptor activity / phagocytic vesicle / ERK1 and ERK2 cascade / extrinsic apoptotic signaling pathway in absence of ligand / substrate adhesion-dependent cell spreading / positive regulation of cell adhesion / transforming growth factor beta receptor signaling pathway / protein kinase C binding / Turbulent (oscillatory, disturbed) flow shear stress activates signaling by PIEZO1 and integrins in endothelial cells / cell-matrix adhesion / Signal transduction by L1 / integrin-mediated signaling pathway / negative regulation of extrinsic apoptotic signaling pathway / cell-cell adhesion / calcium ion transmembrane transport / VEGFA-VEGFR2 Pathway / response to virus / ruffle membrane / integrin binding / positive regulation of angiogenesis / cell migration / positive regulation of cytosolic calcium ion concentration / virus receptor activity / protease binding / angiogenesis / cell adhesion / immune response / positive regulation of cell migration / negative regulation of gene expression / external side of plasma membrane / focal adhesion / positive regulation of cell population proliferation / Neutrophil degranulation / positive regulation of gene expression / symbiont entry into host cell / cell surface / extracellular exosome / metal ion binding / membrane / plasma membrane / cytosol 類似検索 - 分子機能 | |||||||||||||||||||||
| 生物種 |  Homo sapiens (ヒト) / Homo sapiens (ヒト) /  | |||||||||||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.8 Å | |||||||||||||||||||||
 データ登録者 データ登録者 | Cormier A / Campbell MG | |||||||||||||||||||||
| 資金援助 |  米国, 6件 米国, 6件
| |||||||||||||||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2018 ジャーナル: Nat Struct Mol Biol / 年: 2018タイトル: Cryo-EM structure of the αvβ8 integrin reveals a mechanism for stabilizing integrin extension. 著者: Anthony Cormier / Melody G Campbell / Saburo Ito / Shenping Wu / Jianlong Lou / James Marks / Jody L Baron / Stephen L Nishimura / Yifan Cheng /  要旨: Integrins are conformationally flexible cell surface receptors that survey the extracellular environment for their cognate ligands. Interactions with ligands are thought to be linked to global ...Integrins are conformationally flexible cell surface receptors that survey the extracellular environment for their cognate ligands. Interactions with ligands are thought to be linked to global structural rearrangements involving transitions between bent, extended-closed and extended-open forms. Thus far, structural details are lacking for integrins in the extended conformations due to extensive flexibility between the headpiece and legs in this conformation. Here we present single-particle electron cryomicroscopy structures of human αvβ8 integrin in the extended-closed conformation, which has been considered to be a low-affinity intermediate. Our structures show the headpiece rotating about a flexible αv knee, suggesting a ligand surveillance mechanism for integrins in their extended-closed form. Our model predicts that the extended conformation is mainly stabilized by an interface formed between flexible loops in the upper and lower domains of the αv leg. Confirming these findings with the αvβ3 integrin suggests that our model of stabilizing the extended-closed conformation is generalizable to other integrins. | |||||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_7939.map.gz emd_7939.map.gz | 2.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-7939-v30.xml emd-7939-v30.xml emd-7939.xml emd-7939.xml | 53.6 KB 53.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_7939.png emd_7939.png | 104.3 KB | ||
| Filedesc metadata |  emd-7939.cif.gz emd-7939.cif.gz | 10.3 KB | ||
| その他 |  emd_7939_additional_1.map.gz emd_7939_additional_1.map.gz emd_7939_additional_10.map.gz emd_7939_additional_10.map.gz emd_7939_additional_11.map.gz emd_7939_additional_11.map.gz emd_7939_additional_2.map.gz emd_7939_additional_2.map.gz emd_7939_additional_3.map.gz emd_7939_additional_3.map.gz emd_7939_additional_4.map.gz emd_7939_additional_4.map.gz emd_7939_additional_5.map.gz emd_7939_additional_5.map.gz emd_7939_additional_6.map.gz emd_7939_additional_6.map.gz emd_7939_additional_7.map.gz emd_7939_additional_7.map.gz emd_7939_additional_8.map.gz emd_7939_additional_8.map.gz emd_7939_additional_9.map.gz emd_7939_additional_9.map.gz | 59.2 MB 12.1 MB 12.1 MB 59.4 MB 59.3 MB 59.2 MB 2.7 MB 56.6 MB 2.4 MB 14.6 MB 14.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-7939 http://ftp.pdbj.org/pub/emdb/structures/EMD-7939 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-7939 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-7939 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_7939_validation.pdf.gz emd_7939_validation.pdf.gz | 315.2 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_7939_full_validation.pdf.gz emd_7939_full_validation.pdf.gz | 314.8 KB | 表示 | |
| XML形式データ |  emd_7939_validation.xml.gz emd_7939_validation.xml.gz | 6.2 KB | 表示 | |
| CIF形式データ |  emd_7939_validation.cif.gz emd_7939_validation.cif.gz | 7.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7939 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7939 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7939 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7939 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_7939.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_7939.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Headpiece map, 4.8 A resolution, sharpened with a B-factor of -144 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.67 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
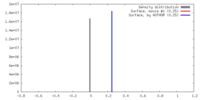
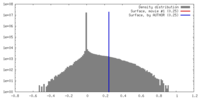
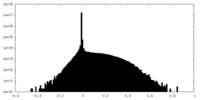
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
+追加マップ: Conformation V, sharpened with a B-factor of -300
+追加マップ: Half-map 01 for headpiece map
+追加マップ: Half-map 02 for headpiece map
+追加マップ: Conformation I, sharpened with a B-factor of -300
+追加マップ: Conformation II, sharpened with a B-factor of -300
+追加マップ: Conformation III, sharpened with a B-factor of -300
+追加マップ: Conformation IV, sharpened with a B-factor of -300
+追加マップ: Conformation VI, sharpened with a B-factor of -300
+追加マップ: Full map, 6.4 A resolution, sharpened with a B-factor of -146
+追加マップ: Half-map 01 for full map
+追加マップ: Half-map 02 for full map
- 試料の構成要素
試料の構成要素
-全体 : Alpha-v Beta-8 Integrin in complex with the Fabs 68 and 8B8
| 全体 | 名称: Alpha-v Beta-8 Integrin in complex with the Fabs 68 and 8B8 |
|---|---|
| 要素 |
|
-超分子 #1: Alpha-v Beta-8 Integrin in complex with the Fabs 68 and 8B8
| 超分子 | 名称: Alpha-v Beta-8 Integrin in complex with the Fabs 68 and 8B8 タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#6 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 312 KDa |
-分子 #1: Integrin alpha-v
| 分子 | 名称: Integrin alpha-v / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 106.758266 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: FNLDVDSPAE YSGPEGSYFG FAVDFFVPSA SSRMFLLVGA PKANTTQPGI VEGGQVLKCD WSSTRRCQPI EFDATGNRDY AKDDPLEFK SHQWFGASVR SKQDKILACA PLYHWRTEMK QEREPVGTCF LQDGTKTVEY APCRSQDIDA DGQGFCQGGF S IDFTKADR ...文字列: FNLDVDSPAE YSGPEGSYFG FAVDFFVPSA SSRMFLLVGA PKANTTQPGI VEGGQVLKCD WSSTRRCQPI EFDATGNRDY AKDDPLEFK SHQWFGASVR SKQDKILACA PLYHWRTEMK QEREPVGTCF LQDGTKTVEY APCRSQDIDA DGQGFCQGGF S IDFTKADR VLLGGPGSFY WQGQLISDQV AEIVSKYDPN VYSIKYNNQL ATRTAQAIFD DSYLGYSVAV GDFNGDGIDD FV SGVPRAA RTLGMVYIYD GKNMSSLYNF TGEQMAAYFG FSVAATDING DDYADVFIGA PLFMDRGSDG KLQEVGQVSV SLQ RASGDF QTTKLNGFEV FARFGSAIAP LGDLDQDGFN DIAIAAPYGG EDKKGIVYIF NGRSTGLNAV PSQILEGQWA ARSM PPSFG YSMKGATDID KNGYPDLIVG AFGVDRAILY RARPVITVNA GLEVYPSILN QDNKTCSLPG TALKVSCFNV RFCLK ADGK GVLPRKLNFQ VELLLDKLKQ KGAIRRALFL YSRSPSHSKN MTISRGGLMQ CEELIAYLRD ESEFRDKLTP ITIFME YRL DYRTAADTTG LQPILNQFTP ANISRQAHIL LDCGEDNVCK PKLEVSVDSD QKKIYIGDDN PLTLIVKAQN QGEGAYE AE LIVSIPLQAD FIGVVRNNEA LARLSCAFKT ENQTRQVVCD LGNPMKAGTQ LLAGLRFSVH QQSEMDTSVK FDLQIQSS N LFDKVSPVVS HKVDLAVLAA VEIRGVSSPD HVFLPIPNWE HKENPETEED VGPVVQHIYE LRNNGPSSFS KAMLHLQWP YKYNNNTLLY ILHYDIDGPM NCTSDMEINP LRIKISSLQT TEKNDTVAGQ GERDHLITKR DLALSEGDIH TLGCGVAQCL KIVCQVGRL DRGKSAILYV KSLLWTETFM NKENQNHSYS LKSSASFNVI EFPYKNLPIE DITNSTLVTT NVTWGIQPAP M PVPVW UniProtKB: Integrin alpha-V |
-分子 #2: Integrin beta-8
| 分子 | 名称: Integrin beta-8 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 70.837234 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: EDNRCASSNA ASCARCLALG PECGWCVQED FISGGSRSER CDIVSNLISK GCSVDSIEYP SVHVIIPTEN EINTQVTPGE VSIQLRPGA EANFMLKVHP LKKYPVDLYY LVDVSASMHN NIEKLNSVGN DLSRKMAFFS RDFRLGFGSY VDKTVSPYIS I HPERIHNQ ...文字列: EDNRCASSNA ASCARCLALG PECGWCVQED FISGGSRSER CDIVSNLISK GCSVDSIEYP SVHVIIPTEN EINTQVTPGE VSIQLRPGA EANFMLKVHP LKKYPVDLYY LVDVSASMHN NIEKLNSVGN DLSRKMAFFS RDFRLGFGSY VDKTVSPYIS I HPERIHNQ CSDYNLDCMP PHGYIHVLSL TENITEFEKA VHRQKISGNI DTPEGGFDAM LQAAVCESHI GWRKEAKRLL LV MTDQTSH LALDSKLAGI VVPNDGNCHL KNNVYVKSTT MEHPSLGQLS EKLIDNNINV IFAVQGKQFH WYKDLLPLLP GTI AGEIES KAANLNNLVV EAYQKLISEV KVQVENQVQG IYFNITAICP DGSRKPGMEG CRNVTSNDEV LFNVTVTMKK CDVT GGKNY AIIKPIGFNE TAKIHIHRNC SCQCEDNRGP KGKCVDETFL DSKCFQCDEN KCHFDEDQFS SESCKSHKDQ PVCSG RGVC VCGKCSCHKI KLGKVYGKYC EKDDFSCPYH HGNLCAGHGE CEAGRCQCFS GWEGDRCQCP SAAAQHCVNS KGQVCS GRG TCVCGRCECT DPRSIGRFCE HCPTCYTACK ENWNCMQCLH PHNLSQAILD QCKTSCALME QQHYVDQTSE CFSSPS UniProtKB: Integrin beta-8 |
-分子 #3: 8B8 heavy chain Fab
| 分子 | 名称: 8B8 heavy chain Fab / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 23.314826 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: EVQLVESGPG LVKPSQSLSL TCSVTGYYIT SSYYWNWIRQ FPGNELEWMG YISYDGSNSY NPSLKNRISI TRDTSKNQFF LKLNSVTTE DIATYFCVRE DYDSFDYWGQ GTTLTVSSAK TTAPSVYPLA PVCGDTTGSS VTLGCLVKGY FPEPVTLTWN S GSLSSGVH ...文字列: EVQLVESGPG LVKPSQSLSL TCSVTGYYIT SSYYWNWIRQ FPGNELEWMG YISYDGSNSY NPSLKNRISI TRDTSKNQFF LKLNSVTTE DIATYFCVRE DYDSFDYWGQ GTTLTVSSAK TTAPSVYPLA PVCGDTTGSS VTLGCLVKGY FPEPVTLTWN S GSLSSGVH TFPAVLQSDL YTLSSSVTVT SSTWPSQSIT CNVAHPASST KVDKK |
-分子 #4: 8B8 light chain Fab
| 分子 | 名称: 8B8 light chain Fab / タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 23.241596 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: EIVLTQSPAI MSAFPGEKVT MTCSASSSVS YIHWYQQKSG TSPKRWIYDT SKLASGVPAR FSGSGSGTSY SLTISSMEAE DAATYYCHQ WTSNPATFGG GTKLEIKAAD AAPTVSIFPP SSEQLTSGGA SVVCFLNNFY PKDINVKWKI DGSERQNGVL N SWTDQDSK ...文字列: EIVLTQSPAI MSAFPGEKVT MTCSASSSVS YIHWYQQKSG TSPKRWIYDT SKLASGVPAR FSGSGSGTSY SLTISSMEAE DAATYYCHQ WTSNPATFGG GTKLEIKAAD AAPTVSIFPP SSEQLTSGGA SVVCFLNNFY PKDINVKWKI DGSERQNGVL N SWTDQDSK DSTYSMSSTL TLTKDEYERH NSYTCEATHK TSTSPIVKSF NRNEC |
-分子 #5: 68 heavy chain Fab
| 分子 | 名称: 68 heavy chain Fab / タイプ: protein_or_peptide / ID: 5 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 22.680303 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: EVQLQQSGAE LMKPGASVKI SCKATGYTFS TYWIEWIKQR PGHGLEWIGD ILPGSGTTNY NEKFKGRATV TADRSSNTAY MQLSSLTSE DSAVYYCARW GWDSYWGQGT LVTVSSASTK GPSVFPLAPS SKSTSGGTAA LGCLVKDYFP EPVTVSWNSG A LTSGVHTF ...文字列: EVQLQQSGAE LMKPGASVKI SCKATGYTFS TYWIEWIKQR PGHGLEWIGD ILPGSGTTNY NEKFKGRATV TADRSSNTAY MQLSSLTSE DSAVYYCARW GWDSYWGQGT LVTVSSASTK GPSVFPLAPS SKSTSGGTAA LGCLVKDYFP EPVTVSWNSG A LTSGVHTF PAVLQSSGLY SLSSVVTVPS SSLGTQTYIC NVNHKPSNTK VDKK |
-分子 #6: 68 light chain Fab
| 分子 | 名称: 68 light chain Fab / タイプ: protein_or_peptide / ID: 6 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 23.420795 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: DIEMTQSPSS LSASLGDRVT ISCSASQGIS NYLNWYQQKP DGTVKLLIYY TSSLHSGVPS RFSGSGSGTD YSLTISNLEP EDIATYYCQ QYSELPYTFG GGTKLEIKRT VAAPSVFIFP PSDEQLKSGT ASVVCLLNNF YPREAKVQWK VDNALQSGNS Q ESVTEQDS ...文字列: DIEMTQSPSS LSASLGDRVT ISCSASQGIS NYLNWYQQKP DGTVKLLIYY TSSLHSGVPS RFSGSGSGTD YSLTISNLEP EDIATYYCQ QYSELPYTFG GGTKLEIKRT VAAPSVFIFP PSDEQLKSGT ASVVCLLNNF YPREAKVQWK VDNALQSGNS Q ESVTEQDS KDSTYSLSST LTLSKADYEK HKVYACEVTH QGLSSPVTKS FNRGEC |
-分子 #10: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 10 / コピー数: 2 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 6.8 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 60 sec. |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 293 K / 装置: FEI VITROBOT MARK III |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI POLARA 300 |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / 撮影したグリッド数: 1 / 実像数: 713 / 平均露光時間: 6.0 sec. / 平均電子線量: 41.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Tecnai Polara / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー






















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)