+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-7770 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Atomic resolution cryo-EM structure of beta-galactosidase | |||||||||
 マップデータ マップデータ | Escherichia coli beta-galactosidase | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | drift correction / radiation damage / drug discovery / precision medicine / computer-aided drug discovery / HYDROLASE | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報alkali metal ion binding / lactose catabolic process / beta-galactosidase complex / beta-galactosidase / beta-galactosidase activity / carbohydrate binding / magnesium ion binding / identical protein binding 類似検索 - 分子機能 | |||||||||
| 生物種 |   | |||||||||
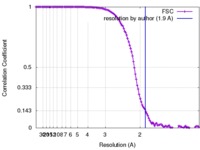
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 1.9 Å | |||||||||
 データ登録者 データ登録者 | Subramaniam S / Bartesaghi A / Banerjee S / Zhu X | |||||||||
 引用 引用 |  ジャーナル: Structure / 年: 2018 ジャーナル: Structure / 年: 2018タイトル: Atomic Resolution Cryo-EM Structure of β-Galactosidase. 著者: Alberto Bartesaghi / Cecilia Aguerrebere / Veronica Falconieri / Soojay Banerjee / Lesley A Earl / Xing Zhu / Nikolaus Grigorieff / Jacqueline L S Milne / Guillermo Sapiro / Xiongwu Wu / Sriram Subramaniam /  要旨: The advent of direct electron detectors has enabled the routine use of single-particle cryo-electron microscopy (EM) approaches to determine structures of a variety of protein complexes at near- ...The advent of direct electron detectors has enabled the routine use of single-particle cryo-electron microscopy (EM) approaches to determine structures of a variety of protein complexes at near-atomic resolution. Here, we report the development of methods to account for local variations in defocus and beam-induced drift, and the implementation of a data-driven dose compensation scheme that significantly improves the extraction of high-resolution information recorded during exposure of the specimen to the electron beam. These advances enable determination of a cryo-EM density map for β-galactosidase bound to the inhibitor phenylethyl β-D-thiogalactopyranoside where the ordered regions are resolved at a level of detail seen in X-ray maps at ∼ 1.5 Å resolution. Using this density map in conjunction with constrained molecular dynamics simulations provides a measure of the local flexibility of the non-covalently bound inhibitor and offers further opportunities for structure-guided inhibitor design. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_7770.map.gz emd_7770.map.gz | 70.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-7770-v30.xml emd-7770-v30.xml emd-7770.xml emd-7770.xml | 21.4 KB 21.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_7770_fsc.xml emd_7770_fsc.xml | 13.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_7770.png emd_7770.png | 197.7 KB | ||
| マスクデータ |  emd_7770_msk_1.map emd_7770_msk_1.map | 147.3 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-7770.cif.gz emd-7770.cif.gz | 6.5 KB | ||
| その他 |  emd_7770_additional.map.gz emd_7770_additional.map.gz emd_7770_half_map_1.map.gz emd_7770_half_map_1.map.gz emd_7770_half_map_2.map.gz emd_7770_half_map_2.map.gz | 53.3 MB 71 MB 71 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-7770 http://ftp.pdbj.org/pub/emdb/structures/EMD-7770 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-7770 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-7770 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_7770_validation.pdf.gz emd_7770_validation.pdf.gz | 1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_7770_full_validation.pdf.gz emd_7770_full_validation.pdf.gz | 1 MB | 表示 | |
| XML形式データ |  emd_7770_validation.xml.gz emd_7770_validation.xml.gz | 19.7 KB | 表示 | |
| CIF形式データ |  emd_7770_validation.cif.gz emd_7770_validation.cif.gz | 25.9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7770 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7770 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7770 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7770 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_7770.map.gz / 形式: CCP4 / 大きさ: 147.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_7770.map.gz / 形式: CCP4 / 大きさ: 147.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Escherichia coli beta-galactosidase | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.637 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
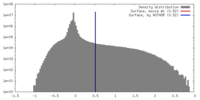
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_7770_msk_1.map emd_7770_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Escherichia coli beta-galactosidase, sharpened map
| ファイル | emd_7770_additional.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Escherichia coli beta-galactosidase, sharpened map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Escherichia coli beta-galactosidase, half map #1
| ファイル | emd_7770_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Escherichia coli beta-galactosidase, half map #1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Escherichia coli beta-galactosidase, half map #2
| ファイル | emd_7770_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Escherichia coli beta-galactosidase, half map #2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Escherichia coli beta-galactosidase bound to phenylethyl beta-D-t...
| 全体 | 名称: Escherichia coli beta-galactosidase bound to phenylethyl beta-D-thiogalactopyranoside (PETG) |
|---|---|
| 要素 |
|
-超分子 #1: Escherichia coli beta-galactosidase bound to phenylethyl beta-D-t...
| 超分子 | 名称: Escherichia coli beta-galactosidase bound to phenylethyl beta-D-thiogalactopyranoside (PETG) タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 465 KDa |
-分子 #1: Beta-galactosidase
| 分子 | 名称: Beta-galactosidase / タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO / EC番号: beta-galactosidase |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 116.181031 KDa |
| 配列 | 文字列: MITDSLAVVL QRRDWENPGV TQLNRLAAHP PFASWRNSEE ARTDRPSQQL RSLNGEWRFA WFPAPEAVPE SWLECDLPEA DTVVVPSNW QMHGYDAPIY TNVTYPITVN PPFVPTENPT GCYSLTFNVD ESWLQEGQTR IIFDGVNSAF HLWCNGRWVG Y GQDSRLPS ...文字列: MITDSLAVVL QRRDWENPGV TQLNRLAAHP PFASWRNSEE ARTDRPSQQL RSLNGEWRFA WFPAPEAVPE SWLECDLPEA DTVVVPSNW QMHGYDAPIY TNVTYPITVN PPFVPTENPT GCYSLTFNVD ESWLQEGQTR IIFDGVNSAF HLWCNGRWVG Y GQDSRLPS EFDLSAFLRA GENRLAVMVL RWSDGSYLED QDMWRMSGIF RDVSLLHKPT TQISDFHVAT RFNDDFSRAV LE AEVQMCG ELRDYLRVTV SLWQGETQVA SGTAPFGGEI IDERGGYADR VTLRLNVENP KLWSAEIPNL YRAVVELHTA DGT LIEAEA CDVGFRVVRI ENGLLLLNGK PLLIRGVNRH EHHPLHGQVM DEQTMVQDIL LMKQNNFNAV RCSHYPNHPL WYTL CDRYG LYVVDEANIE THGMVPMNRL TDDPRWLPAM SERVTRMVQR DRNHPSVIIW SLGNESGHGA NHDALYRWIK SVDPS RPVQ YEGGGADTTA TDIICPMYAR VDEDQPFPAV PKWSIKKWLS LPGETRPLIL CEYAHAMGNS LGGFAKYWQA FRQYPR LQG GFVWDWVDQS LIKYDENGNP WSAYGGDFGD TPNDRQFCMN GLVFADRTPH PALTEAKHQQ QFFQFRLSGQ TIEVTSE YL FRHSDNELLH WMVALDGKPL ASGEVPLDVA PQGKQLIELP ELPQPESAGQ LWLTVRVVQP NATAWSEAGH ISAWQQWR L AENLSVTLPA ASHAIPHLTT SEMDFCIELG NKRWQFNRQS GFLSQMWIGD KKQLLTPLRD QFTRAPLDND IGVSEATRI DPNAWVERWK AAGHYQAEAA LLQCTADTLA DAVLITTAHA WQHQGKTLFI SRKTYRIDGS GQMAITVDVV VASDTPHPAR IGLNCQLAQ VAERVNWLGL GPQENYPDRL TAACFDRWDL PLSDMYTPYV FPSENGLRCG TRELNYGPHQ WRGDFQFNIS R YSQQQLME TSHRHLLHAE EGTWLNIDGF HMGIGGDDSW SPSVSAEFQL SAGRYHYQLV WCQ UniProtKB: Beta-galactosidase |
-分子 #2: 2-phenylethyl 1-thio-beta-D-galactopyranoside
| 分子 | 名称: 2-phenylethyl 1-thio-beta-D-galactopyranoside / タイプ: ligand / ID: 2 / コピー数: 4 / 式: PTQ |
|---|---|
| 分子量 | 理論値: 300.371 Da |
| Chemical component information |  ChemComp-PTQ: |
-分子 #3: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 3 / コピー数: 8 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-分子 #4: SODIUM ION
| 分子 | 名称: SODIUM ION / タイプ: ligand / ID: 4 / コピー数: 8 |
|---|---|
| 分子量 | 理論値: 22.99 Da |
-分子 #5: water
| 分子 | 名称: water / タイプ: ligand / ID: 5 / コピー数: 4194 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2.3 mg/mL |
|---|---|
| 緩衝液 | pH: 8 詳細: 25 mM Tris, pH 8.0, 50 mM NaCl, 2 mM MgCl2, 1.0 mM TCEP |
| グリッド | モデル: Quantifoil R2/2 / 材質: COPPER / メッシュ: 200 / 詳細: plasma cleaned |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 90 % / チャンバー内温度: 90.15 K / 装置: LEICA EM GP / 詳細: Blot for 2 seconds before plunging.. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 温度 | 最低: 79.6 K / 最高: 79.8 K |
| 特殊光学系 | エネルギーフィルター - 名称: GIF Quantum LS エネルギーフィルター - エネルギー下限: 0 eV エネルギーフィルター - エネルギー上限: 20 eV |
| 詳細 | Parallel beam illumination |
| 撮影 | フィルム・検出器のモデル: GATAN K2 QUANTUM (4k x 4k) 検出モード: SUPER-RESOLUTION / デジタル化 - 画像ごとのフレーム数: 1-38 / 実像数: 1539 / 平均露光時間: 7.6 sec. / 平均電子線量: 45.0 e/Å2 / 詳細: Raw micrographs are available from EMPIAR-10061. |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 最大 デフォーカス(補正後): 2.0 µm / 倍率(補正後): 215000 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最小 デフォーカス(公称値): 0.6 µm / 倍率(公称値): 215000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT |
|---|---|
| 得られたモデル |  PDB-6cvm: |
 ムービー
ムービー コントローラー
コントローラー

















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)