+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-7600 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | CryoEM structure of human enterovirus D68 emptied particle (pH 5.5 and 33 degrees Celsius) | |||||||||
 マップデータ マップデータ | sharpened map in which the inner density is masked out | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | virus / genome release / acid | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報picornain 2A / symbiont-mediated suppression of host mRNA export from nucleus / symbiont genome entry into host cell via pore formation in plasma membrane / picornain 3C / T=pseudo3 icosahedral viral capsid / host cell cytoplasmic vesicle membrane / nucleoside-triphosphate phosphatase / channel activity / monoatomic ion transmembrane transport / RNA helicase activity ...picornain 2A / symbiont-mediated suppression of host mRNA export from nucleus / symbiont genome entry into host cell via pore formation in plasma membrane / picornain 3C / T=pseudo3 icosahedral viral capsid / host cell cytoplasmic vesicle membrane / nucleoside-triphosphate phosphatase / channel activity / monoatomic ion transmembrane transport / RNA helicase activity / symbiont-mediated suppression of host innate immune response / endocytosis involved in viral entry into host cell / symbiont-mediated activation of host autophagy / RNA-directed RNA polymerase / cysteine-type endopeptidase activity / viral RNA genome replication / RNA-directed RNA polymerase activity / DNA-templated transcription / virion attachment to host cell / host cell nucleus / structural molecule activity / ATP hydrolysis activity / proteolysis / RNA binding / zinc ion binding / ATP binding / membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  Enterovirus D68 (エンテロウイルス) Enterovirus D68 (エンテロウイルス) | |||||||||
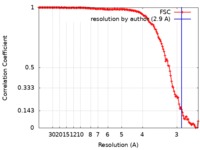
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.9 Å | |||||||||
 データ登録者 データ登録者 | Liu Y / Rossmann MG | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2018 ジャーナル: Proc Natl Acad Sci U S A / 年: 2018タイトル: Molecular basis for the acid-initiated uncoating of human enterovirus D68. 著者: Yue Liu / Ju Sheng / Arno L W van Vliet / Geeta Buda / Frank J M van Kuppeveld / Michael G Rossmann /   要旨: Enterovirus D68 (EV-D68) belongs to a group of enteroviruses that contain a single positive-sense RNA genome surrounded by an icosahedral capsid. Like common cold viruses, EV-D68 mainly causes ...Enterovirus D68 (EV-D68) belongs to a group of enteroviruses that contain a single positive-sense RNA genome surrounded by an icosahedral capsid. Like common cold viruses, EV-D68 mainly causes respiratory infections and is acid-labile. The molecular mechanism by which the acid-sensitive EV-D68 virions uncoat and deliver their genome into a host cell is unknown. Using cryoelectron microscopy (cryo-EM), we have determined the structures of the full native virion and an uncoating intermediate [the A (altered) particle] of EV-D68 at 2.2- and 2.7-Å resolution, respectively. These structures showed that acid treatment of EV-D68 leads to particle expansion, externalization of the viral protein VP1 N termini from the capsid interior, and formation of pores around the icosahedral twofold axes through which the viral RNA can exit. Moreover, because of the low stability of EV-D68, cryo-EM analyses of a mixed population of particles at neutral pH and following acid treatment demonstrated the involvement of multiple structural intermediates during virus uncoating. Among these, a previously undescribed state, the expanded 1 ("E1") particle, shows a majority of internal regions (e.g., the VP1 N termini) to be ordered as in the full native virion. Thus, the E1 particle acts as an intermediate in the transition from full native virions to A particles. Together, the present work delineates the pathway of EV-D68 uncoating and provides the molecular basis for the acid lability of EV-D68 and of the related common cold viruses. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_7600.map.gz emd_7600.map.gz | 202.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-7600-v30.xml emd-7600-v30.xml emd-7600.xml emd-7600.xml | 22.5 KB 22.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_7600_fsc.xml emd_7600_fsc.xml | 15.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_7600.png emd_7600.png | 225.5 KB | ||
| Filedesc metadata |  emd-7600.cif.gz emd-7600.cif.gz | 6.5 KB | ||
| その他 |  emd_7600_half_map_1.map.gz emd_7600_half_map_1.map.gz emd_7600_half_map_2.map.gz emd_7600_half_map_2.map.gz | 57.6 MB 57.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-7600 http://ftp.pdbj.org/pub/emdb/structures/EMD-7600 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-7600 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-7600 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_7600_validation.pdf.gz emd_7600_validation.pdf.gz | 930.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_7600_full_validation.pdf.gz emd_7600_full_validation.pdf.gz | 930.5 KB | 表示 | |
| XML形式データ |  emd_7600_validation.xml.gz emd_7600_validation.xml.gz | 20.9 KB | 表示 | |
| CIF形式データ |  emd_7600_validation.cif.gz emd_7600_validation.cif.gz | 27 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7600 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7600 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7600 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7600 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  6cshMC  7567C  7569C  7571C  7572C  7583C  7589C  7592C  7593C  7598C  7599C  9053C  9054C  9055C  9056C  9057C  9058C  9059C  9060C  6crpC  6crrC  6crsC  6cruC  6cs3C  6cs4C  6cs5C  6cs6C  6csaC  6csgC  6mziC C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_7600.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_7600.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | sharpened map in which the inner density is masked out | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.3 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
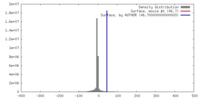
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-ハーフマップ: unsharpened half map in which the inner density is retained
| ファイル | emd_7600_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | unsharpened half map in which the inner density is retained | ||||||||||||
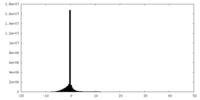
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: unsharpened half map in which the inner density is retained
| ファイル | emd_7600_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | unsharpened half map in which the inner density is retained | ||||||||||||
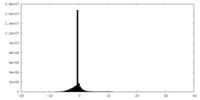
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Enterovirus D68
| 全体 | 名称:  Enterovirus D68 (エンテロウイルス) Enterovirus D68 (エンテロウイルス) |
|---|---|
| 要素 |
|
-超分子 #1: Enterovirus D68
| 超分子 | 名称: Enterovirus D68 / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: all / 詳細: Viruses were grown in RD cells. / NCBI-ID: 42789 / 生物種: Enterovirus D68 / Sci species strain: US/MO/14-18947 / ウイルスタイプ: VIRION / ウイルス・単離状態: STRAIN / ウイルス・エンベロープ: No / ウイルス・中空状態: Yes |
|---|
-分子 #1: viral protein 1
| 分子 | 名称: viral protein 1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Enterovirus D68 (エンテロウイルス) / 株: US/MO/14-18947 Enterovirus D68 (エンテロウイルス) / 株: US/MO/14-18947 |
| 分子量 | 理論値: 32.920309 KDa |
| 配列 | 文字列: IESIIKTATD TVKSEINAEL GVVPSLNAVE TGATSNTEPE EAIQTRTVIN QHGVSETLVE NFLSRAALVS KRSFEYKDHT SSTARADKN FFKWTINTRS FVQLRRKLEL FTYLRFDAEI TILTTVAVNG SGNNTYVGLP DLTLQAMFVP TGALTPEKQD S FHWQSGSN ...文字列: IESIIKTATD TVKSEINAEL GVVPSLNAVE TGATSNTEPE EAIQTRTVIN QHGVSETLVE NFLSRAALVS KRSFEYKDHT SSTARADKN FFKWTINTRS FVQLRRKLEL FTYLRFDAEI TILTTVAVNG SGNNTYVGLP DLTLQAMFVP TGALTPEKQD S FHWQSGSN ASVFFKISDP PARITIPFMC INSAYSVFYD GFAGFEKNGL YGINPADTIG NLCVRIVNEH QPVGFTVTVR VY MKPKHIK AWAPRPPRTL PYMSIANANY KGKERAPNAL SAIIGNRDSV KTMPHNIVNT UniProtKB: Genome polyprotein |
-分子 #2: viral protein 3
| 分子 | 名称: viral protein 3 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Enterovirus D68 (エンテロウイルス) / 株: US/MO/14-18947 Enterovirus D68 (エンテロウイルス) / 株: US/MO/14-18947 |
| 分子量 | 理論値: 27.112814 KDa |
| 配列 | 文字列: GVPTYLLPGS GQFLTTDDHS SAPALPCFNP TPEMHIPGQV RNMLEVVQVE SMMEINNTES AVGMERLKVD ISALTDVDQL LFNIPLDIQ LDGPLRNTLV GNISRYYTHW SGSLEMTFMF CGSFMAAGKL ILCYTPPGGS CPTTRETAML GTHIVWDFGL Q SSVTLIIP ...文字列: GVPTYLLPGS GQFLTTDDHS SAPALPCFNP TPEMHIPGQV RNMLEVVQVE SMMEINNTES AVGMERLKVD ISALTDVDQL LFNIPLDIQ LDGPLRNTLV GNISRYYTHW SGSLEMTFMF CGSFMAAGKL ILCYTPPGGS CPTTRETAML GTHIVWDFGL Q SSVTLIIP WISGSHYRMF NNDAKSTNAN VGYVTCFMQT NLIVPSESSD TCSLIGFIAA KDDFSLRLMR DSPDIGQLDH LH AAEAAYQ UniProtKB: Genome polyprotein |
-分子 #3: viral protein 2
| 分子 | 名称: viral protein 2 / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Enterovirus D68 (エンテロウイルス) / 株: US/MO/14-18947 Enterovirus D68 (エンテロウイルス) / 株: US/MO/14-18947 |
| 分子量 | 理論値: 27.567135 KDa |
| 配列 | 文字列: SPSAEACGYS DRVLQLKLGN SAIVTQEAAN YCCAYGEWPN YLPDHEAVAI DKPTQPETAT DRFYTLKSVK WETGSTGWWW KLPDALNNI GMFGQNVQHH YLYRSGFLIH VQCNATKFHQ GALLVVAIPE HQRGAHNTNT SPGFDDIMKG EEGGTFNHPY V LDDGTSLA ...文字列: SPSAEACGYS DRVLQLKLGN SAIVTQEAAN YCCAYGEWPN YLPDHEAVAI DKPTQPETAT DRFYTLKSVK WETGSTGWWW KLPDALNNI GMFGQNVQHH YLYRSGFLIH VQCNATKFHQ GALLVVAIPE HQRGAHNTNT SPGFDDIMKG EEGGTFNHPY V LDDGTSLA CATIFPHQWI NLRTNNSATI VLPWMNAAPM DFPLRHNQWT LAIIPVVPLG TRTTSSMVPI TVSIAPMCCE FN GLRHAIT Q UniProtKB: Genome polyprotein |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.2 詳細: Phosphate-citrate buffer. Viruses were treated with phosphate-citrate buffer at pH 5.5 and 33 degrees Celsius, and then neutralized back to about pH 7.2. |
|---|---|
| グリッド | モデル: Homemade / 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: CONTINUOUS / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 120 sec. / 前処理 - 雰囲気: AIR / 前処理 - 気圧: 0.02 kPa |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 80 % / チャンバー内温度: 298 K / 装置: GATAN CRYOPLUNGE 3 |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / デジタル化 - サイズ - 横: 3710 pixel / デジタル化 - サイズ - 縦: 3838 pixel / デジタル化 - 画像ごとのフレーム数: 2-40 / 撮影したグリッド数: 1 / 実像数: 950 / 平均露光時間: 8.0 sec. / 平均電子線量: 38.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.5 µm / 最小 デフォーカス(公称値): 0.5 µm / 倍率(公称値): 22500 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 詳細 | A combination of the following approaches was used: (1) model rebuilding using Coot, (2) real space refinement using Phenix, (3) reciprocal space refinment using REFMAC5 (as in standard crystallographic refinement). |
|---|---|
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT / 当てはまり具合の基準: Correlation coefficient |
| 得られたモデル |  PDB-6csh: |
 ムービー
ムービー コントローラー
コントローラー














 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)