+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-7451 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of the Gasdermin A3 membrane pore | |||||||||
 マップデータ マップデータ | Gasdermin A3 membrane pore with C27 symmetry and double-layer architecture | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.6 Å | |||||||||
 データ登録者 データ登録者 | Ruan J / Xia S / Wu H | |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2018 ジャーナル: Nature / 年: 2018タイトル: Cryo-EM structure of the gasdermin A3 membrane pore. 著者: Jianbin Ruan / Shiyu Xia / Xing Liu / Judy Lieberman / Hao Wu /  要旨: Gasdermins mediate inflammatory cell death after cleavage by caspases or other, unknown enzymes. The cleaved N-terminal fragments bind to acidic membrane lipids to form pores, but the mechanism of ...Gasdermins mediate inflammatory cell death after cleavage by caspases or other, unknown enzymes. The cleaved N-terminal fragments bind to acidic membrane lipids to form pores, but the mechanism of pore formation remains unresolved. Here we present the cryo-electron microscopy structures of the 27-fold and 28-fold single-ring pores formed by the N-terminal fragment of mouse GSDMA3 (GSDMA3-NT) at 3.8 and 4.2 Å resolutions, and of a double-ring pore at 4.6 Å resolution. In the 27-fold pore, a 108-stranded anti-parallel β-barrel is formed by two β-hairpins from each subunit capped by a globular domain. We identify a positively charged helix that interacts with the acidic lipid cardiolipin. GSDMA3-NT undergoes radical conformational changes upon membrane insertion to form long, membrane-spanning β-strands. We also observe an unexpected additional symmetric ring of GSDMA3-NT subunits that does not insert into the membrane in the double-ring pore, which may represent a pre-pore state of GSDMA3-NT. These structures provide a basis that explains the activities of several mutant gasdermins, including defective mutants that are associated with cancer. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_7451.map.gz emd_7451.map.gz | 187.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-7451-v30.xml emd-7451-v30.xml emd-7451.xml emd-7451.xml | 14.1 KB 14.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_7451_fsc.xml emd_7451_fsc.xml | 13.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_7451.png emd_7451.png | 63.4 KB | ||
| その他 |  emd_7451_half_map_1.map.gz emd_7451_half_map_1.map.gz emd_7451_half_map_2.map.gz emd_7451_half_map_2.map.gz | 191.8 MB 191.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-7451 http://ftp.pdbj.org/pub/emdb/structures/EMD-7451 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-7451 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-7451 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_7451_validation.pdf.gz emd_7451_validation.pdf.gz | 78.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_7451_full_validation.pdf.gz emd_7451_full_validation.pdf.gz | 77.6 KB | 表示 | |
| XML形式データ |  emd_7451_validation.xml.gz emd_7451_validation.xml.gz | 493 B | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7451 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7451 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7451 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7451 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_7451.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_7451.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Gasdermin A3 membrane pore with C27 symmetry and double-layer architecture | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.1686 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
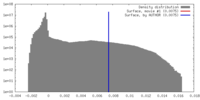
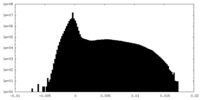
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-ハーフマップ: Half map 1 of the Gasdermin A3 membrane...
| ファイル | emd_7451_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 1 of the Gasdermin A3 membrane pore with C27 symmetry and double-layer architecture | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
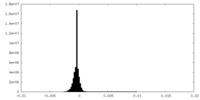
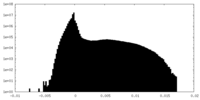
| 密度ヒストグラム |
-ハーフマップ: Half map 2 of the Gasdermin A3 membrane...
| ファイル | emd_7451_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 2 of the Gasdermin A3 membrane pore with C27 symmetry and double-layer architecture | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
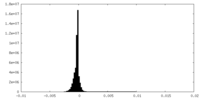
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Gasdermin A3 N domain
| 全体 | 名称: Gasdermin A3 N domain |
|---|---|
| 要素 |
|
-超分子 #1: Gasdermin A3 N domain
| 超分子 | 名称: Gasdermin A3 N domain / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all 詳細: mouse Gasdermin A3 pores with C27 symmetry and double-layer architecture |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  |
| 分子量 | 理論値: 1.62 MDa |
-分子 #1: Gasdermin A3
| 分子 | 名称: Gasdermin A3 / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MHMPVFEDVT RALVRELNPR GDLTPLDSLI DFKHFRPFCL VLRKRKSTLF WGARYVRTDY TLLDLLEPGS SPSDLTDSGN FSFKNMLDVQ VQGLVEVPKT VKVKGTAGLS QSSTLEVQTL SVAPSALENL KKERKLSADH SFLNEMRYHE KNLYVVMEAV EAKQEVTVEQ ...文字列: MHMPVFEDVT RALVRELNPR GDLTPLDSLI DFKHFRPFCL VLRKRKSTLF WGARYVRTDY TLLDLLEPGS SPSDLTDSGN FSFKNMLDVQ VQGLVEVPKT VKVKGTAGLS QSSTLEVQTL SVAPSALENL KKERKLSADH SFLNEMRYHE KNLYVVMEAV EAKQEVTVEQ TGNANAIFSL PSLALLGLQG SLNNNKAVTI PKGCVLAYRV RLLRVFLFNL WDIPYICNDS MQTFPKIRRV PCSAFISPTQ MISEEPEEEK LIGELEVLFQ |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2 mg/mL |
|---|---|
| 緩衝液 | pH: 8 |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / 平均電子線量: 41.1 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー








 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)