+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of Mycobacterium smegmatis EtfD | |||||||||
 マップデータ マップデータ | CryoEM map of Mycobacterium smegmatis EtfD (sharpened) | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | oxidoreductase / membrane protein / electron transport chain / beta oxidation | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報4 iron, 4 sulfur cluster binding / oxidoreductase activity / metal ion binding / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  Mycolicibacterium smegmatis MC2 155 (バクテリア) Mycolicibacterium smegmatis MC2 155 (バクテリア) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.2 Å | |||||||||
 データ登録者 データ登録者 | Courbon GM / Rubinstein JL | |||||||||
| 資金援助 |  カナダ, 1件 カナダ, 1件
| |||||||||
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Publishedタイトル: Mycobacterial EtfD contains an unusual linear [3Fe-4S] cluster and enables beta oxidation to drive proton pumping by the electron transport chain 著者: Courbon GM / Rubinstein JL | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_70545.map.gz emd_70545.map.gz | 118.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-70545-v30.xml emd-70545-v30.xml emd-70545.xml emd-70545.xml | 19.5 KB 19.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_70545_fsc.xml emd_70545_fsc.xml | 10.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_70545.png emd_70545.png | 68.2 KB | ||
| Filedesc metadata |  emd-70545.cif.gz emd-70545.cif.gz | 6.3 KB | ||
| その他 |  emd_70545_additional_1.map.gz emd_70545_additional_1.map.gz emd_70545_half_map_1.map.gz emd_70545_half_map_1.map.gz emd_70545_half_map_2.map.gz emd_70545_half_map_2.map.gz | 63 MB 116.2 MB 116.2 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-70545 http://ftp.pdbj.org/pub/emdb/structures/EMD-70545 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-70545 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-70545 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_70545_validation.pdf.gz emd_70545_validation.pdf.gz | 880 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_70545_full_validation.pdf.gz emd_70545_full_validation.pdf.gz | 879.6 KB | 表示 | |
| XML形式データ |  emd_70545_validation.xml.gz emd_70545_validation.xml.gz | 18.8 KB | 表示 | |
| CIF形式データ |  emd_70545_validation.cif.gz emd_70545_validation.cif.gz | 24.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-70545 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-70545 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-70545 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-70545 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  9ojnMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_70545.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_70545.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | CryoEM map of Mycobacterium smegmatis EtfD (sharpened) | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.024 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: CryoEM map of Mycobacterium smegmatis EtfD (unsharpened)
| ファイル | emd_70545_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | CryoEM map of Mycobacterium smegmatis EtfD (unsharpened) | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: CryoEM map of Mycobacterium smegmatis EtfD (Half B)
| ファイル | emd_70545_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | CryoEM map of Mycobacterium smegmatis EtfD (Half B) | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: CryoEM map of Mycobacterium smegmatis EtfD (Half A)
| ファイル | emd_70545_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | CryoEM map of Mycobacterium smegmatis EtfD (Half A) | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Structure of Mycobacterium smegmatis EtfD
| 全体 | 名称: Structure of Mycobacterium smegmatis EtfD |
|---|---|
| 要素 |
|
-超分子 #1: Structure of Mycobacterium smegmatis EtfD
| 超分子 | 名称: Structure of Mycobacterium smegmatis EtfD / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Mycolicibacterium smegmatis MC2 155 (バクテリア) Mycolicibacterium smegmatis MC2 155 (バクテリア) |
-分子 #1: Iron-sulfur-binding reductase
| 分子 | 名称: Iron-sulfur-binding reductase / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Mycolicibacterium smegmatis MC2 155 (バクテリア) Mycolicibacterium smegmatis MC2 155 (バクテリア) |
| 分子量 | 理論値: 111.045625 KDa |
| 配列 | 文字列: MAHTLEVSRL IIGLLMTAIV LVFAAKRVLW LTKLIRSGQK TLDENGRKND LQKRITTQIT EVFGQTRLLR WSVPGIAHFF TMWGFFVLA SVYLEAYGVL FDPEFHIPFI GRWPVLGFLQ DFFAVAVLLG IIVFAIIRVV REPKKIGRDS RFYGSHTGGA W EILFMIFL ...文字列: MAHTLEVSRL IIGLLMTAIV LVFAAKRVLW LTKLIRSGQK TLDENGRKND LQKRITTQIT EVFGQTRLLR WSVPGIAHFF TMWGFFVLA SVYLEAYGVL FDPEFHIPFI GRWPVLGFLQ DFFAVAVLLG IIVFAIIRVV REPKKIGRDS RFYGSHTGGA W EILFMIFL VIATYALFRG AAVNTLGERF PYQSGAFFSD FMAWILRPLG ATANMWIETV ALMGHIGVML VFLLIVLHSK HL HIGLAPI NVTFKRLPNG LGPLLPMESN GEYIDFEDPA EDAVFGKGKI EDFTWKGYLD FTTCTECGRC QSQCPAWNTG KPL SPKLVI MNLRDHMFAK APYILGEKPS PLESTPEGGL GEKARGEKHE QKHAHDHVPE SGFERILGSG PEQALRPLVG TEEQ GGVID PDVLWSCTNC GACVEQCPVD IEHIDHIVDM RRYQVMVESE FPGELGVLFK NLETKGNPWG QNAKDRTNWI DEVDF DVPV YGEDVDSFDG FEYLFWVGCA GAYEDRAKKT TKAVAELLAT AGVKFLVLGT GETCTGDSAR RSGNEFLFQQ LAAQNV ETI NELFEGVETV DRKIVVTCPH CFNTIGREYP QLGANYSVVH HTQLLNRLVR DKKLVPVKSV SEQNGQPVTY HDPCFLG RH NKVYEAPREL VEASGVTLKE MPRHADRGLC CGAGGARMWM EEHIGKRVNV ERTEEAMDTA STIATGCPFC RVMITDGV D DVAASRNVEK AEVLDVAQLL LNSLDTSKVT LPEKGTAAKE SEKRAAARAE AEAKAEAAAP PVEEAAPEAE APAAPAAGG AEAKPVTGLG MAGAAKRPGA KKAAPAAEAS AAPAAAPAPA KGLGLAGGAK RPGAKKAAAP AAEAPAAPAS DAPPVKGLGL AGGAKRPGA KKTAAAAPAE KPAATEAPEA SATPAAPAAP VKGLGLAAGA KRPGAKKTAA APAEKPAAAE TEAPAPAETA A PAEPAKPE PPVVGLGIAA GARRPGAKKA AAKPAAAPAP AAEKPAEQAA EPEKPAEKPA EPEKPEPPVV GLGIKPGAKR PG KR UniProtKB: Iron-sulfur-binding reductase |
-分子 #2: DODECYL-BETA-D-MALTOSIDE
| 分子 | 名称: DODECYL-BETA-D-MALTOSIDE / タイプ: ligand / ID: 2 / コピー数: 1 / 式: LMT |
|---|---|
| 分子量 | 理論値: 510.615 Da |
| Chemical component information |  ChemComp-LMT: |
-分子 #3: MENAQUINONE-9
| 分子 | 名称: MENAQUINONE-9 / タイプ: ligand / ID: 3 / コピー数: 1 / 式: MQ9 |
|---|---|
| 分子量 | 理論値: 785.233 Da |
| Chemical component information |  ChemComp-MQ9: |
-分子 #4: PROTOPORPHYRIN IX CONTAINING FE
| 分子 | 名称: PROTOPORPHYRIN IX CONTAINING FE / タイプ: ligand / ID: 4 / コピー数: 1 / 式: HEM |
|---|---|
| 分子量 | 理論値: 616.487 Da |
| Chemical component information |  ChemComp-HEM: |
-分子 #5: IRON/SULFUR CLUSTER
| 分子 | 名称: IRON/SULFUR CLUSTER / タイプ: ligand / ID: 5 / コピー数: 2 / 式: SF4 |
|---|---|
| 分子量 | 理論値: 351.64 Da |
| Chemical component information |  ChemComp-FS1: |
-分子 #6: Non-cubane [4Fe-4S]-cluster
| 分子 | 名称: Non-cubane [4Fe-4S]-cluster / タイプ: ligand / ID: 6 / コピー数: 1 / 式: 9S8 |
|---|---|
| 分子量 | 理論値: 351.64 Da |
| Chemical component information | 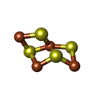 ChemComp-9S8: |
-分子 #7: 1,3,5,7-tetrathia-2$l^{2},4$l^{4},6$l^{2}-triferraspiro[3.3]heptane
| 分子 | 名称: 1,3,5,7-tetrathia-2$l^{2},4$l^{4},6$l^{2}-triferraspiro[3.3]heptane タイプ: ligand / ID: 7 / コピー数: 1 / 式: A1CBX |
|---|---|
| 分子量 | 理論値: 295.795 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: TFS FALCON 4i (4k x 4k) 平均電子線量: 70.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD 最大 デフォーカス(公称値): 2.8000000000000003 µm 最小 デフォーカス(公称値): 0.4 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー













 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)













































