+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-7030 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Staphylococcus aureus phage 80alpha procapsid | |||||||||
 マップデータ マップデータ | Capsid protein and C-terminal part of scaffolding protein in the Staphylococcus aureus phage 80alpha procapsid: clipped map used for model building and refinement in Coot and REFMAC | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | major capsid protein / HK97-like fold / scaffolding protein / procapsid / VIRUS | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報viral scaffold / viral capsid / symbiont-mediated suppression of host innate immune response 類似検索 - 分子機能 | |||||||||
| 生物種 |  Staphylococcus phage 80alpha (ファージ) Staphylococcus phage 80alpha (ファージ) | |||||||||
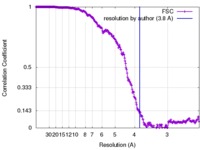
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.8 Å | |||||||||
 データ登録者 データ登録者 | Kizziah JL / Dearborn AD | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2017 ジャーナル: Elife / 年: 2017タイトル: Competing scaffolding proteins determine capsid size during mobilization of pathogenicity islands. 著者: Altaira D Dearborn / Erin A Wall / James L Kizziah / Laura Klenow / Laura K Parker / Keith A Manning / Michael S Spilman / John M Spear / Gail E Christie / Terje Dokland /  要旨: pathogenicity islands (SaPIs), such as SaPI1, exploit specific helper bacteriophages, like 80α, for their high frequency mobilization, a process termed 'molecular piracy'. SaPI1 redirects the ... pathogenicity islands (SaPIs), such as SaPI1, exploit specific helper bacteriophages, like 80α, for their high frequency mobilization, a process termed 'molecular piracy'. SaPI1 redirects the helper's assembly pathway to form small capsids that can only accommodate the smaller SaPI1 genome, but not a complete phage genome. SaPI1 encodes two proteins, CpmA and CpmB, that are responsible for this size redirection. We have determined the structures of the 80α and SaPI1 procapsids to near-atomic resolution by cryo-electron microscopy, and show that CpmB competes with the 80α scaffolding protein (SP) for a binding site on the capsid protein (CP), and works by altering the angle between capsomers. We probed these interactions genetically and identified second-site suppressors of lethal mutations in SP. Our structures show, for the first time, the detailed interactions between SP and CP in a bacteriophage, providing unique insights into macromolecular assembly processes. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_7030.map.gz emd_7030.map.gz | 199.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-7030-v30.xml emd-7030-v30.xml emd-7030.xml emd-7030.xml | 14.1 KB 14.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_7030_fsc.xml emd_7030_fsc.xml | 26.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_7030.png emd_7030.png | 300.2 KB | ||
| Filedesc metadata |  emd-7030.cif.gz emd-7030.cif.gz | 5.6 KB | ||
| その他 |  emd_7030_additional.map.gz emd_7030_additional.map.gz | 974.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-7030 http://ftp.pdbj.org/pub/emdb/structures/EMD-7030 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-7030 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-7030 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_7030_validation.pdf.gz emd_7030_validation.pdf.gz | 582.2 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_7030_full_validation.pdf.gz emd_7030_full_validation.pdf.gz | 581.8 KB | 表示 | |
| XML形式データ |  emd_7030_validation.xml.gz emd_7030_validation.xml.gz | 14.4 KB | 表示 | |
| CIF形式データ |  emd_7030_validation.cif.gz emd_7030_validation.cif.gz | 21.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7030 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7030 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7030 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7030 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_7030.map.gz / 形式: CCP4 / 大きさ: 210.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_7030.map.gz / 形式: CCP4 / 大きさ: 210.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Capsid protein and C-terminal part of scaffolding protein in the Staphylococcus aureus phage 80alpha procapsid: clipped map used for model building and refinement in Coot and REFMAC | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 これらの図は立方格子座標系で作成されたものです | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.21 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
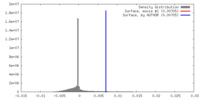
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: Capsid protein and C-terminal part of scaffolding protein...
| ファイル | emd_7030_additional.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Capsid protein and C-terminal part of scaffolding protein in the Staphylococcus aureus phage 80alpha procapsid: final map sharpened with a B-factor of 300 to 6 Angstrom resolution (cutoff at 3 Angstrom) | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
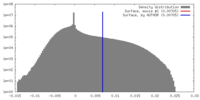
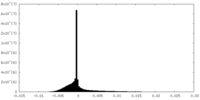
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Staphylococcus phage 80alpha
| 全体 | 名称:  Staphylococcus phage 80alpha (ファージ) Staphylococcus phage 80alpha (ファージ) |
|---|---|
| 要素 |
|
-超分子 #1: Staphylococcus phage 80alpha
| 超分子 | 名称: Staphylococcus phage 80alpha / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: all 詳細: Lysogenic 80alpha with small terminase gene deletion NCBI-ID: 53369 / 生物種: Staphylococcus phage 80alpha / ウイルスタイプ: VIRION / ウイルス・単離状態: SPECIES / ウイルス・エンベロープ: No / ウイルス・中空状態: Yes |
|---|---|
| 宿主 | 生物種:  |
| 分子量 | 理論値: 25.27 MDa |
| ウイルス殻 | Shell ID: 1 / 名称: Procapsid / 直径: 546.0 Å / T番号(三角分割数): 7 |
-分子 #1: Major head protein
| 分子 | 名称: Major head protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 7 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Staphylococcus phage 80alpha (ファージ) Staphylococcus phage 80alpha (ファージ) |
| 分子量 | 理論値: 36.846883 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MEQTQKLKLN LQHFASNNVK PQVFNPDNVM MHEKKDGTLM NEFTTPILQE VMENSKIMQL GKYEPMEGTE KKFTFWADKP GAYWVGEGQ KIETSKATWV NATMRAFKLG VILPVTKEFL NYTYSQFFEE MKPMIAEAFY KKFDEAGILN QGNNPFGKSI A QSIEKTNK ...文字列: MEQTQKLKLN LQHFASNNVK PQVFNPDNVM MHEKKDGTLM NEFTTPILQE VMENSKIMQL GKYEPMEGTE KKFTFWADKP GAYWVGEGQ KIETSKATWV NATMRAFKLG VILPVTKEFL NYTYSQFFEE MKPMIAEAFY KKFDEAGILN QGNNPFGKSI A QSIEKTNK VIKGDFTQDN IIDLEALLED DELEANAFIS KTQNRSLLRK IVDPETKERI YDRNSDSLDG LPVVNLKSSN LK RGELITG DFDKLIYGIP QLIEYKIDET AQLSTVKNED GTPVNLFEQD MVALRATMHV ALHIADDKAF AKLVPADKRT DSV PGEV UniProtKB: Major capsid protein |
-分子 #2: Scaffold protein
| 分子 | 名称: Scaffold protein / タイプ: protein_or_peptide / ID: 2 / コピー数: 7 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Staphylococcus phage 80alpha (ファージ) Staphylococcus phage 80alpha (ファージ) |
| 分子量 | 理論値: 23.410941 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MEENKLKFNL QFFADQSDDP DEPGGDGKKG NPDKKENDEG TEITFTPEQQ KKVDEILERR VAHEKKKADE YAKEKAAEAA KEAAKLAKM NKDQKDEYER EQMEKELEQL RSEKQLNEMR SEARKMLSEA EVDSSDEVVN LVVTDTAEQT KSNVEAFSNA V KKAVNEAV ...文字列: MEENKLKFNL QFFADQSDDP DEPGGDGKKG NPDKKENDEG TEITFTPEQQ KKVDEILERR VAHEKKKADE YAKEKAAEAA KEAAKLAKM NKDQKDEYER EQMEKELEQL RSEKQLNEMR SEARKMLSEA EVDSSDEVVN LVVTDTAEQT KSNVEAFSNA V KKAVNEAV KVNARQSPLT GGDSFNHSTK NKPQNLAEIA RQKRIIKN UniProtKB: Capsid assembly scaffolding protein |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.8 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: DIRECT ELECTRON DE-20 (5k x 3k) 平均電子線量: 40.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー













 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)