+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-6836 | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Human HP1beta-dinucleosome H3K9Me3 | ||||||||||||
 マップデータ マップデータ | HP1 beta dinucleosome-H3K9me3 complex | ||||||||||||
 試料 試料 |
| ||||||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||||||||
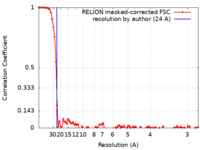
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 24.0 Å | ||||||||||||
 データ登録者 データ登録者 | Machida S / Takizawa Y / Ishimaru M / Sekine S / Sugita Y / Nakayama J / Wolf M / Kurumizaka H | ||||||||||||
| 資金援助 |  日本, 3件 日本, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Mol Cell / 年: 2018 ジャーナル: Mol Cell / 年: 2018タイトル: Structural Basis of Heterochromatin Formation by Human HP1. 著者: Shinichi Machida / Yoshimasa Takizawa / Masakazu Ishimaru / Yukihiko Sugita / Satoshi Sekine / Jun-Ichi Nakayama / Matthias Wolf / Hitoshi Kurumizaka /  要旨: Heterochromatin plays important roles in transcriptional silencing and genome maintenance by the formation of condensed chromatin structures, which determine the epigenetic status of eukaryotic cells. ...Heterochromatin plays important roles in transcriptional silencing and genome maintenance by the formation of condensed chromatin structures, which determine the epigenetic status of eukaryotic cells. The trimethylation of histone H3 lysine 9 (H3K9me3), a target of heterochromatin protein 1 (HP1), is a hallmark of heterochromatin formation. However, the mechanism by which HP1 folds chromatin-containing H3K9me3 into a higher-order structure has not been elucidated. Here we report the three-dimensional structure of the H3K9me3-containing dinucleosomes complexed with human HP1α, HP1β, and HP1γ, determined by cryogenic electron microscopy with a Volta phase plate. In the structures, two H3K9me3 nucleosomes are bridged by a symmetric HP1 dimer. Surprisingly, the linker DNA between the nucleosomes does not directly interact with HP1, thus allowing nucleosome remodeling by the ATP-utilizing chromatin assembly and remodeling factor (ACF). The structure depicts the fundamental architecture of heterochromatin. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_6836.map.gz emd_6836.map.gz | 20.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-6836-v30.xml emd-6836-v30.xml emd-6836.xml emd-6836.xml | 15.9 KB 15.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_6836_fsc.xml emd_6836_fsc.xml | 9.3 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_6836.png emd_6836.png | 30.7 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-6836 http://ftp.pdbj.org/pub/emdb/structures/EMD-6836 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-6836 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-6836 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_6836_validation.pdf.gz emd_6836_validation.pdf.gz | 78.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_6836_full_validation.pdf.gz emd_6836_full_validation.pdf.gz | 77.7 KB | 表示 | |
| XML形式データ |  emd_6836_validation.xml.gz emd_6836_validation.xml.gz | 495 B | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6836 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6836 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6836 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6836 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_6836.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_6836.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | HP1 beta dinucleosome-H3K9me3 complex | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.4 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Human HP1beta-dinucleosome H3K9Me3 complex
| 全体 | 名称: Human HP1beta-dinucleosome H3K9Me3 complex |
|---|---|
| 要素 |
|
-超分子 #1: Human HP1beta-dinucleosome H3K9Me3 complex
| 超分子 | 名称: Human HP1beta-dinucleosome H3K9Me3 complex / タイプ: complex / ID: 1 / 親要素: 0 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  組換プラスミド: pH2A, pH2B, pH3.2K9C/C110A, pH4, pHP1b-pre |
| 分子量 | 理論値: 500 KDa |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | .8 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| |||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 200 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 支持フィルム - Film thickness: 100.0 nm / 前処理 - タイプ: PLASMA CLEANING / 前処理 - 雰囲気: OTHER / 前処理 - 気圧: 9.33 kPa / 詳細: Gatan Solarus | |||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 289 K / 装置: FEI VITROBOT MARK IV / 詳細: 3 second blot, 2.5uL. | |||||||||
| 詳細 | sample was monodisperse |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 温度 | 最低: 77.0 K / 最高: 100.0 K |
| アライメント法 | Coma free - Residual tilt: 0.1 mrad |
| 特殊光学系 | 位相板: VOLTA PHASE PLATE |
| 詳細 | nanoprobe, parallel beam illumination |
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: INTEGRATING / デジタル化 - サイズ - 横: 4096 pixel / デジタル化 - サイズ - 縦: 4096 pixel / デジタル化 - サンプリング間隔: 14.0 µm / デジタル化 - 画像ごとのフレーム数: 1-40 / 撮影したグリッド数: 1 / 実像数: 2683 / 平均露光時間: 1.0 sec. / 平均電子線量: 67.0 e/Å2 詳細: Images were collected with a volta phase plate (VPP) at a target defocus of -500nm |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 最大 デフォーカス(補正後): -0.5 µm / 最小 デフォーカス(補正後): -0.5 µm / 倍率(補正後): 100000 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): -0.5 µm / 最小 デフォーカス(公称値): -0.5 µm / 倍率(公称値): 73000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)