+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-6722 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Drosophila full length cryoEM structure of C3PO complex | |||||||||
 マップデータ マップデータ | cryo-EM structure of full-length C3PO (WT) adopt a closed football-like shape with a hollow interior cavity consisting of four Trax/Translin heterodimers. | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |  | |||||||||
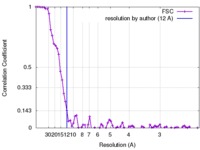
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 12.0 Å | |||||||||
 データ登録者 データ登録者 | Mo X / Yuan AY | |||||||||
 引用 引用 |  ジャーナル: Nucleic Acids Res / 年: 2018 ジャーナル: Nucleic Acids Res / 年: 2018タイトル: Structural insights into Drosophila-C3PO complex assembly and 'Dynamic Side Port' model in substrate entry and release. 著者: Xiaobing Mo / Xia Yang / Yuren Adam Yuan /   要旨: In Drosophila and human, component 3 promoter of RISC (C3PO), a heteromeric complex, enhances RISC assembly and promotes RISC activity. Here, we report crystal structure of full-length Drosophila ...In Drosophila and human, component 3 promoter of RISC (C3PO), a heteromeric complex, enhances RISC assembly and promotes RISC activity. Here, we report crystal structure of full-length Drosophila C3PO (E126Q), an inactive C3PO mutant displaying much weaker RNA binding ability, at 2.1 Å resolution. In addition, we also report the cryo-EM structures of full-length Drosophila C3PO (E126Q), C3PO (WT) and SUMO-C3PO (WT, sumo-TRAX + Translin) particles trapped at different conformations at 12, 19.7 and 12.8 Å resolutions, respectively. Crystal structure of C3PO (E126Q) displays a half-barrel architecture consisting of two Trax/Translin heterodimers, whereas cryo-EM structures of C3PO (E126Q), C3PO (WT) and SUMO-C3PO (WT) adopt a closed football-like shape with a hollow interior cavity. Remarkably, both cryo-EM structures of Drosophila C3PO (E126Q) and Drosophila SUMO-C3PO (WT) particles contain a wide side port (∼25 Å × ∼30 Å versus ∼15 Å × ∼20 Å) for RNA substrate entry and release, formed by a pair of anti-parallel packed long α1 helices of TRAX subunits. Notably, cryo-EM structure of SUMO-C3PO showed that four copies of extra densities belonging to N-terminal SUMO tag are located at the outside shell of SUMO-C3PO particle, which demonstrated that the stoichiometry of TRAX/Translin for the in vitro expressed and assembled full-length Drosophila-SUMO-C3PO particle is 4:4, suggesting Drosophila C3PO is composed by TRAX/translin at a ratio of 4:4. Remarkably, the comparison of the cryo-EM structures suggests that the C3PO side ports regulated by α1 helices of TRAX molecules are highly dynamic. Hence, we propose that C3PO particles could adopt a 'Dynamic Side Port' model to capture/digest nucleic acid duplex substrate and release the digested fragments through the dynamic side ports. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_6722.map.gz emd_6722.map.gz | 6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-6722-v30.xml emd-6722-v30.xml emd-6722.xml emd-6722.xml | 13.6 KB 13.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_6722_fsc.xml emd_6722_fsc.xml | 7.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_6722.png emd_6722.png | 46.6 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-6722 http://ftp.pdbj.org/pub/emdb/structures/EMD-6722 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-6722 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-6722 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_6722_validation.pdf.gz emd_6722_validation.pdf.gz | 78 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_6722_full_validation.pdf.gz emd_6722_full_validation.pdf.gz | 77 KB | 表示 | |
| XML形式データ |  emd_6722_validation.xml.gz emd_6722_validation.xml.gz | 494 B | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6722 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6722 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6722 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6722 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_6722.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_6722.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | cryo-EM structure of full-length C3PO (WT) adopt a closed football-like shape with a hollow interior cavity consisting of four Trax/Translin heterodimers. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.14 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : C3PO
| 全体 | 名称: C3PO |
|---|---|
| 要素 |
|
-超分子 #1: C3PO
| 超分子 | 名称: C3PO / タイプ: complex / ID: 1 / 親要素: 0 詳細: In Drosophila, component 3 promoter of RISC (C3PO)is a complex, consisting of 4 Trax/Translin heterodimers. |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種: 組換株: DH5a / 組換プラスミド: pET-Duet |
| 分子量 | 実験値: 290 KDa |
-超分子 #2: TRAX
| 超分子 | 名称: TRAX / タイプ: complex / ID: 2 / 親要素: 1 / 詳細: Translin-associated factor X |
|---|
-超分子 #3: Translin
| 超分子 | 名称: Translin / タイプ: complex / ID: 3 / 親要素: 1 / 詳細: A key component in C3PO complex |
|---|
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1 mg/mL | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.4 構成要素:
詳細: 20mM Tris (pH 7.4), 500mM NaCl, 1mM DTT and 2mM EDTA | |||||||||||||||
| グリッド | モデル: Quantifoil R2/2 / 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: FORMVAR / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR / 前処理 - 気圧: 101.325 kPa | |||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 298 K / 装置: FEI VITROBOT MARK IV / 詳細: blot for 5 seconds before pluning. | |||||||||||||||
| 詳細 | This sample was monodisperse. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI 12 |
|---|---|
| 温度 | 最低: 70.0 K / 最高: 70.0 K |
| 詳細 | Preliminary grid screening was performed manually. |
| 撮影 | フィルム・検出器のモデル: GATAN ULTRASCAN 4000 (4k x 4k) 撮影したグリッド数: 20 / 実像数: 500 / 平均露光時間: 1.0 sec. / 平均電子線量: 20.0 e/Å2 |
| 電子線 | 加速電圧: 120 kV / 電子線源: LAB6 |
| 電子光学系 | 最大 デフォーカス(補正後): 5.0 µm / 最小 デフォーカス(補正後): 1.2 µm / 倍率(補正後): 67000 / 照射モード: OTHER / 撮影モード: DARK FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 5.0 µm / 最小 デフォーカス(公称値): 1.2 µm / 倍率(公称値): 67000 |
| 試料ステージ | 試料ホルダーモデル: GATAN 626 SINGLE TILT LIQUID NITROGEN CRYO TRANSFER HOLDER ホルダー冷却材: NITROGEN |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: RECIPROCAL / プロトコル: AB INITIO MODEL / 温度因子: 300 |
|---|
 ムービー
ムービー コントローラー
コントローラー



 UCSF Chimera
UCSF Chimera








 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)