+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-6611 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
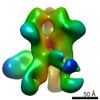
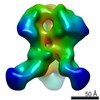
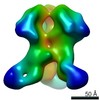
| タイトル | Cryo-electron microscopy structure of chimeric influenza cH5/1 hemagglutinin bound to stalk-targeting antibody, 6F12 | |||||||||
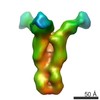
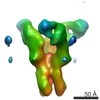
 マップデータ マップデータ | Tomographic subvolume average of virus-bound influenza cH5/1 HA in complex with the stalk-binding antibody, 6F12 | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Influenza / chimeric HA / universal vaccine candidate | |||||||||
| 生物種 |   unidentified influenza virus (インフルエンザウイルス) / unidentified influenza virus (インフルエンザウイルス) /  | |||||||||
| 手法 | サブトモグラム平均法 / クライオ電子顕微鏡法 / 解像度: 25.0 Å | |||||||||
 データ登録者 データ登録者 | Tran EEH / Podolsky KA / Bartesaghi A / Kuybeda O / Grandinetti G / Wohlbold TJ / Tan GS / Nachbagauer R / Palese P / Krammer F / Subramaniam S | |||||||||
 引用 引用 |  ジャーナル: mBio / 年: 2016 ジャーナル: mBio / 年: 2016タイトル: Cryo-electron Microscopy Structures of Chimeric Hemagglutinin Displayed on a Universal Influenza Vaccine Candidate. 著者: Erin E H Tran / Kira A Podolsky / Alberto Bartesaghi / Oleg Kuybeda / Giovanna Grandinetti / Teddy John Wohlbold / Gene S Tan / Raffael Nachbagauer / Peter Palese / Florian Krammer / Sriram Subramaniam /   要旨: Influenza viruses expressing chimeric hemagglutinins (HAs) are important tools in the quest for a universal vaccine. Using cryo-electron tomography, we have determined the structures of a chimeric HA ...Influenza viruses expressing chimeric hemagglutinins (HAs) are important tools in the quest for a universal vaccine. Using cryo-electron tomography, we have determined the structures of a chimeric HA variant that comprises an H1 stalk and an H5 globular head domain (cH5/1 HA) in native and antibody-bound states. We show that cH5/1 HA is structurally different from native HA, displaying a 60° rotation between the stalk and head groups, leading to a novel and unexpected "open" arrangement of HA trimers. cH5/1N1 viruses also display higher glycoprotein density than pH1N1 or H5N1 viruses, but despite these differences, antibodies that target either the stalk or head domains of hemagglutinins still bind to cH5/1 HA with the same consequences as those observed with native H1 or H5 HA. Our results show that a large range of structural plasticity can be tolerated in the chimeric spike scaffold without disrupting structural and geometric aspects of antibody binding. IMPORTANCE: Chimeric hemagglutinin proteins are set to undergo human clinical trials as a universal influenza vaccine candidate, yet no structural information for these proteins is available. Using ...IMPORTANCE: Chimeric hemagglutinin proteins are set to undergo human clinical trials as a universal influenza vaccine candidate, yet no structural information for these proteins is available. Using cryo-electron tomography, we report the first three-dimensional (3D) visualization of chimeric hemagglutinin proteins displayed on the surface of the influenza virus. We show that, unexpectedly, the chimeric hemagglutinin structure differs from those of naturally occurring hemagglutinins by displaying a more open head domain and a dramatically twisted head/stalk arrangement. Despite this unusual spatial relationship between head and stalk regions, virus preparations expressing the chimeric hemagglutinin are fully infectious and display a high glycoprotein density, which likely helps induction of a broadly protective immune response. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_6611.map.gz emd_6611.map.gz | 674.9 KB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-6611-v30.xml emd-6611-v30.xml emd-6611.xml emd-6611.xml | 12.2 KB 12.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_6611.png emd_6611.png | 399.8 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-6611 http://ftp.pdbj.org/pub/emdb/structures/EMD-6611 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-6611 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-6611 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_6611_validation.pdf.gz emd_6611_validation.pdf.gz | 78.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_6611_full_validation.pdf.gz emd_6611_full_validation.pdf.gz | 77.8 KB | 表示 | |
| XML形式データ |  emd_6611_validation.xml.gz emd_6611_validation.xml.gz | 494 B | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6611 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6611 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6611 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6611 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_6611.map.gz / 形式: CCP4 / 大きさ: 908.2 KB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_6611.map.gz / 形式: CCP4 / 大きさ: 908.2 KB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Tomographic subvolume average of virus-bound influenza cH5/1 HA in complex with the stalk-binding antibody, 6F12 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 これらの図は立方格子座標系で作成されたものです | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 4.1 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Tomographic subvolume average of virus-bound influenza cH5/1 HA i...
| 全体 | 名称: Tomographic subvolume average of virus-bound influenza cH5/1 HA in complex with the stalk-binding antibody, 6F12 |
|---|---|
| 要素 |
|
-超分子 #1000: Tomographic subvolume average of virus-bound influenza cH5/1 HA i...
| 超分子 | 名称: Tomographic subvolume average of virus-bound influenza cH5/1 HA in complex with the stalk-binding antibody, 6F12 タイプ: sample / ID: 1000 / Number unique components: 2 |
|---|
-分子 #1: cH5/1 hemagglutinin
| 分子 | 名称: cH5/1 hemagglutinin / タイプ: protein_or_peptide / ID: 1 / 集合状態: trimer / 組換発現: No / データベース: NCBI |
|---|---|
| 由来(天然) | 生物種:   unidentified influenza virus (インフルエンザウイルス) unidentified influenza virus (インフルエンザウイルス)株: cH5/1N1 |
-分子 #2: Immunoglobulin 6F12
| 分子 | 名称: Immunoglobulin 6F12 / タイプ: protein_or_peptide / ID: 2 / 組換発現: No / データベース: NCBI |
|---|---|
| 由来(天然) | 生物種:  |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | サブトモグラム平均法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 / 詳細: 25 mM Tris, 100 mM NaCl |
|---|---|
| グリッド | 詳細: Quantifoil Multi-A 200 mesh carbon grids |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / チャンバー内温度: 90 K / 装置: LEICA EM GP Timed resolved state: Virus was incubated with antibody for ~30 minutes on ice before solution was plunge-frozen 手法: Blot for 6 seconds before plunging. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: Gatan, Inc. エネルギーフィルター - エネルギー下限: 0.0 eV エネルギーフィルター - エネルギー上限: 20.0 eV |
| 日付 | 2015年4月16日 |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 実像数: 10 / 平均電子線量: 120 e/Å2 詳細: Every image is the average of four frames recorded by the direct electron detector. |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 23000 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 2.0 µm / 倍率(公称値): 64000 |
| 試料ステージ | 試料ホルダー: Liquid nitrogen-cooled 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER Tilt series - Axis1 - Min angle: -60 ° / Tilt series - Axis1 - Max angle: 60 ° |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 詳細 | Subtomograms were selected using an automatic selection program. |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 25.0 Å / 解像度の算出法: OTHER / ソフトウェア - 名称:  IMOD / 使用したサブトモグラム数: 134 IMOD / 使用したサブトモグラム数: 134 |
 ムービー
ムービー コントローラー
コントローラー











 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)





















