+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-6500 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Negative stain EM reconstruction of HIV-1 Env AMC008 SOSIP.v4 in complex with Fabs 35O22 and PGV04 | |||||||||
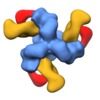
 マップデータ マップデータ | Reconstruction of HIV-1 Env AMC008 SOSIP.v4 in complex with broadly-neutralzing antibody Fabs 35O22 and PGV04 | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | HIV-1 / Env / SOSIP / broadly-neutralizing antibody / trimer | |||||||||
| 生物種 |   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) / Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) /  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / ネガティブ染色法 / 解像度: 15.0 Å | |||||||||
 データ登録者 データ登録者 | de Taeye SW / Ozorowski G / de la Pena AT / Guttman M / Julien J-P / van den Kerkhof TLGM / Burger J / Pritchard LK / Pugach P / Yasmeen A ...de Taeye SW / Ozorowski G / de la Pena AT / Guttman M / Julien J-P / van den Kerkhof TLGM / Burger J / Pritchard LK / Pugach P / Yasmeen A / Bontjer I / Torres JL / Arendt H / DeStefano J / Koff WC / Schuitemaker H / Eggink D / Berkhout B / Dean H / LaBranche C / Crispin M / Montefiori DC / Klasse PJ / Lee KK / Moore JP / Wilson IA / Ward AB / Sanders RW | |||||||||
 引用 引用 |  ジャーナル: Cell / 年: 2015 ジャーナル: Cell / 年: 2015タイトル: Immunogenicity of Stabilized HIV-1 Envelope Trimers with Reduced Exposure of Non-neutralizing Epitopes. 著者: Steven W de Taeye / Gabriel Ozorowski / Alba Torrents de la Peña / Miklos Guttman / Jean-Philippe Julien / Tom L G M van den Kerkhof / Judith A Burger / Laura K Pritchard / Pavel Pugach / ...著者: Steven W de Taeye / Gabriel Ozorowski / Alba Torrents de la Peña / Miklos Guttman / Jean-Philippe Julien / Tom L G M van den Kerkhof / Judith A Burger / Laura K Pritchard / Pavel Pugach / Anila Yasmeen / Jordan Crampton / Joyce Hu / Ilja Bontjer / Jonathan L Torres / Heather Arendt / Joanne DeStefano / Wayne C Koff / Hanneke Schuitemaker / Dirk Eggink / Ben Berkhout / Hansi Dean / Celia LaBranche / Shane Crotty / Max Crispin / David C Montefiori / P J Klasse / Kelly K Lee / John P Moore / Ian A Wilson / Andrew B Ward / Rogier W Sanders /    要旨: The envelope glycoprotein trimer mediates HIV-1 entry into cells. The trimer is flexible, fluctuating between closed and more open conformations and sometimes sampling the fully open, CD4-bound form. ...The envelope glycoprotein trimer mediates HIV-1 entry into cells. The trimer is flexible, fluctuating between closed and more open conformations and sometimes sampling the fully open, CD4-bound form. We hypothesized that conformational flexibility and transient exposure of non-neutralizing, immunodominant epitopes could hinder the induction of broadly neutralizing antibodies (bNAbs). We therefore modified soluble Env trimers to stabilize their closed, ground states. The trimer variants were indeed stabilized in the closed conformation, with a reduced ability to undergo receptor-induced conformational changes and a decreased exposure of non-neutralizing V3-directed antibody epitopes. In rabbits, the stabilized trimers induced similar autologous Tier-1B or Tier-2 NAb titers to those elicited by the corresponding wild-type trimers but lower levels of V3-directed Tier-1A NAbs. Stabilized, closed trimers might therefore be useful components of vaccines aimed at inducing bNAbs. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_6500.map.gz emd_6500.map.gz | 14.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-6500-v30.xml emd-6500-v30.xml emd-6500.xml emd-6500.xml | 14.5 KB 14.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_6500.tif emd_6500.tif | 156.3 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-6500 http://ftp.pdbj.org/pub/emdb/structures/EMD-6500 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-6500 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-6500 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_6500_validation.pdf.gz emd_6500_validation.pdf.gz | 79.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_6500_full_validation.pdf.gz emd_6500_full_validation.pdf.gz | 78.7 KB | 表示 | |
| XML形式データ |  emd_6500_validation.xml.gz emd_6500_validation.xml.gz | 493 B | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6500 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6500 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6500 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6500 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_6500.map.gz / 形式: CCP4 / 大きさ: 15.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_6500.map.gz / 形式: CCP4 / 大きさ: 15.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Reconstruction of HIV-1 Env AMC008 SOSIP.v4 in complex with broadly-neutralzing antibody Fabs 35O22 and PGV04 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.57 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : HIV-1 Env AMC008 SOSIP.v4 in complex with broadly neutralizing an...
| 全体 | 名称: HIV-1 Env AMC008 SOSIP.v4 in complex with broadly neutralizing antibody Fabs 35O22 and PGV04 |
|---|---|
| 要素 |
|
-超分子 #1000: HIV-1 Env AMC008 SOSIP.v4 in complex with broadly neutralizing an...
| 超分子 | 名称: HIV-1 Env AMC008 SOSIP.v4 in complex with broadly neutralizing antibody Fabs 35O22 and PGV04 タイプ: sample / ID: 1000 詳細: All components were purified by size-exclusion chromatography prior to complex formation. 集合状態: 3 of each Fab bound to a trimer of HIV-1 Env SOSIP.v4 Number unique components: 3 |
|---|---|
| 分子量 | 理論値: 720 KDa 手法: Estimation based on average molecular weight of Fab and average mass of SOSIP trimer including glycan mass |
-分子 #1: HIV-1 AMC008 Env SOSIP.v4
| 分子 | 名称: HIV-1 AMC008 Env SOSIP.v4 / タイプ: protein_or_peptide / ID: 1 / Name.synonym: HIV-1 gp140 / コピー数: 1 / 集合状態: Trimer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス)株: AMC008 |
| 分子量 | 理論値: 420 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) / 組換細胞: HEK293F Homo sapiens (ヒト) / 組換細胞: HEK293F |
-分子 #2: Anti-HIV-1 antibody PGV04 Fab
| 分子 | 名称: Anti-HIV-1 antibody PGV04 Fab / タイプ: protein_or_peptide / ID: 2 / コピー数: 3 / 集合状態: Heterodimer of light and heavy chain / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) / 別称: Human Homo sapiens (ヒト) / 別称: Human |
| 分子量 | 理論値: 50 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) / 組換細胞: HEK293F Homo sapiens (ヒト) / 組換細胞: HEK293F |
-分子 #3: Anti-HIV-1 antibody 35O22 Fab
| 分子 | 名称: Anti-HIV-1 antibody 35O22 Fab / タイプ: protein_or_peptide / ID: 3 / コピー数: 3 / 集合状態: Heterodimer of light and heavy chain / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) / 別称: Human Homo sapiens (ヒト) / 別称: Human |
| 分子量 | 理論値: 50 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) / 組換細胞: HEK293F Homo sapiens (ヒト) / 組換細胞: HEK293F |
-実験情報
-構造解析
| 手法 | ネガティブ染色法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.03 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 / 詳細: 50 mM Tris-HCl, 150 mM NaCl |
| 染色 | タイプ: NEGATIVE 詳細: Grids were stained with 2% w/v uranyl formate for 60 seconds. |
| グリッド | 詳細: 400 mesh copper grid with thin carbon support, glow-discharged |
| 凍結 | 凍結剤: NONE / 装置: OTHER |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | OTHER |
|---|---|
| アライメント法 | Legacy - 非点収差: Objective lens astigmatism was corrected at 92,000 times magnification. |
| 詳細 | Tilt series of -50, -40, -30, -20, -10, 0 degrees |
| 日付 | 2015年2月19日 |
| 撮影 | カテゴリ: CCD / フィルム・検出器のモデル: FEI CETA (4k x 4k) / 実像数: 152 / 平均電子線量: 25 e/Å2 |
| Tilt angle max | 0 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 1.0 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 92000 |
| 試料ステージ | 試料ホルダーモデル: SIDE ENTRY, EUCENTRIC / Tilt angle min: -50 |
- 画像解析
画像解析
| 詳細 | Particles were selected using an automatic selection program and aligned into class averages using Iterative MSA/MRA. Classes containing fully decorated (3 molecules of each Fab per trimer) complexes were used to generate a common lines model that was later refined using particles from those classes. |
|---|---|
| 最終 再構成 | アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 15.0 Å / 解像度の算出法: OTHER / ソフトウェア - 名称: EMAN2, SPARX / 使用した粒子像数: 24522 |
 ムービー
ムービー コントローラー
コントローラー









 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)





















