+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-6387 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | A new view of essential vertex proteins on herpesvirus capsids observed inside intact virions | |||||||||
 マップデータ マップデータ | Reconstruction of pseudorabies virus capsid imaged inside virions | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Pseudorabies virus / PRV / capsid / virion / CVSC / pUL17 / pUL25 / pUL36 | |||||||||
| 生物種 |   Suid herpesvirus 1 (ヘルペスウイルス) Suid herpesvirus 1 (ヘルペスウイルス) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 7.2 Å | |||||||||
 データ登録者 データ登録者 | Huet A / Makhov AM / Huffman JB / Vos M / Homa FL / Conway JF | |||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2016 ジャーナル: Nat Struct Mol Biol / 年: 2016タイトル: Extensive subunit contacts underpin herpesvirus capsid stability and interior-to-exterior allostery. 著者: Alexis Huet / Alexander M Makhov / Jamie B Huffman / Matthijn Vos / Fred L Homa / James F Conway /   要旨: The herpesvirus capsid is a complex protein assembly that includes hundreds of copies of four major subunits and lesser numbers of several minor proteins, all of which are essential for infectivity. ...The herpesvirus capsid is a complex protein assembly that includes hundreds of copies of four major subunits and lesser numbers of several minor proteins, all of which are essential for infectivity. Cryo-electron microscopy is uniquely suited for studying interactions that govern the assembly and function of such large functional complexes. Here we report two high-quality capsid structures, from human herpes simplex virus type 1 (HSV-1) and the animal pseudorabies virus (PRV), imaged inside intact virions at ~7-Å resolution. From these, we developed a complete model of subunit and domain organization and identified extensive networks of subunit contacts that underpin capsid stability and form a pathway that may signal the completion of DNA packaging from the capsid interior to outer surface, thereby initiating nuclear egress. Differences in the folding and orientation of subunit domains between herpesvirus capsids suggest that common elements have been modified for specific functions. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_6387.map.gz emd_6387.map.gz | 389.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-6387-v30.xml emd-6387-v30.xml emd-6387.xml emd-6387.xml | 10.9 KB 10.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_6387.tif emd_6387.tif emd_6387_1.tif emd_6387_1.tif | 5.2 MB 2 MB | ||
| その他 |  EMD-6387_1.tif EMD-6387_1.tif | 2 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-6387 http://ftp.pdbj.org/pub/emdb/structures/EMD-6387 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-6387 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-6387 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_6387_validation.pdf.gz emd_6387_validation.pdf.gz | 77.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_6387_full_validation.pdf.gz emd_6387_full_validation.pdf.gz | 76.9 KB | 表示 | |
| XML形式データ |  emd_6387_validation.xml.gz emd_6387_validation.xml.gz | 494 B | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6387 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6387 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6387 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6387 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_6387.map.gz / 形式: CCP4 / 大きさ: 782.7 MB / タイプ: IMAGE STORED AS SIGNED INTEGER (2 BYTES) ダウンロード / ファイル: emd_6387.map.gz / 形式: CCP4 / 大きさ: 782.7 MB / タイプ: IMAGE STORED AS SIGNED INTEGER (2 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Reconstruction of pseudorabies virus capsid imaged inside virions | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 これらの図は立方格子座標系で作成されたものです | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.16 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
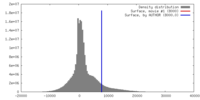
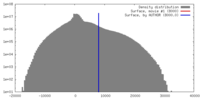
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-その他
- 試料の構成要素
試料の構成要素
-全体 : Capsid of pseudorabies virus imaged in intact virions
| 全体 | 名称: Capsid of pseudorabies virus imaged in intact virions |
|---|---|
| 要素 |
|
-超分子 #1000: Capsid of pseudorabies virus imaged in intact virions
| 超分子 | 名称: Capsid of pseudorabies virus imaged in intact virions タイプ: sample / ID: 1000 / 集合状態: Icosahedral T=16 / Number unique components: 1 |
|---|---|
| 分子量 | 実験値: 200 MDa / 理論値: 200 MDa |
-超分子 #1: Suid herpesvirus 1
| 超分子 | 名称: Suid herpesvirus 1 / タイプ: virus / ID: 1 / 詳細: Native Becker / NCBI-ID: 10345 / 生物種: Suid herpesvirus 1 / ウイルスタイプ: VIRION / ウイルス・単離状態: SPECIES / ウイルス・エンベロープ: Yes / ウイルス・中空状態: No |
|---|---|
| 宿主 | 生物種:  |
| Host system | 生物種:  Chlorocebus aethiops (ミドリザル) / 組換細胞: green monkey kidney (Vero) cells Chlorocebus aethiops (ミドリザル) / 組換細胞: green monkey kidney (Vero) cells |
| 分子量 | 実験値: 200 MDa / 理論値: 200 MDa |
| ウイルス殻 | Shell ID: 1 / 直径: 1250 Å / T番号(三角分割数): 16 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| グリッド | 詳細: Quantifoil R2/1 |
|---|---|
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 90 % / チャンバー内温度: 120 K / 装置: FEI VITROBOT MARK IV / 手法: 6 second blot |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| アライメント法 | Legacy - 非点収差: Objective lens astigmatism was corrected at 75,000x magnification. |
| 詳細 | Nanoprobe |
| 日付 | 2013年4月14日 |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: FEI FALCON II (4k x 4k) デジタル化 - サンプリング間隔: 14 µm / 実像数: 32956 / 平均電子線量: 30 e/Å2 / 詳細: 2 second exposures; no use of movie mode |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 81000 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 5.0 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 75000 |
| 試料ステージ | 試料ホルダー: Nitrogen cooled 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 詳細 | Manual selection, processing with AUTO3DEM |
|---|---|
| CTF補正 | 詳細: By micrograph |
| 最終 再構成 | アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 7.2 Å / 解像度の算出法: OTHER / ソフトウェア - 名称: AUTO3DEM, CTFFIND3 詳細: Final map calculated from 25637 images of capsids inside virions. Resolution was verified by comparing cryoEM data with band-limited renditions of the HSV VP5 upper domain fragment, PDB 1NO7. 使用した粒子像数: 13242 |
-原子モデル構築 1
| 初期モデル | PDB ID: Chain - Chain ID: A |
|---|---|
| ソフトウェア | 名称:  Chimera Chimera |
| 詳細 | Manual docking |
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
 ムービー
ムービー コントローラー
コントローラー













 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)