+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of the urea-treated empty bacteriophage T5 portal complex | |||||||||||||||
 マップデータ マップデータ | ||||||||||||||||
 試料 試料 |
| |||||||||||||||
 キーワード キーワード | Complex / VIRAL PROTEIN | |||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報cytoplasmic icosahedral capsid assembly / symbiont genome ejection through host cell envelope, long flexible tail mechanism / viral capsid assembly / virion component / viral capsid / host cell cytoplasm 類似検索 - 分子機能 | |||||||||||||||
| 生物種 |  Escherichia phage T5 (ファージ) Escherichia phage T5 (ファージ) | |||||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.0 Å | |||||||||||||||
 データ登録者 データ登録者 | Peng YN / Liu HR | |||||||||||||||
| 資金援助 |  中国, 4件 中国, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: Int J Mol Sci / 年: 2024 ジャーナル: Int J Mol Sci / 年: 2024タイトル: Structures of Mature and Urea-Treated Empty Bacteriophage T5: Insights into Siphophage Infection and DNA Ejection. 著者: Yuning Peng / Huanrong Tang / Hao Xiao / Wenyuan Chen / Jingdong Song / Jing Zheng / Hongrong Liu /  要旨: T5 is a siphophage that has been extensively studied by structural and biochemical methods. However, the complete in situ structures of T5 before and after DNA ejection remain unknown. In this study, ...T5 is a siphophage that has been extensively studied by structural and biochemical methods. However, the complete in situ structures of T5 before and after DNA ejection remain unknown. In this study, we used cryo-electron microscopy (cryo-EM) to determine the structures of mature T5 (a laboratory-adapted, fiberless T5 mutant) and urea-treated empty T5 (lacking the tip complex) at near-atomic resolutions. Atomic models of the head, connector complex, tail tube, and tail tip were built for mature T5, and atomic models of the connector complex, comprising the portal protein pb7, adaptor protein p144, and tail terminator protein p142, were built for urea-treated empty T5. Our findings revealed that the aforementioned proteins did not undergo global conformational changes before and after DNA ejection, indicating that these structural features were conserved among most myophages and siphophages. The present study elucidates the underlying mechanisms of siphophage infection and DNA ejection. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_60695.map.gz emd_60695.map.gz | 164.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-60695-v30.xml emd-60695-v30.xml emd-60695.xml emd-60695.xml | 17.3 KB 17.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_60695.png emd_60695.png | 62.1 KB | ||
| Filedesc metadata |  emd-60695.cif.gz emd-60695.cif.gz | 5.6 KB | ||
| その他 |  emd_60695_half_map_1.map.gz emd_60695_half_map_1.map.gz emd_60695_half_map_2.map.gz emd_60695_half_map_2.map.gz | 164.9 MB 164.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-60695 http://ftp.pdbj.org/pub/emdb/structures/EMD-60695 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-60695 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-60695 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_60695_validation.pdf.gz emd_60695_validation.pdf.gz | 1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_60695_full_validation.pdf.gz emd_60695_full_validation.pdf.gz | 1 MB | 表示 | |
| XML形式データ |  emd_60695_validation.xml.gz emd_60695_validation.xml.gz | 15.2 KB | 表示 | |
| CIF形式データ |  emd_60695_validation.cif.gz emd_60695_validation.cif.gz | 18.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-60695 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-60695 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-60695 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-60695 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  9imvMC  8zviC  9ilpC  9ilvC 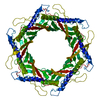 9imhC  9inyC  9iozC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_60695.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_60695.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.3 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #1
| ファイル | emd_60695_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_60695_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Escherichia phage T5
| 全体 | 名称:  Escherichia phage T5 (ファージ) Escherichia phage T5 (ファージ) |
|---|---|
| 要素 |
|
-超分子 #1: Escherichia phage T5
| 超分子 | 名称: Escherichia phage T5 / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: all / NCBI-ID: 2695836 / 生物種: Escherichia phage T5 / ウイルスタイプ: VIRION / ウイルス・単離状態: SPECIES / ウイルス・エンベロープ: No / ウイルス・中空状態: No |
|---|
-分子 #1: Portal protein pb7
| 分子 | 名称: Portal protein pb7 / タイプ: protein_or_peptide / ID: 1 / コピー数: 12 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Escherichia phage T5 (ファージ) Escherichia phage T5 (ファージ) |
| 分子量 | 理論値: 45.137234 KDa |
| 配列 | 文字列: MGFKSWITEK LNPGQRIIRD MEPVSHRTNR KPFTTGQAYS KIEILNRTAN MVIDSAAECS YTVGDKYNIV TYANGVKTKT LDTLLNVRP NPFMDISTFR RLVVTDLLFE GCAYIYWDGT SLYHVPAALM QVEADANKFI KKFIFNNQIN YRVDEIIFIK D NSYVCGTN ...文字列: MGFKSWITEK LNPGQRIIRD MEPVSHRTNR KPFTTGQAYS KIEILNRTAN MVIDSAAECS YTVGDKYNIV TYANGVKTKT LDTLLNVRP NPFMDISTFR RLVVTDLLFE GCAYIYWDGT SLYHVPAALM QVEADANKFI KKFIFNNQIN YRVDEIIFIK D NSYVCGTN SQISGQSRVA TVIDSLEKRS KMLNFKEKFL DNGTVIGLIL ETDEILNKKL RERKQEELQL DYNPSTGQSS VL ILDGGMK AKPYSQISSF KDLDFKEDIE GFNKSICLAF GVPQVLLDGG NNANIRPNIE LFYYMTIIPM LNKLTSSLTF FFG YKITPN TKEVAALTPD KEAEAKHLTS LVNNGIITGN EARSELNLEP LDDEQMNKIR IPANVAGSAT GVSGQEGGRP KGST EGD UniProtKB: Portal protein |
-分子 #2: Head completion protein
| 分子 | 名称: Head completion protein / タイプ: protein_or_peptide / ID: 2 / コピー数: 12 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Escherichia phage T5 (ファージ) Escherichia phage T5 (ファージ) |
| 分子量 | 理論値: 19.231912 KDa |
| 配列 | 文字列: MQIITAEDYR LYGGLKRPEL ESGVEVMITA ANALITSLLG MDDADAVDQL ITTKPTRKKY FLSSPSATSV TKMTINDKEI DPEQYKLYS DGVILLKFNP PEGYMDVEYT QGGFNPMPED LKLAACMLVD HWHKQDYRQA RTIGGETVTF NNTKSGIPEH I RTIIEVYR RV UniProtKB: Head completion protein |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 32.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.2 µm / 最小 デフォーカス(公称値): 1.6 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー










 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)