+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | 3D structure of Y-50 TCR-TMM-CD1b ternary complex | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | IMMUNE SYSTEM | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報exogenous lipid antigen binding / antigen processing and presentation, endogenous lipid antigen via MHC class Ib / lipopeptide binding / endogenous lipid antigen binding / antigen processing and presentation, exogenous lipid antigen via MHC class Ib / negative regulation of receptor binding / early endosome lumen / Nef mediated downregulation of MHC class I complex cell surface expression / DAP12 interactions / transferrin transport ...exogenous lipid antigen binding / antigen processing and presentation, endogenous lipid antigen via MHC class Ib / lipopeptide binding / endogenous lipid antigen binding / antigen processing and presentation, exogenous lipid antigen via MHC class Ib / negative regulation of receptor binding / early endosome lumen / Nef mediated downregulation of MHC class I complex cell surface expression / DAP12 interactions / transferrin transport / cellular response to iron ion / Endosomal/Vacuolar pathway / Antigen Presentation: Folding, assembly and peptide loading of class I MHC / peptide antigen assembly with MHC class II protein complex / antigen processing and presentation of exogenous protein antigen via MHC class Ib, TAP-dependent / cellular response to iron(III) ion / MHC class II protein complex / negative regulation of forebrain neuron differentiation / ER to Golgi transport vesicle membrane / peptide antigen assembly with MHC class I protein complex / regulation of erythrocyte differentiation / regulation of iron ion transport / response to molecule of bacterial origin / MHC class I peptide loading complex / HFE-transferrin receptor complex / T cell mediated cytotoxicity / antigen processing and presentation of endogenous peptide antigen via MHC class I / positive regulation of T cell cytokine production / antigen processing and presentation of exogenous peptide antigen via MHC class II / positive regulation of immune response / MHC class I protein complex / positive regulation of T cell activation / peptide antigen binding / positive regulation of receptor-mediated endocytosis / negative regulation of neurogenesis / positive regulation of T cell mediated cytotoxicity / cellular response to nicotine / multicellular organismal-level iron ion homeostasis / Modulation by Mtb of host immune system / specific granule lumen / phagocytic vesicle membrane / recycling endosome membrane / Immunoregulatory interactions between a Lymphoid and a non-Lymphoid cell / Interferon gamma signaling / negative regulation of epithelial cell proliferation / MHC class II protein complex binding / late endosome membrane / sensory perception of smell / positive regulation of cellular senescence / tertiary granule lumen / DAP12 signaling / T cell differentiation in thymus / ER-Phagosome pathway / negative regulation of neuron projection development / protein refolding / early endosome membrane / protein homotetramerization / adaptive immune response / amyloid fibril formation / intracellular iron ion homeostasis / learning or memory / endosome membrane / immune response / Amyloid fiber formation / endoplasmic reticulum lumen / Golgi membrane / lysosomal membrane / external side of plasma membrane / focal adhesion / intracellular membrane-bounded organelle / Neutrophil degranulation / SARS-CoV-2 activates/modulates innate and adaptive immune responses / structural molecule activity / cell surface / endoplasmic reticulum / Golgi apparatus / protein homodimerization activity / extracellular space / extracellular exosome / extracellular region / identical protein binding / membrane / plasma membrane / cytosol 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.18 Å | |||||||||
 データ登録者 データ登録者 | Asa M / Hirose M / Nagae M / Yamasaki S / Kato T | |||||||||
| 資金援助 |  日本, 1件 日本, 1件
| |||||||||
 引用 引用 |  ジャーナル: J Clin Invest / 年: 2024 ジャーナル: J Clin Invest / 年: 2024タイトル: A conserved human CD4+ T cell subset recognizing the mycobacterial adjuvant trehalose monomycolate. 著者: Yuki Sakai / Minori Asa / Mika Hirose / Wakana Kusuhara / Nagatoshi Fujiwara / Hiroto Tamashima / Takahiro Ikazaki / Shiori Oka / Kota Kuraba / Kentaro Tanaka / Takashi Yoshiyama / Masamichi ...著者: Yuki Sakai / Minori Asa / Mika Hirose / Wakana Kusuhara / Nagatoshi Fujiwara / Hiroto Tamashima / Takahiro Ikazaki / Shiori Oka / Kota Kuraba / Kentaro Tanaka / Takashi Yoshiyama / Masamichi Nagae / Yoshihiko Hoshino / Daisuke Motooka / Ildiko Van Rhijn / Xiuyuan Lu / Eri Ishikawa / D Branch Moody / Takayuki Kato / Shinsuke Inuki / Go Hirai / Sho Yamasaki /    要旨: Mycobacterium tuberculosis causes human tuberculosis (TB). As mycobacteria are protected by a thick lipid cell wall, humans have developed immune responses against diverse mycobacterial lipids. Most ...Mycobacterium tuberculosis causes human tuberculosis (TB). As mycobacteria are protected by a thick lipid cell wall, humans have developed immune responses against diverse mycobacterial lipids. Most of these immunostimulatory lipids are known as adjuvants acting through innate immune receptors, such as C-type lectin receptors. Although a few mycobacterial lipid antigens activate unconventional T cells, the antigenicity of most adjuvantic lipids is unknown. Here, we identified that trehalose monomycolate (TMM), an abundant mycobacterial adjuvant, activated human T cells bearing a unique αβ T cell receptor (αβTCR). This recognition was restricted by CD1b, a monomorphic antigen-presenting molecule conserved in primates but not mice. Single-cell TCR-RNA-Seq using newly established CD1b-TMM tetramers revealed that TMM-specific T cells were present as CD4+ effector memory T cells in the periphery of uninfected donors but expressed IFN-γ, TNF, and anti-mycobacterial effectors upon TMM stimulation. TMM-specific T cells were detected in cord blood and PBMCs of donors without bacillus Calmette-Guérin vaccination but were expanded in patients with active TB. A cryo-electron microscopy study of CD1b-TMM-TCR complexes revealed unique antigen recognition by conserved features of TCRs, positively charged CDR3α, and long CDR3β regions. These results indicate that humans have a commonly shared and preformed CD4+ T cell subset recognizing a typical mycobacterial adjuvant as an antigen. Furthermore, the dual role of TMM justifies reconsideration of the mechanism of action of adjuvants. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_60321.map.gz emd_60321.map.gz | 136.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-60321-v30.xml emd-60321-v30.xml emd-60321.xml emd-60321.xml | 24.9 KB 24.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_60321.png emd_60321.png | 42.4 KB | ||
| Filedesc metadata |  emd-60321.cif.gz emd-60321.cif.gz | 7.3 KB | ||
| その他 |  emd_60321_half_map_1.map.gz emd_60321_half_map_1.map.gz emd_60321_half_map_2.map.gz emd_60321_half_map_2.map.gz | 254.9 MB 254.9 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-60321 http://ftp.pdbj.org/pub/emdb/structures/EMD-60321 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-60321 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-60321 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_60321_validation.pdf.gz emd_60321_validation.pdf.gz | 977.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_60321_full_validation.pdf.gz emd_60321_full_validation.pdf.gz | 977.6 KB | 表示 | |
| XML形式データ |  emd_60321_validation.xml.gz emd_60321_validation.xml.gz | 16.5 KB | 表示 | |
| CIF形式データ |  emd_60321_validation.cif.gz emd_60321_validation.cif.gz | 19.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-60321 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-60321 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-60321 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-60321 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8zoxMC  8xubC  8zo4C M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_60321.map.gz / 形式: CCP4 / 大きさ: 274.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_60321.map.gz / 形式: CCP4 / 大きさ: 274.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.675 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #1
| ファイル | emd_60321_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
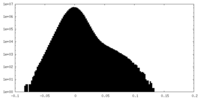
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_60321_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Ternary complex of Y-50TCR,TMM and CD1b
| 全体 | 名称: Ternary complex of Y-50TCR,TMM and CD1b |
|---|---|
| 要素 |
|
-超分子 #1: Ternary complex of Y-50TCR,TMM and CD1b
| 超分子 | 名称: Ternary complex of Y-50TCR,TMM and CD1b / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#4 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: T-cell surface glycoprotein CD1b
| 分子 | 名称: T-cell surface glycoprotein CD1b / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 31.00599 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MASEHAFQGP TSFHVIQTSS FTNSTWAQTQ GSGWLDDLQI HGWDSDSGTA IFLKPWSKGN FSDKEVAELE EIFRVYIFGF AREVQDFAG DFQMKYPFEI QGIAGCELHS GGAIVSFLRG ALGGLDFLSV KNASCVPSPE GGSRAQKFCA LIIQYQGIME T VRILLYET ...文字列: MASEHAFQGP TSFHVIQTSS FTNSTWAQTQ GSGWLDDLQI HGWDSDSGTA IFLKPWSKGN FSDKEVAELE EIFRVYIFGF AREVQDFAG DFQMKYPFEI QGIAGCELHS GGAIVSFLRG ALGGLDFLSV KNASCVPSPE GGSRAQKFCA LIIQYQGIME T VRILLYET CPRYLLGVLN AGKADLQRQV KPEAWLSSGP SPGPGRLQLV CHVSGFYPKP VWVMWMRGEQ EQQGTQLGDI LP NANWTWY LRATLDVADG EAAGLSCRVK HSSLEGQDII LYW UniProtKB: T-cell surface glycoprotein CD1b |
-分子 #2: Beta-2-microglobulin
| 分子 | 名称: Beta-2-microglobulin / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 11.879356 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MIQRTPKIQV YSRHPAENGK SNFLNCYVSG FHPSDIEVDL LKNGERIEKV EHSDLSFSKD WSFYLLYYTE FTPTEKDEYA CRVNHVTLS QPKIVKWDRD M UniProtKB: Beta-2-microglobulin |
-分子 #3: Y-50 TCR alpha
| 分子 | 名称: Y-50 TCR alpha / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 23.14266 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: AQKITQTQPG MFVQEKEAVT LDCTYDTSDQ SYGLFWYKQP SSGEMIFLIY QGSYDEQNAT EGRYSLNFQK ARKSANLVIS ASQLGDSAM YFCAMRRNTG RRALTFGSGT RLQVQPNIQN PDPAVYQLRD SKSSDKFVCL FTDFDSQINV SQSKDSDVYI T DKCVLDMR ...文字列: AQKITQTQPG MFVQEKEAVT LDCTYDTSDQ SYGLFWYKQP SSGEMIFLIY QGSYDEQNAT EGRYSLNFQK ARKSANLVIS ASQLGDSAM YFCAMRRNTG RRALTFGSGT RLQVQPNIQN PDPAVYQLRD SKSSDKFVCL FTDFDSQINV SQSKDSDVYI T DKCVLDMR SMDFKSNSAV AWSNKSDFTC ANAFNNSIIP EDTFFPS |
-分子 #4: Y-50 TCR beta
| 分子 | 名称: Y-50 TCR beta / タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 28.191834 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: DTEVTQTPKH LVMGMTNKKS LKCEQHMGHR AMYWYKQKAK KPPELMFVYS YEKLSINESV PSRFSPECPN SSLLNLHLHA LQPEDSALY LCASSHLGLA GGIDGTDTQY FGPGTRLTVL EDLKNVFPPE VAVFEPSKAE ISRTQKATLV CLATGFYPPH V ELSWWVNG ...文字列: DTEVTQTPKH LVMGMTNKKS LKCEQHMGHR AMYWYKQKAK KPPELMFVYS YEKLSINESV PSRFSPECPN SSLLNLHLHA LQPEDSALY LCASSHLGLA GGIDGTDTQY FGPGTRLTVL EDLKNVFPPE VAVFEPSKAE ISRTQKATLV CLATGFYPPH V ELSWWVNG KEVHDGVCTD PQPLKEQPAL NDSRYALSSR LRVSATFWQD PRNHFRCQVQ FYGLSENDEW TQDRAKPVTQ IV SAEAWGR AD |
-分子 #5: TETRACOSYL PALMITATE
| 分子 | 名称: TETRACOSYL PALMITATE / タイプ: ligand / ID: 5 / コピー数: 1 / 式: 6UL |
|---|---|
| 分子量 | 理論値: 593.062 Da |
| Chemical component information |  ChemComp-6UL: |
-分子 #6: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 6 / コピー数: 3 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-分子 #7: alpha-D-glucopyranose
| 分子 | 名称: alpha-D-glucopyranose / タイプ: ligand / ID: 7 / コピー数: 1 / 式: GLC |
|---|---|
| 分子量 | 理論値: 180.156 Da |
| Chemical component information |  ChemComp-GLC: |
-分子 #8: [(2R,3S,4S,5R,6S)-3,4,5,6-tetrakis(oxidanyl)oxan-2-yl]methyl (2R,...
| 分子 | 名称: [(2R,3S,4S,5R,6S)-3,4,5,6-tetrakis(oxidanyl)oxan-2-yl]methyl (2R,3R)-3-oxidanyl-2-tetradecyl-octadecanoate タイプ: ligand / ID: 8 / コピー数: 1 / 式: A1L2B |
|---|---|
| 分子量 | 理論値: 658.989 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1 mg/mL |
|---|---|
| 緩衝液 | pH: 8 |
| グリッド | モデル: Quantifoil R0.6/1 / 材質: GOLD / メッシュ: 200 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. / 前処理 - 雰囲気: AIR |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 温度 | 最低: 78.2 K / 最高: 78.2 K |
| 特殊光学系 | 球面収差補正装置: CEOS corrector was used. / エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) デジタル化 - サイズ - 横: 5760 pixel / デジタル化 - サイズ - 縦: 4092 pixel / 撮影したグリッド数: 1 / 実像数: 8533 / 平均露光時間: 3.169 sec. / 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 最大 デフォーカス(補正後): 2.036 µm / 最小 デフォーカス(補正後): 0.521 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 0.06 mm / 最大 デフォーカス(公称値): 1.8 µm / 最小 デフォーカス(公称値): 0.6 µm / 倍率(公称値): 105000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー






 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)