+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-5945 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Pyruvate Carboxylase tetramer in asymmetric architecture | |||||||||
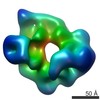
 マップデータ マップデータ | CryoEM of tetrameric Pyruvate Carboxylase in asymmetric architecture. Calculated after sorting of a catalytically active population. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | enzyme / tetrameric / biotin carboxylase | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 11.5 Å | |||||||||
 データ登録者 データ登録者 | Lasso G / Yu LPC / Gil D / Lazaro M / Tong L / Valle M | |||||||||
 引用 引用 |  ジャーナル: Structure / 年: 2014 ジャーナル: Structure / 年: 2014タイトル: Functional conformations for pyruvate carboxylase during catalysis explored by cryoelectron microscopy. 著者: Gorka Lasso / Linda P C Yu / David Gil / Melisa Lázaro / Liang Tong / Mikel Valle /   要旨: The tetrameric enzyme pyruvate carboxylase (PC), a biotin-dependent carboxylase, produces oxaloacetate by two consecutive reactions that take place in distant active sites. Previous crystal ...The tetrameric enzyme pyruvate carboxylase (PC), a biotin-dependent carboxylase, produces oxaloacetate by two consecutive reactions that take place in distant active sites. Previous crystal structures revealed two different configurations for PC tetramers, the so-called symmetric and asymmetric, which were understood as characteristic molecular architectures for PC from different organisms. We have analyzed PC samples from Staphylococcus aureus while the enzyme generates oxaloacetate, expecting PC tetramers to display the conformational landscape relevant for its functioning. Using cryoelectron microscopy (cryo-EM) and sorting techniques, we detect previously defined symmetric and asymmetric architectures, demonstrating that PC maps both arrangements by large conformational changes. Furthermore, we observe that each configuration is coupled to one of the two consecutive enzymatic reactions. The findings describe the structural transitions relevant for the allosteric control of the multifunctional PC and demonstrate that by cryo-EM and classification, we can characterize freely working macromolecules. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_5945.map.gz emd_5945.map.gz | 2.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-5945-v30.xml emd-5945-v30.xml emd-5945.xml emd-5945.xml | 9.9 KB 9.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  400_5945.gif 400_5945.gif 80_5945.gif 80_5945.gif | 49.6 KB 3.9 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-5945 http://ftp.pdbj.org/pub/emdb/structures/EMD-5945 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-5945 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-5945 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_5945_validation.pdf.gz emd_5945_validation.pdf.gz | 78.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_5945_full_validation.pdf.gz emd_5945_full_validation.pdf.gz | 77.7 KB | 表示 | |
| XML形式データ |  emd_5945_validation.xml.gz emd_5945_validation.xml.gz | 494 B | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5945 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5945 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5945 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5945 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_5945.map.gz / 形式: CCP4 / 大きさ: 25.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_5945.map.gz / 形式: CCP4 / 大きさ: 25.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | CryoEM of tetrameric Pyruvate Carboxylase in asymmetric architecture. Calculated after sorting of a catalytically active population. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.75 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Pyruvate Carboxylase
| 全体 | 名称: Pyruvate Carboxylase |
|---|---|
| 要素 |
|
-超分子 #1000: Pyruvate Carboxylase
| 超分子 | 名称: Pyruvate Carboxylase / タイプ: sample / ID: 1000 / 集合状態: Homotetramer / Number unique components: 1 |
|---|---|
| 分子量 | 理論値: 520 KDa |
-分子 #1: pyruvate carboxylate
| 分子 | 名称: pyruvate carboxylate / タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 集合状態: tetramer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 520 KDa |
| 組換発現 | 生物種:  |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.05 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 詳細: 16.6 mM Tris-HCl, 1.7 mM acetyl-CoA, 15.7 mM pyruvate, 4.1 mM MgCl2, 1.7 mM DTT, 166.6 mM NaCl, 8.3 mM KHCO3, 1.66 mM ATP. |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / 装置: FEI VITROBOT MARK II |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | JEOL 2200FSC |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: Omega in-column |
| 日付 | 2011年1月1日 |
| 撮影 | カテゴリ: FILM / フィルム・検出器のモデル: KODAK SO-163 FILM / デジタル化 - スキャナー: ZEISS SCAI / デジタル化 - サンプリング間隔: 7 µm / 実像数: 50 / 平均電子線量: 15 e/Å2 / ビット/ピクセル: 16 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.0 mm / 倍率(公称値): 40000 |
| 試料ステージ | 試料ホルダーモデル: GATAN LIQUID NITROGEN |
- 画像解析
画像解析
| CTF補正 | 詳細: Volume correction in defocus groups |
|---|---|
| 最終 再構成 | アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 11.5 Å / 解像度の算出法: OTHER / ソフトウェア - 名称: Relion, Spider / 使用した粒子像数: 14032 |
-原子モデル構築 1
| 初期モデル | PDB ID: |
|---|---|
| ソフトウェア | 名称: MDFF |
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT |
 ムービー
ムービー コントローラー
コントローラー












 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)






















