+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-5919 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Negative stain reconstruction of PGT151 Fab in complex with JR-FL Env with transmembrane region | |||||||||
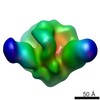
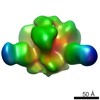
 マップデータ マップデータ | PGT151 Fab in complex with JR-FL Env dCT trimer | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | HIV-1 envelope glycoprotein trimer / broadly neutralizing antibody / PGT151 | |||||||||
| 生物種 |   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) / Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) /  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / ネガティブ染色法 / 解像度: 19.0 Å | |||||||||
 データ登録者 データ登録者 | Lee JH / Blattner C / Wilson IA / Ward AB | |||||||||
 引用 引用 |  ジャーナル: Immunity / 年: 2014 ジャーナル: Immunity / 年: 2014タイトル: Structural delineation of a quaternary, cleavage-dependent epitope at the gp41-gp120 interface on intact HIV-1 Env trimers. 著者: Claudia Blattner / Jeong Hyun Lee / Kwinten Sliepen / Ronald Derking / Emilia Falkowska / Alba Torrents de la Peña / Albert Cupo / Jean-Philippe Julien / Marit van Gils / Peter S Lee / ...著者: Claudia Blattner / Jeong Hyun Lee / Kwinten Sliepen / Ronald Derking / Emilia Falkowska / Alba Torrents de la Peña / Albert Cupo / Jean-Philippe Julien / Marit van Gils / Peter S Lee / Wenjie Peng / James C Paulson / Pascal Poignard / Dennis R Burton / John P Moore / Rogier W Sanders / Ian A Wilson / Andrew B Ward /   要旨: All previously characterized broadly neutralizing antibodies to the HIV-1 envelope glycoprotein (Env) target one of four major sites of vulnerability. Here, we define and structurally characterize a ...All previously characterized broadly neutralizing antibodies to the HIV-1 envelope glycoprotein (Env) target one of four major sites of vulnerability. Here, we define and structurally characterize a unique epitope on Env that is recognized by a recently discovered family of human monoclonal antibodies (PGT151-PGT158). The PGT151 epitope is comprised of residues and glycans at the interface of gp41 and gp120 within a single protomer and glycans from both subunits of a second protomer and represents a neutralizing epitope that is dependent on both gp120 and gp41. Because PGT151 binds only to properly formed, cleaved trimers, this distinctive property, and its ability to stabilize Env trimers, has enabled the successful purification of mature, cleaved Env trimers from the cell surface as a complex with PGT151. Here we compare the structural and functional properties of membrane-extracted Env trimers from several clades with those of the soluble, cleaved SOSIP gp140 trimer. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_5919.map.gz emd_5919.map.gz | 10.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-5919-v30.xml emd-5919-v30.xml emd-5919.xml emd-5919.xml | 14.1 KB 14.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  400_5919.gif 400_5919.gif 80_5919.gif 80_5919.gif | 28.4 KB 3 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-5919 http://ftp.pdbj.org/pub/emdb/structures/EMD-5919 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-5919 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-5919 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_5919_validation.pdf.gz emd_5919_validation.pdf.gz | 78.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_5919_full_validation.pdf.gz emd_5919_full_validation.pdf.gz | 77.4 KB | 表示 | |
| XML形式データ |  emd_5919_validation.xml.gz emd_5919_validation.xml.gz | 493 B | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5919 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5919 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5919 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5919 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_5919.map.gz / 形式: CCP4 / 大きさ: 15.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_5919.map.gz / 形式: CCP4 / 大きさ: 15.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | PGT151 Fab in complex with JR-FL Env dCT trimer | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.18 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Fab fragment of HIV-1 Env antibody PGT151 in complex with cleaved...
| 全体 | 名称: Fab fragment of HIV-1 Env antibody PGT151 in complex with cleaved JR-FL trimer containing the transmembrane region |
|---|---|
| 要素 |
|
-超分子 #1000: Fab fragment of HIV-1 Env antibody PGT151 in complex with cleaved...
| 超分子 | 名称: Fab fragment of HIV-1 Env antibody PGT151 in complex with cleaved JR-FL trimer containing the transmembrane region タイプ: sample / ID: 1000 / 集合状態: 2 Fabs bind one Env Trimer / Number unique components: 2 |
|---|---|
| 分子量 | 理論値: 535 KDa |
-分子 #1: HIV-1 Envelope glycoprotein
| 分子 | 名称: HIV-1 Envelope glycoprotein / タイプ: protein_or_peptide / ID: 1 / Name.synonym: Env 詳細: The cleaved JR-FL trimer was selectively isolated using PGT151. The trimer contains E168K mutation for PG9 binding, and the transmembrane domain. It does not include the cytoplasmic domain. コピー数: 1 / 集合状態: Trimer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス)株: JR-FL / 別称: HIV-1 |
| 分子量 | 理論値: 435 MDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) / 組換細胞: HEK293F / 組換プラスミド: phCMVIII Homo sapiens (ヒト) / 組換細胞: HEK293F / 組換プラスミド: phCMVIII |
-分子 #2: PGT151 Antibody Fab fragment
| 分子 | 名称: PGT151 Antibody Fab fragment / タイプ: protein_or_peptide / ID: 2 / Name.synonym: PGT151 Fab / 詳細: Heavy chain-light chain dimer Fab fragment / コピー数: 2 / 集合状態: dimer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) / 別称: Human / 細胞: B-cell Homo sapiens (ヒト) / 別称: Human / 細胞: B-cell |
| 分子量 | 理論値: 49.5 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) / 組換細胞: HEK293F Homo sapiens (ヒト) / 組換細胞: HEK293F |
-実験情報
-構造解析
| 手法 | ネガティブ染色法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.01 mg/mL |
|---|---|
| 緩衝液 | pH: 7.2 / 詳細: 50 mM Tris, 150 mM NaCl, 0.1% DDM |
| 染色 | タイプ: NEGATIVE / 詳細: Sample diluted to <0.001% DDM, immediately applied to grid briefly, then stained for 30-45 seconds with 2% uranyl formate. |
| グリッド | 詳細: plasma-cleaned carbon coated 400 mesh grid |
| 凍結 | 凍結剤: NONE / 装置: OTHER |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI F20 |
|---|---|
| 温度 | 最低: 293 K / 最高: 293 K |
| アライメント法 | Legacy - 非点収差: Objective astigmatism corrected at 100,000 times magnification |
| 詳細 | Data collected in 5 degree increments |
| 日付 | 2012年9月8日 |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: GATAN ULTRASCAN 4000 (4k x 4k) 実像数: 460 / 平均電子線量: 25 e/Å2 / ビット/ピクセル: 16 |
| Tilt angle min | 0 |
| 電子線 | 加速電圧: 120 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 0.9 µm / 最小 デフォーカス(公称値): 0.6 µm / 倍率(公称値): 100000 |
| 試料ステージ | 試料ホルダーモデル: SIDE ENTRY, EUCENTRIC / Tilt angle max: 55 |
| 実験機器 |  モデル: Tecnai F20 / 画像提供: FEI Company |
- 画像解析
画像解析
| CTF補正 | 詳細: not corrected |
|---|---|
| 最終 再構成 | アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 19.0 Å / 解像度の算出法: OTHER / ソフトウェア - 名称: XMIPP, EMAN, Sparx 詳細: Reference free 2D class averages generated using XMIPP and Sparx to sort particles. RCT model used as initial model and projection matching carried out in EMAN. 使用した粒子像数: 10568 |
-原子モデル構築 1
| 初期モデル | PDB ID: Chain - #0 - Chain ID: A / Chain - #1 - Chain ID: B / Chain - #2 - Chain ID: E / Chain - #3 - Chain ID: F / Chain - #4 - Chain ID: I / Chain - #5 - Chain ID: J |
|---|---|
| ソフトウェア | 名称:  Chimera Chimera |
| 詳細 | The PGV04 Fabs in the structure were removed prior to fitting. |
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
-原子モデル構築 2
| 初期モデル | PDB ID: Chain - #0 - Chain ID: H / Chain - #1 - Chain ID: L |
|---|---|
| ソフトウェア | 名称:  Chimera Chimera |
| 詳細 | Two Fabs were fit independently of each other into the EM reconstruction. |
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
 ムービー
ムービー コントローラー
コントローラー












 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)






















