+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-5770 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Electron cryo-microscopy of a chemical cross-linked K87C/S199C Mm-cpn with ABDM (illuminated, ATP-AlFx) | |||||||||
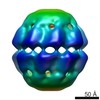
 マップデータ マップデータ | chemically cross-linked K87C/S199C Mm-cpn with ABDM (Illuminated, ATP-AlFx) | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | protein engineering / light-gated nanocage / ABDM cross-linked Mm-cpn | |||||||||
| 生物種 |  Methanococcus maripaludis (古細菌) Methanococcus maripaludis (古細菌) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 16.6 Å | |||||||||
 データ登録者 データ登録者 | Hoersch D / Roh SH / Chiu W / Kortemme T | |||||||||
 引用 引用 |  ジャーナル: Nat Nanotechnol / 年: 2013 ジャーナル: Nat Nanotechnol / 年: 2013タイトル: Reprogramming an ATP-driven protein machine into a light-gated nanocage. 著者: Daniel Hoersch / Soung-Hun Roh / Wah Chiu / Tanja Kortemme /  要旨: Natural protein assemblies have many sophisticated architectures and functions, creating nanoscale storage containers, motors and pumps. Inspired by these systems, protein monomers have been ...Natural protein assemblies have many sophisticated architectures and functions, creating nanoscale storage containers, motors and pumps. Inspired by these systems, protein monomers have been engineered to self-assemble into supramolecular architectures including symmetrical, metal-templated and cage-like structures. The complexity of protein machines, however, has made it difficult to create assemblies with both defined structures and controllable functions. Here we report protein assemblies that have been engineered to function as light-controlled nanocontainers. We show that an adenosine-5'-triphosphate-driven group II chaperonin, which resembles a barrel with a built-in lid, can be reprogrammed to open and close on illumination with different wavelengths of light. By engineering photoswitchable azobenzene-based molecules into the structure, light-triggered changes in interatomic distances in the azobenzene moiety are able to drive large-scale conformational changes of the protein assembly. The different states of the assembly can be visualized with single-particle cryo-electron microscopy, and the nanocages can be used to capture and release non-native cargos. Similar strategies that switch atomic distances with light could be used to build other controllable nanoscale machines. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_5770.map.gz emd_5770.map.gz | 26.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-5770-v30.xml emd-5770-v30.xml emd-5770.xml emd-5770.xml | 9.1 KB 9.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_5770_1.jpg emd_5770_1.jpg | 57.2 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-5770 http://ftp.pdbj.org/pub/emdb/structures/EMD-5770 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-5770 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-5770 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_5770_validation.pdf.gz emd_5770_validation.pdf.gz | 78.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_5770_full_validation.pdf.gz emd_5770_full_validation.pdf.gz | 77.5 KB | 表示 | |
| XML形式データ |  emd_5770_validation.xml.gz emd_5770_validation.xml.gz | 494 B | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5770 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5770 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5770 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5770 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_5770.map.gz / 形式: CCP4 / 大きさ: 28.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_5770.map.gz / 形式: CCP4 / 大きさ: 28.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | chemically cross-linked K87C/S199C Mm-cpn with ABDM (Illuminated, ATP-AlFx) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.17 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : chemically cross-linked K87C/S199C Mm-cpn with ABDM (illuminated,...
| 全体 | 名称: chemically cross-linked K87C/S199C Mm-cpn with ABDM (illuminated, ATP-AlFx) |
|---|---|
| 要素 |
|
-超分子 #1000: chemically cross-linked K87C/S199C Mm-cpn with ABDM (illuminated,...
| 超分子 | 名称: chemically cross-linked K87C/S199C Mm-cpn with ABDM (illuminated, ATP-AlFx) タイプ: sample / ID: 1000 / 集合状態: homohexadecamer / Number unique components: 1 |
|---|---|
| 分子量 | 実験値: 1 MDa / 理論値: 1 MDa / 手法: size exclusion column |
-分子 #1: mutant Methanococcus maripaludis chaperonin
| 分子 | 名称: mutant Methanococcus maripaludis chaperonin / タイプ: protein_or_peptide / ID: 1 / Name.synonym: mutant Mm-cpn / 詳細: ATP-AlFx / 集合状態: 16-mer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  Methanococcus maripaludis (古細菌) Methanococcus maripaludis (古細菌) |
| 分子量 | 実験値: 1 MDa |
| 組換発現 | 生物種:  |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.25 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 詳細: 50 mM KCl, 5 mM MgCl2, 20 mM HEPES, 10% glycerol, 0.1% OG, 1% PEG8000 |
| グリッド | 詳細: 200-mesh R1.2/1.3 holey-carbon grids, glow discharged |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / 装置: FEI VITROBOT MARK III |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | JEOL 2010F |
|---|---|
| 温度 | 最低: 100 K |
| 日付 | 2012年11月30日 |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: GATAN ULTRASCAN 4000 (4k x 4k) 実像数: 54 / 平均電子線量: 20 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 55000 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 3.5 µm / 最小 デフォーカス(公称値): 1.5 µm / 倍率(公称値): 50000 |
| 試料ステージ | 試料ホルダー: Gatan 626 single tilt cryo transfer holder (70) 試料ホルダーモデル: GATAN LIQUID NITROGEN |
- 画像解析
画像解析
| 詳細 | EMAN2 standard package |
|---|---|
| CTF補正 | 詳細: particle based |
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 16.6 Å / 解像度の算出法: OTHER / ソフトウェア - 名称: EMAN2 / 使用した粒子像数: 3718 |
 ムービー
ムービー コントローラー
コントローラー













 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)





















