+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-5588 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Electron cryo-microscopy of the yeast Mediator Cdk8 kinase module | |||||||||
 マップデータ マップデータ | Map of the CDK8 kinase module including Med12, Med13, Cdk8, and CycC | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Mediator / kinase module / CDK8 / Med12 / Med13 / CycC | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 15.0 Å | |||||||||
 データ登録者 データ登録者 | Tsai KL / Sato S / Tomomori-Sato C / Conaway RC / Conaway JW / Asturias FJ | |||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2013 ジャーナル: Nat Struct Mol Biol / 年: 2013タイトル: A conserved Mediator-CDK8 kinase module association regulates Mediator-RNA polymerase II interaction. 著者: Kuang-Lei Tsai / Shigeo Sato / Chieri Tomomori-Sato / Ronald C Conaway / Joan W Conaway / Francisco J Asturias /  要旨: The CDK8 kinase module (CKM) is a conserved, dissociable Mediator subcomplex whose component subunits were genetically linked to the RNA polymerase II (RNAPII) C-terminal domain (CTD) and ...The CDK8 kinase module (CKM) is a conserved, dissociable Mediator subcomplex whose component subunits were genetically linked to the RNA polymerase II (RNAPII) C-terminal domain (CTD) and individually recognized as transcriptional repressors before Mediator was identified as a pre-eminent complex in eukaryotic transcription regulation. We used macromolecular EM and biochemistry to investigate the subunit organization, structure and Mediator interaction of the Saccharomyces cerevisiae CKM. We found that interaction of the CKM with Mediator's middle module interferes with CTD-dependent RNAPII binding to a previously unknown middle-module CTD-binding site and with the holoenzyme formation process. Taken together, our results reveal the basis for CKM repression, clarify the origin of the connection between CKM subunits and the CTD and suggest that a combination of competitive interactions and conformational changes that facilitate holoenzyme formation underlie the mechanism of transcription regulation by Mediator. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_5588.map.gz emd_5588.map.gz | 10.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-5588-v30.xml emd-5588-v30.xml emd-5588.xml emd-5588.xml | 12.2 KB 12.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_5588.jpg emd_5588.jpg | 71.5 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-5588 http://ftp.pdbj.org/pub/emdb/structures/EMD-5588 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-5588 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-5588 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_5588_validation.pdf.gz emd_5588_validation.pdf.gz | 78.2 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_5588_full_validation.pdf.gz emd_5588_full_validation.pdf.gz | 77.3 KB | 表示 | |
| XML形式データ |  emd_5588_validation.xml.gz emd_5588_validation.xml.gz | 494 B | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5588 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5588 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5588 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5588 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_5588.map.gz / 形式: CCP4 / 大きさ: 11.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_5588.map.gz / 形式: CCP4 / 大きさ: 11.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Map of the CDK8 kinase module including Med12, Med13, Cdk8, and CycC | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.1 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
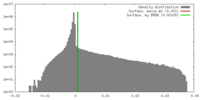
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Cdk8 kinase module of yeast Mediator complex
| 全体 | 名称: Cdk8 kinase module of yeast Mediator complex |
|---|---|
| 要素 |
|
-超分子 #1000: Cdk8 kinase module of yeast Mediator complex
| 超分子 | 名称: Cdk8 kinase module of yeast Mediator complex / タイプ: sample / ID: 1000 / Number unique components: 4 |
|---|---|
| 分子量 | 実験値: 430 KDa / 理論値: 430 KDa |
-分子 #1: Med12
| 分子 | 名称: Med12 / タイプ: protein_or_peptide / ID: 1 / Name.synonym: Srb8 / コピー数: 1 / 組換発現: No / データベース: NCBI |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 実験値: 167 KDa / 理論値: 167 KDa |
-分子 #2: Med13
| 分子 | 名称: Med13 / タイプ: protein_or_peptide / ID: 2 / Name.synonym: Srb9 / コピー数: 1 / 組換発現: No / データベース: NCBI |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 実験値: 160 KDa / 理論値: 160 KDa |
-分子 #3: Cdk8
| 分子 | 名称: Cdk8 / タイプ: protein_or_peptide / ID: 3 / Name.synonym: Srb10 / コピー数: 1 / 組換発現: No / データベース: NCBI |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 実験値: 63 KDa / 理論値: 63 KDa |
-分子 #4: CycC
| 分子 | 名称: CycC / タイプ: protein_or_peptide / ID: 4 / Name.synonym: Srb11 / コピー数: 1 / 組換発現: No / データベース: NCBI |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 実験値: 38 KDa / 理論値: 38 KDa |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.020 mg/mL |
|---|---|
| 緩衝液 | pH: 7.6 詳細: 20mM HEPES, 200mM potassium acetate, 5mM beta-mercaptoethanol |
| グリッド | 詳細: 300 mesh Cu/Rh grids with continuous carbon, glow discharged in amylamine atmosphere |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / チャンバー内温度: 77 K / 装置: HOMEMADE PLUNGER |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI F20 |
|---|---|
| 温度 | 最低: 80 K / 最高: 110 K / 平均: 105 K |
| アライメント法 | Legacy - 非点収差: Objective lens astigmatism was corrected at 125,000 times magnification using a CCD camera |
| 日付 | 2011年12月1日 |
| 撮影 | カテゴリ: FILM / フィルム・検出器のモデル: KODAK SO-163 FILM デジタル化 - スキャナー: NIKON SUPER COOLSCAN 9000 デジタル化 - サンプリング間隔: 6.35 µm / 実像数: 154 / 平均電子線量: 15 e/Å2 / ビット/ピクセル: 16 |
| 電子線 | 加速電圧: 120 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 60400 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 1.2 mm / 最大 デフォーカス(公称値): 4.0 µm / 最小 デフォーカス(公称値): 1.5 µm / 倍率(公称値): 60000 |
| 試料ステージ | 試料ホルダーモデル: GATAN LIQUID NITROGEN |
| 実験機器 |  モデル: Tecnai F20 / 画像提供: FEI Company |
- 画像解析
画像解析
| CTF補正 | 詳細: Each particle |
|---|---|
| 最終 再構成 | アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 15.0 Å / 解像度の算出法: FSC 0.5 CUT-OFF / ソフトウェア - 名称: SPARX, SPIDER / 使用した粒子像数: 70000 |
 ムービー
ムービー コントローラー
コントローラー









 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)