+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-5278 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
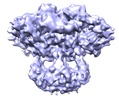
| タイトル | 3D structure of a full-length type 1 inositol 1,4,5-trisphosphate receptor in the closed state | |||||||||
 マップデータ マップデータ | This is a Cryo-EM density map of IP3R1 in the closed state. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | type 1 inositol 1 / 4 / 5-trisphosphate receptor / calcium channel / closed state / single particle cryo-EM | |||||||||
| 機能・相同性 | RIH domain / endoplasmic reticulum membrane 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / ネガティブ染色法 / 解像度: 9.5 Å | |||||||||
 データ登録者 データ登録者 | Ludtke SJ / Tran TP / Ngo QT / Moiseenkova-Bell VY / Chiu W / Serysheva II | |||||||||
 引用 引用 |  ジャーナル: Structure / 年: 2011 ジャーナル: Structure / 年: 2011タイトル: Flexible architecture of IP3R1 by Cryo-EM. 著者: Steven J Ludtke / Thao P Tran / Que T Ngo / Vera Yu Moiseenkova-Bell / Wah Chiu / Irina I Serysheva /  要旨: Inositol 1,4,5-trisphosphate receptors (IP3Rs) play a fundamental role in generating Ca2+ signals that trigger many cellular processes in virtually all eukaryotic cells. Thus far, the three- ...Inositol 1,4,5-trisphosphate receptors (IP3Rs) play a fundamental role in generating Ca2+ signals that trigger many cellular processes in virtually all eukaryotic cells. Thus far, the three-dimensional (3D) structure of these channels has remained extremely controversial. Here, we report a subnanometer resolution electron cryomicroscopy (cryo-EM) structure of a fully functional type 1 IP3R from cerebellum in the closed state. The transmembrane region reveals a twisted bundle of four α helices, one from each subunit, that form a funnel shaped structure around the 4-fold symmetry axis, strikingly similar to the ion-conduction pore of K+ channels. The lumenal face of IP3R1 has prominent densities that surround the pore entrance and similar to the highly structured turrets of Kir channels. 3D statistical analysis of the cryo-EM density map identifies high variance in the cytoplasmic region. This structural variation could be attributed to genuine structural flexibility of IP3R1. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_5278.map.gz emd_5278.map.gz | 56.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-5278-v30.xml emd-5278-v30.xml emd-5278.xml emd-5278.xml | 10.7 KB 10.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_5278.png emd_5278.png | 1.2 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-5278 http://ftp.pdbj.org/pub/emdb/structures/EMD-5278 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-5278 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-5278 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_5278_validation.pdf.gz emd_5278_validation.pdf.gz | 79.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_5278_full_validation.pdf.gz emd_5278_full_validation.pdf.gz | 78.4 KB | 表示 | |
| XML形式データ |  emd_5278_validation.xml.gz emd_5278_validation.xml.gz | 494 B | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5278 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5278 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5278 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5278 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_5278.map.gz / 形式: CCP4 / 大きさ: 62.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_5278.map.gz / 形式: CCP4 / 大きさ: 62.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | This is a Cryo-EM density map of IP3R1 in the closed state. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.81 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Type 1 Inositol 1,4,5-Trisphosphate Receptor
| 全体 | 名称: Type 1 Inositol 1,4,5-Trisphosphate Receptor |
|---|---|
| 要素 |
|
-超分子 #1000: Type 1 Inositol 1,4,5-Trisphosphate Receptor
| 超分子 | 名称: Type 1 Inositol 1,4,5-Trisphosphate Receptor / タイプ: sample / ID: 1000 詳細: The sample was frozen immediately upon channel protein purification 集合状態: Tetramer / Number unique components: 1 |
|---|---|
| 分子量 | 理論値: 1.3 MDa |
-分子 #1: Ion channel
| 分子 | 名称: Ion channel / タイプ: protein_or_peptide / ID: 1 / Name.synonym: IP3R1 詳細: IP3R1 channel was solubilized with CHAPS from rat cerebellum and purified by immunoaffinity chromatography. コピー数: 4 / 集合状態: Tetramer / 組換発現: No / データベース: NCBI |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 1.3 MDa |
| 配列 | GO: endoplasmic reticulum membrane / InterPro: RIH domain |
-実験情報
-構造解析
| 手法 | ネガティブ染色法, クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.5 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 詳細: 50 mM Tris-HCl, 150 mM sodium chloride, 1 mM DTT, 1 mM EDTA, 0.4% CHAPS, 5% sucrose, protease inhibitors |
| 染色 | タイプ: NEGATIVE / 詳細: Frozen-hydrated |
| グリッド | 詳細: 400 mesh Quantifoil holey grids covered with a continuous carbon film |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 101 K / 装置: OTHER / 詳細: Vitrification instrument: Vitrobot (FEI) / 手法: Blot for 2-3 sec before plunging |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | JEOL 2010F |
|---|---|
| 温度 | 最低: 103 K / 最高: 104 K / 平均: 103 K |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: GENERIC GATAN (4k x 4k) デジタル化 - サンプリング間隔: 1.81 µm / 実像数: 869 / 平均電子線量: 18 e/Å2 / ビット/ピクセル: 16 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.0 mm / 最大 デフォーカス(公称値): 3.5 µm / 最小 デフォーカス(公称値): 1.4 µm / 倍率(公称値): 60000 |
| 試料ステージ | 試料ホルダー: Side entry liquid nitrogen-cooled / 試料ホルダーモデル: GATAN LIQUID NITROGEN |
- 画像解析
画像解析
| 詳細 | 869 CCD frames |
|---|---|
| CTF補正 | 詳細: Per particle phase-flipping with amplitude correction of class-averages |
| 最終 再構成 | アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 9.5 Å / 解像度の算出法: FSC 0.5 CUT-OFF / ソフトウェア - 名称: EMAN1.9 詳細: Direct Fourier Inversion based on Wiener-filtered and CTF amplitude-corrected class-averages 使用した粒子像数: 37231 |
 ムービー
ムービー コントローラー
コントローラー






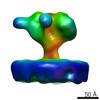
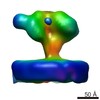

 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)





















