+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Bacterial LAT transporter BASC in complex with L-Ala and NB53 | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Transporter / nanobody / aminoacid transporter / MEMBRANE PROTEIN | |||||||||
| 機能・相同性 | : / L-amino acid transmembrane transporter activity / Amino acid/polyamine transporter I / Amino acid permease / membrane / Putative amino acid/polyamine transport protein 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Carnobacterium sp. (バクテリア) / Carnobacterium sp. (バクテリア) /  Carnobacterium sp. AT7 (バクテリア) / Carnobacterium sp. AT7 (バクテリア) /  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.9 Å | |||||||||
 データ登録者 データ登録者 | Fort J / Palacin M | |||||||||
| 資金援助 |  スペイン, 2件 スペイン, 2件
| |||||||||
 引用 引用 |  ジャーナル: PNAS Nexus / 年: 2025 ジャーナル: PNAS Nexus / 年: 2025タイトル: The conserved lysine residue in transmembrane helix 5 is pivotal for the cytoplasmic gating of the L-amino acid transporters. 著者: Joana Fort / Adrià Nicolàs-Aragó / Luca Maggi / Maria Martinez-Molledo / Despoina Kapiki / Paula González-Novoa / Patricia Gómez-Gejo / Niels Zijlstra / Susanna Bodoy / Els Pardon / Jan ...著者: Joana Fort / Adrià Nicolàs-Aragó / Luca Maggi / Maria Martinez-Molledo / Despoina Kapiki / Paula González-Novoa / Patricia Gómez-Gejo / Niels Zijlstra / Susanna Bodoy / Els Pardon / Jan Steyaert / Oscar Llorca / Modesto Orozco / Thorben Cordes / Manuel Palacín /    要旨: L-Amino acid transporters (LATs) play a key role in a wide range of physiological processes. Defects in LATs can lead to neurological disorders and aminoacidurias, while the overexpression of these ...L-Amino acid transporters (LATs) play a key role in a wide range of physiological processes. Defects in LATs can lead to neurological disorders and aminoacidurias, while the overexpression of these transporters is related to cancer. BasC is a bacterial LAT transporter with an APC fold. In this study, to monitor the cytoplasmic motion of BasC, we developed a single-molecule Förster resonance energy transfer assay that can characterize the conformational states of the intracellular gate in solution at room temperature. Based on combined biochemical and biophysical data and molecular dynamics simulations, we propose a model in which the conserved lysine residue in TM5 supports TM1a to explore both open and closed states within the cytoplasmic gate under apo conditions. This equilibrium can be altered by substrates, mutation of conserved lysine 154 in TM5, or a transport-blocking nanobody interacting with TM1a. Overall, these findings provide insights into the transport mechanism of BasC and highlight the significance of the lysine residue in TM5 in the cytoplasmic gating of LATs. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_51912.map.gz emd_51912.map.gz | 253.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-51912-v30.xml emd-51912-v30.xml emd-51912.xml emd-51912.xml | 21 KB 21 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_51912.png emd_51912.png | 64.1 KB | ||
| Filedesc metadata |  emd-51912.cif.gz emd-51912.cif.gz | 6.8 KB | ||
| その他 |  emd_51912_half_map_1.map.gz emd_51912_half_map_1.map.gz emd_51912_half_map_2.map.gz emd_51912_half_map_2.map.gz | 475.7 MB 475.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-51912 http://ftp.pdbj.org/pub/emdb/structures/EMD-51912 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-51912 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-51912 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_51912_validation.pdf.gz emd_51912_validation.pdf.gz | 1.1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_51912_full_validation.pdf.gz emd_51912_full_validation.pdf.gz | 1.1 MB | 表示 | |
| XML形式データ |  emd_51912_validation.xml.gz emd_51912_validation.xml.gz | 19 KB | 表示 | |
| CIF形式データ |  emd_51912_validation.cif.gz emd_51912_validation.cif.gz | 22.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-51912 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-51912 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-51912 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-51912 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  9h76MC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_51912.map.gz / 形式: CCP4 / 大きさ: 512 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_51912.map.gz / 形式: CCP4 / 大きさ: 512 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.51 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_51912_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_51912_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : BASC-NB53 complex
| 全体 | 名称: BASC-NB53 complex |
|---|---|
| 要素 |
|
-超分子 #1: BASC-NB53 complex
| 超分子 | 名称: BASC-NB53 complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:  Carnobacterium sp. (バクテリア) Carnobacterium sp. (バクテリア) |
| 分子量 | 理論値: 45 kDa/nm |
-分子 #1: Putative amino acid/polyamine transport protein
| 分子 | 名称: Putative amino acid/polyamine transport protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Carnobacterium sp. AT7 (バクテリア) Carnobacterium sp. AT7 (バクテリア) |
| 分子量 | 理論値: 46.696609 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MKEVSGITAL TVVVGTVIGA GIFFKPTAVY GAAGAPGLGL LAWFVAGIIT IAGGLTVAEI GTIYPQTGGM MIYLEKVYGR WLGFLVGWA QMVIYYPANI AALAIIFATQ FVNLFALSDS TIVPTAILTS IFLMGVNFLG TKYSGWIQTL ATILKLIPLV V IIVAGLLY ...文字列: MKEVSGITAL TVVVGTVIGA GIFFKPTAVY GAAGAPGLGL LAWFVAGIIT IAGGLTVAEI GTIYPQTGGM MIYLEKVYGR WLGFLVGWA QMVIYYPANI AALAIIFATQ FVNLFALSDS TIVPTAILTS IFLMGVNFLG TKYSGWIQTL ATILKLIPLV V IIVAGLLY PGGGVIRLVP FSVETHPVLT SFGSALIATL FAYDGWINVG TLAGEMKNPG KMLPKVIIGG LSIVMAVYLL TN IAYLFVL DSSQLAGTDT PAALVASHLF EGIGSKLVTI GILISVFGGI NGYIISGLRV PYALATQKML PFSDWFARIN PKT NLPING GLVMLGIAIV MILTGQFNQL TDLIVFVIWF FITLTFIAVI ILRKTQPDIE RPYRVPFYPV IPLIAIIGGL YIIF NTLIV QPKNAFIGIL LTLIGIPIYF YCKKKYGSPE UniProtKB: Putative amino acid/polyamine transport protein |
-分子 #2: Nanobody NB53
| 分子 | 名称: Nanobody NB53 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 14.257789 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MAQVQLVESG GGLVQAGGSL RLSCAASGIA FSRMSMAWYR QDPGKQRALV ARITNDGSTY YDDSVKGRFT ISRDNAKNTV HLQMNSLKP EDTAVYYCNA QLVAWSENYW GQGTQVTVSS HHHHHHEPEA |
-分子 #3: ALANINE
| 分子 | 名称: ALANINE / タイプ: ligand / ID: 3 / コピー数: 1 / 式: ALA |
|---|---|
| 分子量 | 理論値: 89.093 Da |
| Chemical component information | 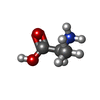 ChemComp-ALA: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2.9 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 |
| グリッド | モデル: C-flat-1.2/1.3 / 材質: GRAPHENE OXIDE / メッシュ: 300 |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 撮影したグリッド数: 1 / 実像数: 38963 / 平均露光時間: 0.022 sec. / 平均電子線量: 1.04 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.4 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー





 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)





































