+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 9h76 | |||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Title | Bacterial LAT transporter BASC in complex with L-Ala and NB53 | |||||||||||||||||||||||||||||||||
 Components Components |
| |||||||||||||||||||||||||||||||||
 Keywords Keywords | MEMBRANE PROTEIN / Transporter / nanobody / aminoacid transporter | |||||||||||||||||||||||||||||||||
| Function / homology | : / L-amino acid transmembrane transporter activity / Amino acid/polyamine transporter I / Amino acid permease / membrane / ALANINE / Putative amino acid/polyamine transport protein Function and homology information Function and homology information | |||||||||||||||||||||||||||||||||
| Biological species |  Carnobacterium sp. AT7 (bacteria) Carnobacterium sp. AT7 (bacteria) | |||||||||||||||||||||||||||||||||
| Method | ELECTRON MICROSCOPY / single particle reconstruction / cryo EM / Resolution: 2.9 Å | |||||||||||||||||||||||||||||||||
 Authors Authors | Fort, J. / Palacin, M. | |||||||||||||||||||||||||||||||||
| Funding support |  Spain, 2items Spain, 2items
| |||||||||||||||||||||||||||||||||
 Citation Citation |  Journal: PNAS Nexus / Year: 2025 Journal: PNAS Nexus / Year: 2025Title: The conserved lysine residue in transmembrane helix 5 is pivotal for the cytoplasmic gating of the L-amino acid transporters. Authors: Joana Fort / Adrià Nicolàs-Aragó / Luca Maggi / Maria Martinez-Molledo / Despoina Kapiki / Paula González-Novoa / Patricia Gómez-Gejo / Niels Zijlstra / Susanna Bodoy / Els Pardon / Jan ...Authors: Joana Fort / Adrià Nicolàs-Aragó / Luca Maggi / Maria Martinez-Molledo / Despoina Kapiki / Paula González-Novoa / Patricia Gómez-Gejo / Niels Zijlstra / Susanna Bodoy / Els Pardon / Jan Steyaert / Oscar Llorca / Modesto Orozco / Thorben Cordes / Manuel Palacín /    Abstract: L-Amino acid transporters (LATs) play a key role in a wide range of physiological processes. Defects in LATs can lead to neurological disorders and aminoacidurias, while the overexpression of these ...L-Amino acid transporters (LATs) play a key role in a wide range of physiological processes. Defects in LATs can lead to neurological disorders and aminoacidurias, while the overexpression of these transporters is related to cancer. BasC is a bacterial LAT transporter with an APC fold. In this study, to monitor the cytoplasmic motion of BasC, we developed a single-molecule Förster resonance energy transfer assay that can characterize the conformational states of the intracellular gate in solution at room temperature. Based on combined biochemical and biophysical data and molecular dynamics simulations, we propose a model in which the conserved lysine residue in TM5 supports TM1a to explore both open and closed states within the cytoplasmic gate under apo conditions. This equilibrium can be altered by substrates, mutation of conserved lysine 154 in TM5, or a transport-blocking nanobody interacting with TM1a. Overall, these findings provide insights into the transport mechanism of BasC and highlight the significance of the lysine residue in TM5 in the cytoplasmic gating of LATs. | |||||||||||||||||||||||||||||||||
| History |
|
- Structure visualization
Structure visualization
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  9h76.cif.gz 9h76.cif.gz | 114.7 KB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb9h76.ent.gz pdb9h76.ent.gz | 86.3 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  9h76.json.gz 9h76.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/h7/9h76 https://data.pdbj.org/pub/pdb/validation_reports/h7/9h76 ftp://data.pdbj.org/pub/pdb/validation_reports/h7/9h76 ftp://data.pdbj.org/pub/pdb/validation_reports/h7/9h76 | HTTPS FTP |
|---|
-Related structure data
| Related structure data |  51912MC M: map data used to model this data C: citing same article ( |
|---|---|
| Similar structure data | Similarity search - Function & homology  F&H Search F&H Search |
- Links
Links
- Assembly
Assembly
| Deposited unit | 
|
|---|---|
| 1 |
|
- Components
Components
| #1: Protein | Mass: 46696.609 Da / Num. of mol.: 1 Source method: isolated from a genetically manipulated source Source: (gene. exp.)  Carnobacterium sp. AT7 (bacteria) / Gene: CAT7_03719 / Production host: Carnobacterium sp. AT7 (bacteria) / Gene: CAT7_03719 / Production host:  |
|---|---|
| #2: Antibody | Mass: 14257.789 Da / Num. of mol.: 1 Source method: isolated from a genetically manipulated source Source: (gene. exp.)   |
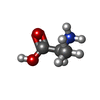
| #3: Chemical | ChemComp-ALA / |
| Has ligand of interest | Y |
| Has protein modification | Y |
-Experimental details
-Experiment
| Experiment | Method: ELECTRON MICROSCOPY |
|---|---|
| EM experiment | Aggregation state: PARTICLE / 3D reconstruction method: single particle reconstruction |
- Sample preparation
Sample preparation
| Component | Name: BASC-NB53 complex / Type: COMPLEX / Entity ID: #1-#2 / Source: RECOMBINANT |
|---|---|
| Molecular weight | Value: 45 kDa/nm / Experimental value: NO |
| Source (natural) | Organism:  Carnobacterium sp. (bacteria) Carnobacterium sp. (bacteria) |
| Source (recombinant) | Organism:  |
| Buffer solution | pH: 7.4 |
| Specimen | Conc.: 2.9 mg/ml / Embedding applied: NO / Shadowing applied: NO / Staining applied: NO / Vitrification applied: YES |
| Specimen support | Grid material: GRAPHENE OXIDE / Grid mesh size: 300 divisions/in. / Grid type: C-flat-1.2/1.3 |
| Vitrification | Cryogen name: ETHANE |
- Electron microscopy imaging
Electron microscopy imaging
| Experimental equipment |  Model: Titan Krios / Image courtesy: FEI Company |
|---|---|
| Microscopy | Model: TFS KRIOS |
| Electron gun | Electron source:  FIELD EMISSION GUN / Accelerating voltage: 300 kV / Illumination mode: FLOOD BEAM FIELD EMISSION GUN / Accelerating voltage: 300 kV / Illumination mode: FLOOD BEAM |
| Electron lens | Mode: BRIGHT FIELD / Nominal defocus max: 2400 nm / Nominal defocus min: 1000 nm |
| Image recording | Average exposure time: 0.022 sec. / Electron dose: 1.04 e/Å2 / Film or detector model: GATAN K3 (6k x 4k) / Num. of grids imaged: 1 / Num. of real images: 38963 |
- Processing
Processing
| EM software |
| ||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Image processing | Details: 32312 images selected after Ctfs and Motion correct. | ||||||||||||||||||||||||||||||||||||||||
| CTF correction | Type: PHASE FLIPPING AND AMPLITUDE CORRECTION | ||||||||||||||||||||||||||||||||||||||||
| Particle selection | Num. of particles selected: 2000000 Details: particle picked using CS template picker with low resolution volume abtained previously | ||||||||||||||||||||||||||||||||||||||||
| 3D reconstruction | Resolution: 2.9 Å / Resolution method: FSC 0.143 CUT-OFF / Num. of particles: 324910 / Symmetry type: POINT | ||||||||||||||||||||||||||||||||||||||||
| Refine LS restraints |
|
 Movie
Movie Controller
Controller






 PDBj
PDBj