+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-5154 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Negative stain EM of the recombinant Acidianus tengchongensis chaperonin beta. | |||||||||
 マップデータ マップデータ | This is the negative stain microscopy reconstruction of the recombinant thermosome beta from Acidianus tengchongensis | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | rATcpn-beta / Group II chaperonin / Thermosome | |||||||||
| 生物種 |  Acidianus tengchongensis (古細菌) Acidianus tengchongensis (古細菌) | |||||||||
| 手法 | 単粒子再構成法 / ネガティブ染色法 / 解像度: 14.0 Å | |||||||||
 データ登録者 データ登録者 | Huo Y / Hu Z / Zhang K / Wang L / Zhai Y / Zhou Q / Lander G / Zhu J / He Y / Pang X ...Huo Y / Hu Z / Zhang K / Wang L / Zhai Y / Zhou Q / Lander G / Zhu J / He Y / Pang X / Xu W / Bartlam M / Dong Z / Sun F | |||||||||
 引用 引用 |  ジャーナル: Structure / 年: 2010 ジャーナル: Structure / 年: 2010タイトル: Crystal structure of group II chaperonin in the open state. 著者: Yanwu Huo / Zhongjun Hu / Kai Zhang / Li Wang / Yujia Zhai / Qiangjun Zhou / Gabe Lander / Jiang Zhu / Yongzhi He / Xiaoyun Pang / Wei Xu / Mark Bartlam / Zhiyang Dong / Fei Sun /  要旨: Thermosomes are group II chaperonins responsible for protein refolding in an ATP-dependent manner. Little is known regarding the conformational changes of thermosomes during their functional cycle ...Thermosomes are group II chaperonins responsible for protein refolding in an ATP-dependent manner. Little is known regarding the conformational changes of thermosomes during their functional cycle due to a lack of high-resolution structure in the open state. Here, we report the first complete crystal structure of thermosome (rATcpnβ) in the open state from Acidianus tengchongensis. There is a ∼30° rotation of the apical and lid domains compared with the previous closed structure. Besides, the structure reveals a conspicuous hydrophobic patch in the lid domain, and residues locating in this patch are conserved across species. Both the closed and open forms of rATcpnβ were also reconstructed by electron microscopy (EM). Structural fitting revealed the detailed conformational change from the open to the closed state. Structural comparison as well as protease K digestion indicated only ATP binding without hydrolysis does not induce chamber closure of thermosome. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_5154.map.gz emd_5154.map.gz | 13.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-5154-v30.xml emd-5154-v30.xml emd-5154.xml emd-5154.xml | 9.9 KB 9.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_5154_1.png emd_5154_1.png | 186.8 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-5154 http://ftp.pdbj.org/pub/emdb/structures/EMD-5154 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-5154 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-5154 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_5154_validation.pdf.gz emd_5154_validation.pdf.gz | 79.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_5154_full_validation.pdf.gz emd_5154_full_validation.pdf.gz | 78.6 KB | 表示 | |
| XML形式データ |  emd_5154_validation.xml.gz emd_5154_validation.xml.gz | 493 B | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5154 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5154 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5154 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5154 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_5154.map.gz / 形式: CCP4 / 大きさ: 15.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_5154.map.gz / 形式: CCP4 / 大きさ: 15.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | This is the negative stain microscopy reconstruction of the recombinant thermosome beta from Acidianus tengchongensis | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Negative stain EM of the recombinant Acidianus tengchongensis cha...
| 全体 | 名称: Negative stain EM of the recombinant Acidianus tengchongensis chaperonin beta |
|---|---|
| 要素 |
|
-超分子 #1000: Negative stain EM of the recombinant Acidianus tengchongensis cha...
| 超分子 | 名称: Negative stain EM of the recombinant Acidianus tengchongensis chaperonin beta タイプ: sample / ID: 1000 / 詳細: The sample was monodisperse / 集合状態: octadecamer / Number unique components: 18 |
|---|---|
| 分子量 | 実験値: 1.08 MDa / 理論値: 1.08 MDa |
-分子 #1: rATcpn-beta
| 分子 | 名称: rATcpn-beta / タイプ: protein_or_peptide / ID: 1 / Name.synonym: chaperonin beta / コピー数: 18 / 集合状態: 18mer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  Acidianus tengchongensis (古細菌) / 株: S5 / 細胞中の位置: cytoplasm Acidianus tengchongensis (古細菌) / 株: S5 / 細胞中の位置: cytoplasm |
| 分子量 | 理論値: 1.08 MDa |
| 組換発現 | 生物種:  |
-実験情報
-構造解析
| 手法 | ネガティブ染色法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| 染色 | タイプ: NEGATIVE 詳細: Grids with adsorbed protein floated on 4% w/v uranyl acetate for 25 seconds. |
| グリッド | 詳細: 200 mesh gold grid |
| 凍結 | 凍結剤: NONE / 装置: OTHER |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI 20 |
|---|---|
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: GATAN ULTRASCAN 1000 (2k x 2k) デジタル化 - サンプリング間隔: 14 µm / 実像数: 100 / ビット/ピクセル: 16 |
| 電子線 | 加速電圧: 200 kV / 電子線源: LAB6 |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2 mm / 最大 デフォーカス(公称値): 0.8 µm / 最小 デフォーカス(公称値): 0.3 µm / 倍率(公称値): 70000 |
| 試料ステージ | 試料ホルダー: Eucentric / 試料ホルダーモデル: SIDE ENTRY, EUCENTRIC |
- 画像解析
画像解析
| 詳細 | The particles were selected by hand. |
|---|---|
| CTF補正 | 詳細: each photo |
| 最終 再構成 | アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 14.0 Å / 解像度の算出法: OTHER / ソフトウェア - 名称: EMAN / 使用した粒子像数: 2328 |
 ムービー
ムービー コントローラー
コントローラー



 UCSF Chimera
UCSF Chimera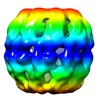









 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)





















