登録情報 データベース : EMDB / ID : EMD-51247タイトル Structure of a mononucleosome bound by one copy of Chd1 with the DBD on the exit-side DNA. Mononucleosome bound by one copy of Chd1 with the DBD on the exit-side DNA 複合体 : Chd1 bound to a hexasome-nucleosome complex with a dyad-to-dyad distance of 103 bpタンパク質・ペプチド : Histone H3.2タンパク質・ペプチド : Histone H4タンパク質・ペプチド : Histone H2A type 1タンパク質・ペプチド : Histone H2B 1.1DNA : DNA (231-MER)DNA : DNA (231-MER)タンパク質・ペプチド : Chromo domain-containing protein 1リガンド : ADENOSINE-5'-DIPHOSPHATEリガンド : BERYLLIUM TRIFLUORIDE IONリガンド : MAGNESIUM ION / / / / 機能・相同性 分子機能 ドメイン・相同性 構成要素
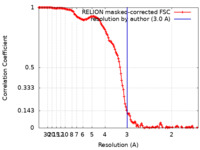
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 Saccharomyces cerevisiae S288C (パン酵母) / Xenopus laevis (アフリカツメガエル) / synthetic construct (人工物) 手法 / / 解像度 : 3.0 Å Engeholm M / Roske JJ / Oberbeckmann E / Dienemann C / Lidschreiber M / Cramer P / Farnung L 資金援助 Organization Grant number 国 German Research Foundation (DFG) European Research Council (ERC) European Union
ジャーナル : Mol Cell / 年 : 2024タイトル : Resolution of transcription-induced hexasome-nucleosome complexes by Chd1 and FACT.著者 : Maik Engeholm / Johann J Roske / Elisa Oberbeckmann / Christian Dienemann / Michael Lidschreiber / Patrick Cramer / Lucas Farnung / 要旨 : To maintain the nucleosome organization of transcribed genes, ATP-dependent chromatin remodelers collaborate with histone chaperones. Here, we show that at the 5' ends of yeast genes, RNA polymerase ... To maintain the nucleosome organization of transcribed genes, ATP-dependent chromatin remodelers collaborate with histone chaperones. Here, we show that at the 5' ends of yeast genes, RNA polymerase II (RNAPII) generates hexasomes that occur directly adjacent to nucleosomes. The resulting hexasome-nucleosome complexes are then resolved by Chd1. We present two cryoelectron microscopy (cryo-EM) structures of Chd1 bound to a hexasome-nucleosome complex before and after restoration of the missing inner H2A/H2B dimer by FACT. Chd1 uniquely interacts with the complex, positioning its ATPase domain to shift the hexasome away from the nucleosome. In the absence of the inner H2A/H2B dimer, its DNA-binding domain (DBD) packs against the ATPase domain, suggesting an inhibited state. Restoration of the dimer by FACT triggers a rearrangement that displaces the DBD and stimulates Chd1 remodeling. Our results demonstrate how chromatin remodelers interact with a complex nucleosome assembly and suggest how Chd1 and FACT jointly support transcription by RNAPII. 履歴 登録 2024年8月4日 - ヘッダ(付随情報) 公開 2024年9月18日 - マップ公開 2024年9月18日 - 更新 2024年10月2日 - 現状 2024年10月2日 処理サイト : PDBe / 状態 : 公開
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報
 マップデータ
マップデータ 試料
試料 キーワード
キーワード 機能・相同性情報
機能・相同性情報
 データ登録者
データ登録者 ドイツ, European Union, 2件
ドイツ, European Union, 2件  引用
引用 ジャーナル: Mol Cell / 年: 2024
ジャーナル: Mol Cell / 年: 2024


 構造の表示
構造の表示 ダウンロードとリンク
ダウンロードとリンク emd_51247.map.gz
emd_51247.map.gz EMDBマップデータ形式
EMDBマップデータ形式 emd-51247-v30.xml
emd-51247-v30.xml emd-51247.xml
emd-51247.xml EMDBヘッダ
EMDBヘッダ emd_51247_fsc.xml
emd_51247_fsc.xml FSCデータファイル
FSCデータファイル emd_51247.png
emd_51247.png emd_51247_msk_1.map
emd_51247_msk_1.map マスクマップ
マスクマップ emd-51247.cif.gz
emd-51247.cif.gz emd_51247_additional_1.map.gz
emd_51247_additional_1.map.gz emd_51247_half_map_1.map.gz
emd_51247_half_map_1.map.gz emd_51247_half_map_2.map.gz
emd_51247_half_map_2.map.gz http://ftp.pdbj.org/pub/emdb/structures/EMD-51247
http://ftp.pdbj.org/pub/emdb/structures/EMD-51247 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-51247
ftp://ftp.pdbj.org/pub/emdb/structures/EMD-51247 emd_51247_validation.pdf.gz
emd_51247_validation.pdf.gz EMDB検証レポート
EMDB検証レポート emd_51247_full_validation.pdf.gz
emd_51247_full_validation.pdf.gz emd_51247_validation.xml.gz
emd_51247_validation.xml.gz emd_51247_validation.cif.gz
emd_51247_validation.cif.gz https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-51247
https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-51247 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-51247
ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-51247 リンク
リンク EMDB (EBI/PDBe) /
EMDB (EBI/PDBe) /  EMDataResource
EMDataResource マップ
マップ ダウンロード / ファイル: emd_51247.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES)
ダウンロード / ファイル: emd_51247.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) emd_51247_msk_1.map
emd_51247_msk_1.map 試料の構成要素
試料の構成要素 解析
解析 試料調製
試料調製 電子顕微鏡法
電子顕微鏡法 FIELD EMISSION GUN
FIELD EMISSION GUN
 ムービー
ムービー コントローラー
コントローラー






















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)













































 Trichoplusia ni (イラクサキンウワバ)
Trichoplusia ni (イラクサキンウワバ)