+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | E.coli gyrase holocomplex with chirally wrapped 217 bp DNA fragment | ||||||||||||
 マップデータ マップデータ | composite map | ||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | DNA gyrase / type II topoisomerase / supercoiling / DNA crossover / ISOMERASE | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報negative regulation of DNA-templated DNA replication / DNA topoisomerase type II (double strand cut, ATP-hydrolyzing) complex / DNA negative supercoiling activity / DNA topoisomerase type II (double strand cut, ATP-hydrolyzing) activity / DNA topoisomerase (ATP-hydrolysing) / DNA topological change / ATP-dependent activity, acting on DNA / DNA-templated DNA replication / chromosome / response to xenobiotic stimulus ...negative regulation of DNA-templated DNA replication / DNA topoisomerase type II (double strand cut, ATP-hydrolyzing) complex / DNA negative supercoiling activity / DNA topoisomerase type II (double strand cut, ATP-hydrolyzing) activity / DNA topoisomerase (ATP-hydrolysing) / DNA topological change / ATP-dependent activity, acting on DNA / DNA-templated DNA replication / chromosome / response to xenobiotic stimulus / response to antibiotic / DNA-templated transcription / DNA binding / ATP binding / metal ion binding / identical protein binding / membrane / cytosol / cytoplasm 類似検索 - 分子機能 | ||||||||||||
| 生物種 |   Escherichia phage Mu (ファージ) Escherichia phage Mu (ファージ) | ||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.32 Å | ||||||||||||
 データ登録者 データ登録者 | Michalczyk E / Ghilarov D | ||||||||||||
| 資金援助 |  英国, 英国,  ポーランド, 3件 ポーランド, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Proceedings of the National Academy of Sciences USA ジャーナル: Proceedings of the National Academy of Sciences USA年: 2024 タイトル: Structure of Escherichia coli DNA gyrase with chirally wrapped DNA supports ratchet-and-pawl mechanism for an ATP-powered supercoiling motor 著者: Michalczyk E / Pakosz-Stepien Z / Liston J / Gittins O / Pabis M / Heddle JG / Ghilarov D | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
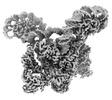
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_51222.map.gz emd_51222.map.gz | 241.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-51222-v30.xml emd-51222-v30.xml emd-51222.xml emd-51222.xml | 21.7 KB 21.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_51222.png emd_51222.png | 101.8 KB | ||
| Filedesc metadata |  emd-51222.cif.gz emd-51222.cif.gz | 7.7 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-51222 http://ftp.pdbj.org/pub/emdb/structures/EMD-51222 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-51222 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-51222 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_51222_validation.pdf.gz emd_51222_validation.pdf.gz | 461.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_51222_full_validation.pdf.gz emd_51222_full_validation.pdf.gz | 461.4 KB | 表示 | |
| XML形式データ |  emd_51222_validation.xml.gz emd_51222_validation.xml.gz | 7.9 KB | 表示 | |
| CIF形式データ |  emd_51222_validation.cif.gz emd_51222_validation.cif.gz | 9.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-51222 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-51222 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-51222 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-51222 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  9gbvMC C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_51222.map.gz / 形式: CCP4 / 大きさ: 512 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_51222.map.gz / 形式: CCP4 / 大きさ: 512 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | composite map | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.86 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Ternary complex of E.coli DNA gyrase with 217 bp linear dsDNA fragment
| 全体 | 名称: Ternary complex of E.coli DNA gyrase with 217 bp linear dsDNA fragment |
|---|---|
| 要素 |
|
-超分子 #1: Ternary complex of E.coli DNA gyrase with 217 bp linear dsDNA fragment
| 超分子 | 名称: Ternary complex of E.coli DNA gyrase with 217 bp linear dsDNA fragment タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#4 |
|---|
-超分子 #2: E. coli DNA gyrase
| 超分子 | 名称: E. coli DNA gyrase / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:  |
-超分子 #3: dsDNA
| 超分子 | 名称: dsDNA / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #3-#4 |
|---|---|
| 由来(天然) | 生物種:  Escherichia phage Mu (ファージ) Escherichia phage Mu (ファージ) |
-分子 #1: DNA gyrase subunit A
| 分子 | 名称: DNA gyrase subunit A / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO / EC番号: DNA topoisomerase (ATP-hydrolysing) |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 97.646195 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: SSDLAREITP VNIEEELKSS YLDYAMSVIV GRALPDVRDG LKPVHRRVLY AMNVLGNDWN KAYKKSARVV GDVIGKYHPH GDSAVYDTI VRMAQPFSLR YMLVDGQGNF GSIDGDSAAA MRYTEIRLAK IAHELMADLE KETVDFVDNY DGTEKIPDVM P TKIPNLLV ...文字列: SSDLAREITP VNIEEELKSS YLDYAMSVIV GRALPDVRDG LKPVHRRVLY AMNVLGNDWN KAYKKSARVV GDVIGKYHPH GDSAVYDTI VRMAQPFSLR YMLVDGQGNF GSIDGDSAAA MRYTEIRLAK IAHELMADLE KETVDFVDNY DGTEKIPDVM P TKIPNLLV NGSSGIAVGM ATNIPPHNLT EVINGCLAYI DDEDISIEGL MEHIPGPDFP TAAIINGRRG IEEAYRTGRG KV YIRARAE VEVDAKTGRE TIIVHEIPYQ VNKARLIEKI AELVKEKRVE GISALRDESD KDGMRIVIEV KRDAVGEVVL NNL YSQTQL QVSFGINMVA LHHGQPKIMN LKDIIAAFVR HRREVVTRRT IFELRKARDR AHILEALAVA LANIDPIIEL IRHA PTPAE AKTALVANPW QLGNVAAMLE RAGDDAARPE WLEPEFGVRD GLYYLTEQQA QAILDLRLQK LTGLEHEKLL DEYKE LLDQ IAELLRILGS ADRLMEVIRE ELELVREQFG DKRRTEITAN SADINLEDLI TQEDVVVTLS HQGYVKYQPL SEYEAQ RRG GKGKSAARIK EEDFIDRLLV ANTHDHILCF SSRGRVYSMK VYQLPEATRG ARGRPIVNLL PLEQDERITA ILPVTEF EE GVKVFMATAN GTVKKTVLTE FNRLRTAGKV AIKLVDGDEL IGVDLTSGED EVMLFSAEGK VVRFKESSVR AMGCNTTG V RGIRLGEGDK VVSLIVPRGD GAILTATQNG YGKRTAVAEY PTKSRATKGV ISIKVTERNG LVVGAVQVDD CDQIMMITD AGTLVRTRVS EISIVGRNTQ GVILIRTAED ENVVGLQRVA EPVDEEDLDT IDGSAAEGDD EIAPEVDVDD EPEEELEVLF UniProtKB: DNA gyrase subunit A |
-分子 #2: DNA gyrase subunit B
| 分子 | 名称: DNA gyrase subunit B / タイプ: protein_or_peptide / ID: 2 / コピー数: 2 / 光学異性体: LEVO / EC番号: DNA topoisomerase (ATP-hydrolysing) |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 90.891734 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: GPSNSYDSSS IKVLKGLDAV RKRPGMYIGD TDDGTGLHHM VFEVVDNAID EALAGHCKEI IVTIHADNSV SVQDDGRGIP TGIHPEEGV SAAEVIMTVL HAGGKFDDNS YKVSGGLHGV GVSVVNALSQ KLELVIQREG KIHRQIYEHG VPQAPLAVTG E TEKTGTMV ...文字列: GPSNSYDSSS IKVLKGLDAV RKRPGMYIGD TDDGTGLHHM VFEVVDNAID EALAGHCKEI IVTIHADNSV SVQDDGRGIP TGIHPEEGV SAAEVIMTVL HAGGKFDDNS YKVSGGLHGV GVSVVNALSQ KLELVIQREG KIHRQIYEHG VPQAPLAVTG E TEKTGTMV RFWPSLETFT NVTEFEYEIL AKRLRELSFL NSGVSIRLRD KRDGKEDHFH YEGGIKAFVE YLNKNKTPIH PN IFYFSTE KDGIGVEVAL QWNDGFQENI YCFTNNIPQR DGGTHLAGFR AAMTRTLNAY MDKEGYSKKA KVSATGDDAR EGL IAVVSV KVPDPKFSSQ TKDKLVSSEV KSAVEQQMNE LLAEYLLENP TDAKIVVGKI IDAARAREAA RRAREMTRRK GALD LAGLP GKLADCQERD PALSELYLVE GDSAGGSAKQ GRNRKNQAIL PLKGKILNVE KARFDKMLSS QEVATLITAL GCGIG RDEY NPDKLRYHSI IIMTDADVDG SHIRTLLLTF FYRQMPEIVE RGHVYIAQPP LYKVKKGKQE QYIKDDEAMD QYQISI ALD GATLHTNASA PALAGEALEK LVSEYNATQK MINRMERRYP KAMLKELIYQ PTLTEADLSD EQTVTRWVNA LVSELND KE QHGSQWKFDV HTNAEQNLFE PIVRVRTHGV DTDYPLDHEF ITGGEYRRIC TLGEKLRGLL EEDAFIERGE RRQPVASF E QALDWLVKES RRGLSIQRYK GLGEMNPEQL WETTMDPESR RMLRVTVKDA IAADQLFTTL MGDAVEPRRA FIEENALKA ANIDIENLYF Q UniProtKB: DNA gyrase subunit B |
-分子 #3: Mu217 DNA (159-MER)
| 分子 | 名称: Mu217 DNA (159-MER) / タイプ: dna / ID: 3 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:  Escherichia phage Mu (ファージ) Escherichia phage Mu (ファージ) |
| 分子量 | 理論値: 65.926109 KDa |
| 配列 | 文字列: (DT)(DT)(DC)(DC)(DT)(DG)(DC)(DG)(DC)(DG) (DT)(DC)(DC)(DT)(DT)(DA)(DT)(DA)(DT)(DG) (DG)(DG)(DG)(DC)(DT)(DG)(DG)(DA)(DT) (DA)(DA)(DG)(DA)(DC)(DC)(DG)(DG)(DG)(DG) (DA) (DA)(DC)(DA)(DG)(DG) ...文字列: (DT)(DT)(DC)(DC)(DT)(DG)(DC)(DG)(DC)(DG) (DT)(DC)(DC)(DT)(DT)(DA)(DT)(DA)(DT)(DG) (DG)(DG)(DG)(DC)(DT)(DG)(DG)(DA)(DT) (DA)(DA)(DG)(DA)(DC)(DC)(DG)(DG)(DG)(DG) (DA) (DA)(DC)(DA)(DG)(DG)(DA)(DA)(DA) (DT)(DT)(DT)(DT)(DT)(DG)(DA)(DT)(DG)(DC) (DC)(DA) (DT)(DC)(DA)(DG)(DA)(DA)(DA) (DA)(DC)(DG)(DC)(DG)(DT)(DC)(DA)(DG)(DC) (DG)(DC)(DC) (DG)(DC)(DT)(DC)(DT)(DG) (DA)(DG)(DG)(DC)(DA)(DA)(DT)(DA)(DA)(DA) (DC)(DA)(DG)(DA) (DA)(DT)(DC)(DA)(DG) (DG)(DC)(DA)(DT)(DA)(DA)(DA)(DA)(DT)(DC) (DA)(DC)(DC)(DC)(DG) (DC)(DA)(DC)(DA) (DG)(DA)(DT)(DT)(DT)(DT)(DT)(DT)(DA)(DA) (DA)(DA)(DC)(DG)(DC)(DG) (DC)(DC)(DA) (DC)(DG)(DG)(DG)(DA)(DT)(DT)(DT)(DT)(DT) (DA)(DA)(DA)(DC)(DC)(DG)(DG) (DT)(DA) (DT)(DT)(DT)(DA)(DA)(DC)(DG)(DG)(DT)(DG) (DT)(DA)(DT)(DG)(DA)(DA)(DT)(DC) (DC) (DC)(DG)(DT)(DT)(DT)(DT)(DA)(DT)(DC)(DT) (DT)(DC)(DC)(DT)(DT)(DT)(DC)(DA)(DC) (DT)(DT)(DT)(DC)(DT)(DT)(DT)(DC)(DT)(DC) (DC)(DA)(DG)(DT) GENBANK: GENBANK: M74911.1 |
-分子 #4: Mu217 DNA (159-MER)
| 分子 | 名称: Mu217 DNA (159-MER) / タイプ: dna / ID: 4 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:  Escherichia phage Mu (ファージ) Escherichia phage Mu (ファージ) |
| 分子量 | 理論値: 66.202258 KDa |
| 配列 | 文字列: (DA)(DC)(DT)(DG)(DG)(DA)(DG)(DA)(DA)(DA) (DG)(DA)(DA)(DA)(DG)(DT)(DG)(DA)(DA)(DA) (DG)(DG)(DA)(DA)(DG)(DA)(DT)(DA)(DA) (DA)(DA)(DC)(DG)(DG)(DG)(DA)(DT)(DT)(DC) (DA) (DT)(DA)(DC)(DA)(DC) ...文字列: (DA)(DC)(DT)(DG)(DG)(DA)(DG)(DA)(DA)(DA) (DG)(DA)(DA)(DA)(DG)(DT)(DG)(DA)(DA)(DA) (DG)(DG)(DA)(DA)(DG)(DA)(DT)(DA)(DA) (DA)(DA)(DC)(DG)(DG)(DG)(DA)(DT)(DT)(DC) (DA) (DT)(DA)(DC)(DA)(DC)(DC)(DG)(DT) (DT)(DA)(DA)(DA)(DT)(DA)(DC)(DC)(DG)(DG) (DT)(DT) (DT)(DA)(DA)(DA)(DA)(DA)(DT) (DC)(DC)(DC)(DG)(DT)(DG)(DG)(DC)(DG)(DC) (DG)(DT)(DT) (DT)(DT)(DA)(DA)(DA)(DA) (DA)(DA)(DT)(DC)(DT)(DG)(DT)(DG)(DC)(DG) (DG)(DG)(DT)(DG) (DA)(DT)(DT)(DT)(DT) (DA)(DT)(DG)(DC)(DC)(DT)(DG)(DA)(DT)(DT) (DC)(DT)(DG)(DT)(DT) (DT)(DA)(DT)(DT) (DG)(DC)(DC)(DT)(DC)(DA)(DG)(DA)(DG)(DC) (DG)(DG)(DC)(DG)(DC)(DT) (DG)(DA)(DC) (DG)(DC)(DG)(DT)(DT)(DT)(DT)(DC)(DT)(DG) (DA)(DT)(DG)(DG)(DC)(DA)(DT) (DC)(DA) (DA)(DA)(DA)(DA)(DT)(DT)(DT)(DC)(DC)(DT) (DG)(DT)(DT)(DC)(DC)(DC)(DC)(DG) (DG) (DT)(DC)(DT)(DT)(DA)(DT)(DC)(DC)(DA)(DG) (DC)(DC)(DC)(DC)(DA)(DT)(DA)(DT)(DA) (DA)(DG)(DG)(DA)(DC)(DG)(DC)(DG)(DC)(DA) (DG)(DG)(DA)(DA) GENBANK: GENBANK: M74911.1 |
-分子 #5: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 5 / コピー数: 2 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / チャンバー内温度: 283 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 41.84 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.1 µm / 最小 デフォーカス(公称値): 0.9 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | プロトコル: AB INITIO MODEL |
|---|---|
| 得られたモデル |  PDB-9gbv: |
 ムービー
ムービー コントローラー
コントローラー












 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)