+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-5114 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | BK channel with membrane density subtracted | |||||||||
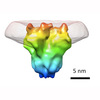
 マップデータ マップデータ | Surface rendering of the inside-out BK channel map with membrane density subtracted. The mesh shows a membrane-patch difference map. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Potassium channel | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 17.0 Å | |||||||||
 データ登録者 データ登録者 | Wang L / Sigworth FJ | |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2009 ジャーナル: Nature / 年: 2009タイトル: Structure of the BK potassium channel in a lipid membrane from electron cryomicroscopy. 著者: Liguo Wang / Fred J Sigworth /  要旨: A long-sought goal in structural biology has been the imaging of membrane proteins in their membrane environments. This goal has been achieved with electron crystallography in those special cases ...A long-sought goal in structural biology has been the imaging of membrane proteins in their membrane environments. This goal has been achieved with electron crystallography in those special cases where a protein forms highly ordered arrays in lipid bilayers. It has also been achieved by NMR methods in proteins up to 50 kilodaltons (kDa) in size, although milligram quantities of protein and isotopic labelling are required. For structural analysis of large soluble proteins in microgram quantities, an increasingly powerful method that does not require crystallization is single-particle reconstruction from electron microscopy of cryogenically cooled samples (electron cryomicroscopy (cryo-EM)). Here we report the first single-particle cryo-EM study of a membrane protein, the human large-conductance calcium- and voltage-activated potassium channel (BK), in a lipid environment. The new method is called random spherically constrained (RSC) single-particle reconstruction. BK channels, members of the six-transmembrane-segment (6TM) ion channel family, were reconstituted at low density into lipid vesicles (liposomes), and their function was verified by a potassium flux assay. Vesicles were also frozen in vitreous ice and imaged in an electron microscope. From images of 8,400 individual protein particles, a three-dimensional (3D) reconstruction of the BK channel and its membrane environment was obtained at a resolution of 1.7-2.0 nm. Not requiring the formation of crystals, the RSC approach promises to be useful in the structural study of many other membrane proteins as well. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_5114.map.gz emd_5114.map.gz | 3.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-5114-v30.xml emd-5114-v30.xml emd-5114.xml emd-5114.xml | 9.4 KB 9.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_5114_1.jpg emd_5114_1.jpg | 62.5 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-5114 http://ftp.pdbj.org/pub/emdb/structures/EMD-5114 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-5114 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-5114 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_5114_validation.pdf.gz emd_5114_validation.pdf.gz | 77.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_5114_full_validation.pdf.gz emd_5114_full_validation.pdf.gz | 76.9 KB | 表示 | |
| XML形式データ |  emd_5114_validation.xml.gz emd_5114_validation.xml.gz | 493 B | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5114 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5114 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5114 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5114 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_5114.map.gz / 形式: CCP4 / 大きさ: 3.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_5114.map.gz / 形式: CCP4 / 大きさ: 3.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Surface rendering of the inside-out BK channel map with membrane density subtracted. The mesh shows a membrane-patch difference map. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.5334 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
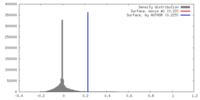
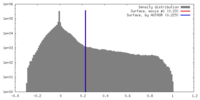
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : hSlo protein was reconstituted into POPC liposomes.
| 全体 | 名称: hSlo protein was reconstituted into POPC liposomes. |
|---|---|
| 要素 |
|
-超分子 #1000: hSlo protein was reconstituted into POPC liposomes.
| 超分子 | 名称: hSlo protein was reconstituted into POPC liposomes. / タイプ: sample / ID: 1000 / 集合状態: tetramer of human Slo1 subunits / Number unique components: 1 |
|---|---|
| 分子量 | 実験値: 500 KDa / 理論値: 500 KDa |
-分子 #1: hSlo
| 分子 | 名称: hSlo / タイプ: protein_or_peptide / ID: 1 / Name.synonym: BK channel alpha subunit / コピー数: 4 / 集合状態: Tetramer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) / 別称: Human / 細胞: HEK cell line / 細胞中の位置: Plasma membrane Homo sapiens (ヒト) / 別称: Human / 細胞: HEK cell line / 細胞中の位置: Plasma membrane |
| 分子量 | 理論値: 125 KDa |
| 組換発現 | 生物種: HEK 293 cell / 組換プラスミド: pcDNA3 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 詳細: Solution outside proteoliposomes, 13.5mM KCl, 0.5mM NaCl, 0.1mM EDTA, 2mM Hepes |
|---|---|
| グリッド | 詳細: home-made holey carbon film |
| 凍結 | 凍結剤: ETHANE / 装置: OTHER / 手法: Manual blot for 2-4 seconds before plunging |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI F30 |
|---|---|
| 温度 | 平均: 93 K |
| 特殊光学系 | エネルギーフィルター - 名称: GIF エネルギーフィルター - エネルギー下限: 0.0 eV エネルギーフィルター - エネルギー上限: 30.0 eV |
| 日付 | 2008年7月15日 |
| 撮影 | カテゴリ: CCD / フィルム・検出器のモデル: GENERIC CCD / 平均電子線量: 20 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.0 mm / 最大 デフォーカス(公称値): 5.0 µm / 最小 デフォーカス(公称値): 2.5 µm / 倍率(公称値): 50000 |
| 試料ステージ | 試料ホルダー: Eucentric / 試料ホルダーモデル: GATAN LIQUID NITROGEN |
| 実験機器 |  モデル: Tecnai F30 / 画像提供: FEI Company |
- 画像解析
画像解析
| CTF補正 | 詳細: Each micrograph |
|---|---|
| 最終 再構成 | アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 17.0 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: Homemade Matlab code 詳細: Alignment and reconstruction were modeled on Frealign but with constraints on theta and phi according to the spherical geometry of lipid vesicles. 使用した粒子像数: 3400 |
| 最終 角度割当 | 詳細: phi 90degrees, theta 180 degrees, psi 360 degrees |
 ムービー
ムービー コントローラー
コントローラー












 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)