+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Endophilin B1 dimer bound to nanodisc center | |||||||||
 マップデータ マップデータ | Focused map of an Endophilin B1 dimer bound to the center of a nanodisc | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | BAR / N-BAR / endophilin / membrane / curvature / cardiolipin / MSP2N2 / nanodisc / peripheral membrane protein / APOPTOSIS | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報lysophosphatidic acid acyltransferase activity / 'de novo' post-translational protein folding / positive regulation of membrane tubulation / phosphatidic acid biosynthetic process / autophagic cell death / protein localization to vacuolar membrane / positive regulation of autophagosome assembly / phosphatidylinositol 3-kinase activator activity / receptor catabolic process / membrane fission ...lysophosphatidic acid acyltransferase activity / 'de novo' post-translational protein folding / positive regulation of membrane tubulation / phosphatidic acid biosynthetic process / autophagic cell death / protein localization to vacuolar membrane / positive regulation of autophagosome assembly / phosphatidylinositol 3-kinase activator activity / receptor catabolic process / membrane fission / membrane organization / positive regulation of protein targeting to mitochondrion / autophagosome membrane / regulation of macroautophagy / cellular response to glucose starvation / positive regulation of autophagy / cellular response to amino acid starvation / regulation of cytokinesis / fatty acid binding / positive regulation of protein-containing complex assembly / regulation of protein stability / autophagy / midbody / cytoplasmic vesicle / mitochondrial outer membrane / nuclear body / cadherin binding / Golgi membrane / apoptotic process / protein homodimerization activity / protein-containing complex / identical protein binding / membrane / cytosol / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
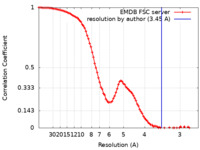
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.45 Å | |||||||||
 データ登録者 データ登録者 | Thorlacius A / Sundborger-Lunna A | |||||||||
| 資金援助 |  スウェーデン, 1件 スウェーデン, 1件
| |||||||||
 引用 引用 |  ジャーナル: Commun Biol / 年: 2025 ジャーナル: Commun Biol / 年: 2025タイトル: Peripheral membrane protein endophilin B1 probes, perturbs and permeabilizes lipid bilayers. 著者: Arni Thorlacius / Maksim Rulev / Oscar Sundberg / Anna Sundborger-Lunna /  要旨: Bin/Amphiphysin/Rvs167 (BAR) domain containing proteins are peripheral membrane proteins that regulate intracellular membrane curvature. BAR protein endophilin B1 plays a key role in multiple ...Bin/Amphiphysin/Rvs167 (BAR) domain containing proteins are peripheral membrane proteins that regulate intracellular membrane curvature. BAR protein endophilin B1 plays a key role in multiple cellular processes critical for oncogenesis, including autophagy and apoptosis. Amphipathic regions in endophilin B1 drive membrane association and tubulation through membrane scaffolding. Our understanding of exactly how BAR proteins like endophilin B1 promote highly diverse intracellular membrane remodeling events in the cell is severely limited due to lack of high-resolution structural information. Here we present the highest resolution cryo-EM structure of a BAR protein to date and the first structures of a BAR protein bound to a lipid bicelle. Using neural networks, we can effectively sort particle species of different stoichiometries, revealing the tremendous flexibility of post-membrane binding, pre-polymer BAR dimer organization and membrane deformation. We also show that endophilin B1 efficiently permeabilizes negatively charged liposomes that contain mitochondria-specific lipid cardiolipin and propose a new model for Bax-mediated cell death. #1:  ジャーナル: Biorxiv / 年: 2024 ジャーナル: Biorxiv / 年: 2024タイトル: Peripheral membrane protein endophilin B1 probes, perturbs and permeabilizes lipid bilayers 著者: Thorlacius A / Rulev M / Sundberg O / Sundborger-Lunna A | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_50984.map.gz emd_50984.map.gz | 32.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-50984-v30.xml emd-50984-v30.xml emd-50984.xml emd-50984.xml | 16.7 KB 16.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_50984_fsc.xml emd_50984_fsc.xml | 11.8 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_50984.png emd_50984.png | 24.9 KB | ||
| マスクデータ |  emd_50984_msk_1.map emd_50984_msk_1.map | 64 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-50984.cif.gz emd-50984.cif.gz | 6.3 KB | ||
| その他 |  emd_50984_half_map_1.map.gz emd_50984_half_map_1.map.gz emd_50984_half_map_2.map.gz emd_50984_half_map_2.map.gz | 59.4 MB 59.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-50984 http://ftp.pdbj.org/pub/emdb/structures/EMD-50984 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-50984 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-50984 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_50984_validation.pdf.gz emd_50984_validation.pdf.gz | 805.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_50984_full_validation.pdf.gz emd_50984_full_validation.pdf.gz | 804.9 KB | 表示 | |
| XML形式データ |  emd_50984_validation.xml.gz emd_50984_validation.xml.gz | 14.8 KB | 表示 | |
| CIF形式データ |  emd_50984_validation.cif.gz emd_50984_validation.cif.gz | 20.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-50984 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-50984 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-50984 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-50984 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_50984.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_50984.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Focused map of an Endophilin B1 dimer bound to the center of a nanodisc | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.328 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_50984_msk_1.map emd_50984_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_50984_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_50984_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Endophilin B1 dimers bound to a nanodisc
| 全体 | 名称: Endophilin B1 dimers bound to a nanodisc |
|---|---|
| 要素 |
|
-超分子 #1: Endophilin B1 dimers bound to a nanodisc
| 超分子 | 名称: Endophilin B1 dimers bound to a nanodisc / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Endophilin-B1
| 分子 | 名称: Endophilin-B1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 40.843246 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MNIMDFNVKK LAADAGTFLS RAVQFTEEKL GQAEKTELDA HLENLLSKAE CTKIWTEKIM KQTEVLLQPN PNARIEEFVY EKLDRKAPS RINNPELLGQ YMIDAGTEFG PGTAYGNALI KCGETQKRIG TADRELIQTS ALNFLTPLRN FIEGDYKTIA K ERKLLQNK ...文字列: MNIMDFNVKK LAADAGTFLS RAVQFTEEKL GQAEKTELDA HLENLLSKAE CTKIWTEKIM KQTEVLLQPN PNARIEEFVY EKLDRKAPS RINNPELLGQ YMIDAGTEFG PGTAYGNALI KCGETQKRIG TADRELIQTS ALNFLTPLRN FIEGDYKTIA K ERKLLQNK RLDLDAAKTR LKKAKAAETR NSSEQELRIT QSEFDRQAEI TRLLLEGISS THAHHLRCLN DFVEAQMTYY AQ CYQYMLD LQKQLGSFPS NYLSNNNQTS VTPVPSVLPN AIGSSAMAST SGLVITSPSN LSDLKECSGS RKARVLYDYD AAN STELSL LADEVITVFS VVGMDSDWLM GERGNQKGKV PITYLELLN UniProtKB: Endophilin-B1 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1.8 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 / 詳細: 20 mM Tris-HCl, 100 mM NaCl, 0.5 mM EDTA, pH 7.4 |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 40.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.2 µm / 最小 デフォーカス(公称値): 1.2 µm / 倍率(公称値): 130000 |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | PDB ID: Chain - Chain ID: A / Chain - Residue range: 11-252 / Chain - Source name: AlphaFold / Chain - Initial model type: in silico model |
|---|---|
| 詳細 | Alphafold model was processed in PHENIX, docked into the density map using UCSF Chimera, refined using all-chain refinement in coot followed by real-space refinement with Servalcat. |
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
| 得られたモデル |  PDB-9g2u: |
 ムービー
ムービー コントローラー
コントローラー









 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)