+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of the BcsEFRQ regulatory subcomplex for E. coli cellulose secretion in non-saturating c-di-GMP (local) | |||||||||
 マップデータ マップデータ | Deep Emhancer-sharpened map of the BcsE2F2 regulatory subcomplex from the E. coli cellulose secretion system in non-saturating c-di-GMP (local) | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Bacterial cellulose secretion / MEMBRANE PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報negative regulation of cell division / cytoplasmic side of plasma membrane / cell division / ATP hydrolysis activity / ATP binding / cytosol 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.76 Å | |||||||||
 データ登録者 データ登録者 | Anso I / Krasteva PV | |||||||||
| 資金援助 | European Union, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2024 ジャーナル: Nat Commun / 年: 2024タイトル: Structural basis for synthase activation and cellulose modification in the E. coli Type II Bcs secretion system. 著者: Itxaso Anso / Samira Zouhir / Thibault Géry Sana / Petya Violinova Krasteva /   要旨: Bacterial cellulosic polymers constitute a prevalent class of biofilm matrix exopolysaccharides that are synthesized by several types of bacterial cellulose secretion (Bcs) systems, which include ...Bacterial cellulosic polymers constitute a prevalent class of biofilm matrix exopolysaccharides that are synthesized by several types of bacterial cellulose secretion (Bcs) systems, which include conserved cyclic diguanylate (c-di-GMP)-dependent cellulose synthase modules together with diverse accessory subunits. In E. coli, the biogenesis of phosphoethanolamine (pEtN)-modified cellulose relies on the BcsRQABEFG macrocomplex, encompassing inner-membrane and cytosolic subunits, and an outer membrane porin, BcsC. Here, we use cryogenic electron microscopy to shed light on the molecular mechanisms of BcsA-dependent recruitment and stabilization of a trimeric BcsG pEtN-transferase for polymer modification, and a dimeric BcsF-dependent recruitment of an otherwise cytosolic BcsERQ regulatory complex. We further demonstrate that BcsE, a secondary c-di-GMP sensor, can remain dinucleotide-bound and retain the essential-for-secretion BcsRQ partners onto the synthase even in the absence of direct c-di-GMP-synthase complexation, likely lowering the threshold for c-di-GMP-dependent synthase activation. Such activation-by-proxy mechanism could allow Bcs secretion system activity even in the absence of substantial intracellular c-di-GMP increase, and is reminiscent of other widespread synthase-dependent polysaccharide secretion systems where dinucleotide sensing and/or synthase stabilization are carried out by key co-polymerase subunits. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_50633.map.gz emd_50633.map.gz | 2.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-50633-v30.xml emd-50633-v30.xml emd-50633.xml emd-50633.xml | 25.7 KB 25.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_50633_fsc.xml emd_50633_fsc.xml | 16.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_50633.png emd_50633.png | 50.2 KB | ||
| マスクデータ |  emd_50633_msk_1.map emd_50633_msk_1.map | 476.8 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-50633.cif.gz emd-50633.cif.gz | 7.6 KB | ||
| その他 |  emd_50633_additional_1.map.gz emd_50633_additional_1.map.gz emd_50633_half_map_1.map.gz emd_50633_half_map_1.map.gz emd_50633_half_map_2.map.gz emd_50633_half_map_2.map.gz | 224.8 MB 441.8 MB 441.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-50633 http://ftp.pdbj.org/pub/emdb/structures/EMD-50633 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-50633 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-50633 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_50633_validation.pdf.gz emd_50633_validation.pdf.gz | 877.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_50633_full_validation.pdf.gz emd_50633_full_validation.pdf.gz | 877 KB | 表示 | |
| XML形式データ |  emd_50633_validation.xml.gz emd_50633_validation.xml.gz | 25.8 KB | 表示 | |
| CIF形式データ |  emd_50633_validation.cif.gz emd_50633_validation.cif.gz | 34.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-50633 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-50633 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-50633 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-50633 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  9fp2MC  9fmtC  9fmvC  9fmzC  9fnnC  9fo7C  9fp0C C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_50633.map.gz / 形式: CCP4 / 大きさ: 476.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_50633.map.gz / 形式: CCP4 / 大きさ: 476.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Deep Emhancer-sharpened map of the BcsE2F2 regulatory subcomplex from the E. coli cellulose secretion system in non-saturating c-di-GMP (local) | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.839 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_50633_msk_1.map emd_50633_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Unsharpened map of the BcsE2F2 regulatory subcomplex from...
| ファイル | emd_50633_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unsharpened map of the BcsE2F2 regulatory subcomplex from the E. coli cellulose secretion system in non-saturating c-di-GMP (local) | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half-map of the BcsE2F2 regulatory subcomplex from the...
| ファイル | emd_50633_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half-map of the BcsE2F2 regulatory subcomplex from the E. coli cellulose secretion system in non-saturating c-di-GMP (local) | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half-map of the BcsE2F2 regulatory subcomplex from the...
| ファイル | emd_50633_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half-map of the BcsE2F2 regulatory subcomplex from the E. coli cellulose secretion system in non-saturating c-di-GMP (local) | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Locally refined BcsEFRQ regulatory subcomplex in non-saturating c...
| 全体 | 名称: Locally refined BcsEFRQ regulatory subcomplex in non-saturating c-di-GMP from the E. coli cellulose secretion system |
|---|---|
| 要素 |
|
-超分子 #1: Locally refined BcsEFRQ regulatory subcomplex in non-saturating c...
| 超分子 | 名称: Locally refined BcsEFRQ regulatory subcomplex in non-saturating c-di-GMP from the E. coli cellulose secretion system タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#4 詳細: Local refinement in the context of an assembled macrocomplex |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 990 KDa |
-分子 #1: Cyclic di-GMP binding protein BcsE
| 分子 | 名称: Cyclic di-GMP binding protein BcsE / タイプ: protein_or_peptide / ID: 1 詳細: Purified Bcs macrocomplex with stoichiometry BcsA-BcsB6-BcsR2-BcsQ2-BcsE2-BcsF2-BcsG3 コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 60.967539 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MASWSHPQFE KGSMRDIVDP VFSIGISSLW DELRHMPAGG VWWFNVDRHE DAISLANQTI ASQAETAHVA VISMDSDPAK IFQLDDSQG PEKIKLFSML NHEKGLYYLT RDLQCSIDPH NYLFILVCAN NAWQNIPAER LRSWLDKMNK WSRLNHCSLL V INPGNNND ...文字列: MASWSHPQFE KGSMRDIVDP VFSIGISSLW DELRHMPAGG VWWFNVDRHE DAISLANQTI ASQAETAHVA VISMDSDPAK IFQLDDSQG PEKIKLFSML NHEKGLYYLT RDLQCSIDPH NYLFILVCAN NAWQNIPAER LRSWLDKMNK WSRLNHCSLL V INPGNNND KQFSLLLEEY RSLFGLASLR FQGDQHLLDI AFWCNEKGVS ARQQLSVQQQ NGIWTLVQSE EAEIQPRSDE KR ILSNVAV LEGAPPLSEH WQLFNNNEVL FNEARTAQAA TVVFSLQQNA QIEPLARSIH TLRRQRGSAM KILVRENTAS LRA TDERLL LACGANMVIP WNAPLSRCLT MIESVQGQKF SRYVPEDITT LLSMTQPLKL RGFQKWDVFC NAVNNMMNNP LLPA HGKGV LVALRPVPGI RVEQALTLCR PNRTGDIMTI GGNRLVLFLS FCRINDLDTA LNHIFPLPTG DIFSNRMVWF EDDQI SAEL VQMRLLAPEQ WGMPLPLTQS SKPVINAEHD GRHWRRIPEP MRLLDDAVER SS |
-分子 #2: Cell division protein
| 分子 | 名称: Cell division protein / タイプ: protein_or_peptide / ID: 2 詳細: Purified Bcs macrocomplex with stoichiometry BcsA-BcsB6-BcsR2-BcsQ2-BcsE2-BcsF2-BcsG3 コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 27.960832 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MAVLGLQGVR GGVGTTTITA ALAWSLQMLG ENVLVVDACP DNLLRLSFNV DFTHRQGWAR AMLDGQDWRD AGLRYTSQLD LLPFGQLSI EEQENPQHWQ TRLSDICSGL QQLKASGRYQ WILIDLPRDA SQITHQLLSL CDHSLAIVNV DANCHIRLHQ Q ALPDGAHI ...文字列: MAVLGLQGVR GGVGTTTITA ALAWSLQMLG ENVLVVDACP DNLLRLSFNV DFTHRQGWAR AMLDGQDWRD AGLRYTSQLD LLPFGQLSI EEQENPQHWQ TRLSDICSGL QQLKASGRYQ WILIDLPRDA SQITHQLLSL CDHSLAIVNV DANCHIRLHQ Q ALPDGAHI LINNFRIGSQ VQDDIYQLWL QSQRRLLPML IHRDEAMAEC LAAKQPVGEY RSDALAAEEI LTLANWCLLN YS GLKTPVG SKS UniProtKB: Cell division protein |
-分子 #3: Protein YhjR
| 分子 | 名称: Protein YhjR / タイプ: protein_or_peptide / ID: 3 詳細: Purified Bcs macrocomplex with stoichiometry BcsA-BcsB6-BcsR2-BcsQ2-BcsE2-BcsF2-BcsG3 コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 8.645541 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MGSSHHHHHH HHAAGSNNNE PDTLPDPAIG YIFQNDIVAL KQAFSLPDID YADISQREQL AAALKRWPLL AEFAQQK |
-分子 #4: Cellulose biosynthesis protein BcsF
| 分子 | 名称: Cellulose biosynthesis protein BcsF / タイプ: protein_or_peptide / ID: 4 詳細: Purified Bcs macrocomplex with stoichiometry BcsA-BcsB6-BcsR2-BcsQ2-BcsE2-BcsF2-BcsG3 コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 7.378975 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MMTISDIIEI IVVCALIFFP LGYLARHSLR RIRDTLRLFF AKPRYVKPAG TLRRTEKARA TKK |
-分子 #5: 9,9'-[(2R,3R,3aS,5S,7aR,9R,10R,10aS,12S,14aR)-3,5,10,12-tetrahydr...
| 分子 | 名称: 9,9'-[(2R,3R,3aS,5S,7aR,9R,10R,10aS,12S,14aR)-3,5,10,12-tetrahydroxy-5,12-dioxidooctahydro-2H,7H-difuro[3,2-d:3',2'-j][1,3,7,9,2,8]tetraoxadiphosphacyclododecine-2,9-diyl]bis(2-amino-1,9-dihydro-6H-purin-6-one) タイプ: ligand / ID: 5 / コピー数: 4 / 式: C2E |
|---|---|
| 分子量 | 理論値: 690.411 Da |
| Chemical component information | 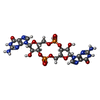 ChemComp-C2E: |
-分子 #6: ADENOSINE-5'-TRIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-TRIPHOSPHATE / タイプ: ligand / ID: 6 / コピー数: 2 / 式: ATP |
|---|---|
| 分子量 | 理論値: 507.181 Da |
| Chemical component information |  ChemComp-ATP: |
-分子 #7: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 7 / コピー数: 2 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2 mg/mL |
|---|---|
| 緩衝液 | pH: 8 詳細: 120 mM NaCL 20 mM HEPES pH8 5 mM MgCl2 10 uM ApppCp 4 uM c-di-GMP 0.01% LM-NPG |
| グリッド | モデル: UltrAuFoil R1.2/1.3 / メッシュ: 300 / 支持フィルム - 材質: GOLD / 支持フィルム - トポロジー: HOLEY |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K |
| 詳細 | Purified Bcs macrocomplex with stoichiometry BcsA-BcsB6-BcsR2-BcsQ2-BcsE2-BcsF2-BcsG3 |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Quantum LS / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 撮影したグリッド数: 2 / 実像数: 20022 / 平均電子線量: 49.35 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.1 µm / 最小 デフォーカス(公称値): 0.3 µm |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル |
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT | |||||||||
| 得られたモデル |  PDB-9fp2: |
 ムービー
ムービー コントローラー
コントローラー












 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)























































