登録情報 データベース : EMDB / ID : EMD-50183タイトル cryo-EM structure of LST2 TOS peptide bound to human mTOR complex 1 複合体 : mTORC1 in complex with LST2 TOS peptideタンパク質・ペプチド : Serine/threonine-protein kinase mTORタンパク質・ペプチド : Target of rapamycin complex subunit LST8タンパク質・ペプチド : Regulatory-associated protein of mTORタンパク質・ペプチド : Lateral signaling target protein 2 homologリガンド : INOSITOL HEXAKISPHOSPHATE / / / / / / 機能・相同性 分子機能 ドメイン・相同性 構成要素
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 Homo sapiens (ヒト)手法 / / 解像度 : 3.68 Å Craigie LM / Maier T 資金援助 Organization Grant number 国 Swiss National Science Foundation 179323 H2020 Marie Curie Actions of the European Commission 812830 European Union
ジャーナル : Proc Natl Acad Sci U S A / 年 : 2024タイトル : mTORC1 phosphorylates and stabilizes LST2 to negatively regulate EGFR.著者 : Stefania Battaglioni / Louise-Marie Craigie / Sofia Filippini / Timm Maier / Michael N Hall / 要旨 : TORC1 (target of rapamycin complex 1) is a highly conserved protein kinase that plays a central role in regulating cell growth. Given the role of mammalian TORC1 (mTORC1) in metabolism and disease, ... TORC1 (target of rapamycin complex 1) is a highly conserved protein kinase that plays a central role in regulating cell growth. Given the role of mammalian TORC1 (mTORC1) in metabolism and disease, understanding mTORC1 downstream signaling and feedback loops is important. mTORC1 recognizes some of its substrates via a five amino acid binding sequence called the TOR signaling (TOS) motif. mTORC1 binding to a TOS motif facilitates phosphorylation of a distinct, distal site. Here, we show that LST2, also known as ZFYVE28, contains a TOS motif (amino acids 401 to 405) and is directly phosphorylated by mTORC1 at serine 670 (S670). mTORC1-mediated S670 phosphorylation promotes LST2 monoubiquitination on lysine 87 (K87). Monoubiquitinated LST2 is stable and displays a broad reticular distribution. When mTORC1 is inactive, unphosphorylated LST2 is degraded by the proteasome. The absence of LST2 enhances EGFR (epidermal growth factor receptor) signaling. We propose that mTORC1 negatively feeds back on its upstream receptor EGFR via LST2. 履歴 登録 2024年4月26日 - ヘッダ(付随情報) 公開 2024年8月14日 - マップ公開 2024年8月14日 - 更新 2024年8月28日 - 現状 2024年8月28日 処理サイト : PDBe / 状態 : 公開
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報
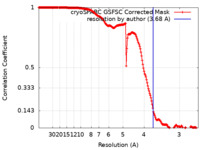
 マップデータ
マップデータ 試料
試料 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) データ登録者
データ登録者 スイス, European Union, 2件
スイス, European Union, 2件  引用
引用 ジャーナル: Proc Natl Acad Sci U S A / 年: 2024
ジャーナル: Proc Natl Acad Sci U S A / 年: 2024
 構造の表示
構造の表示 ダウンロードとリンク
ダウンロードとリンク emd_50183.map.gz
emd_50183.map.gz EMDBマップデータ形式
EMDBマップデータ形式 emd-50183-v30.xml
emd-50183-v30.xml emd-50183.xml
emd-50183.xml EMDBヘッダ
EMDBヘッダ emd_50183_fsc.xml
emd_50183_fsc.xml FSCデータファイル
FSCデータファイル emd_50183.png
emd_50183.png emd-50183.cif.gz
emd-50183.cif.gz emd_50183_half_map_1.map.gz
emd_50183_half_map_1.map.gz emd_50183_half_map_2.map.gz
emd_50183_half_map_2.map.gz http://ftp.pdbj.org/pub/emdb/structures/EMD-50183
http://ftp.pdbj.org/pub/emdb/structures/EMD-50183 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-50183
ftp://ftp.pdbj.org/pub/emdb/structures/EMD-50183 emd_50183_validation.pdf.gz
emd_50183_validation.pdf.gz EMDB検証レポート
EMDB検証レポート emd_50183_full_validation.pdf.gz
emd_50183_full_validation.pdf.gz emd_50183_validation.xml.gz
emd_50183_validation.xml.gz emd_50183_validation.cif.gz
emd_50183_validation.cif.gz https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-50183
https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-50183 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-50183
ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-50183 リンク
リンク EMDB (EBI/PDBe) /
EMDB (EBI/PDBe) /  EMDataResource
EMDataResource マップ
マップ ダウンロード / ファイル: emd_50183.map.gz / 形式: CCP4 / 大きさ: 512 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES)
ダウンロード / ファイル: emd_50183.map.gz / 形式: CCP4 / 大きさ: 512 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) 試料の構成要素
試料の構成要素 Homo sapiens (ヒト)
Homo sapiens (ヒト) Homo sapiens (ヒト)
Homo sapiens (ヒト)
 Homo sapiens (ヒト)
Homo sapiens (ヒト)
 Homo sapiens (ヒト)
Homo sapiens (ヒト)
 Homo sapiens (ヒト)
Homo sapiens (ヒト)
 解析
解析 試料調製
試料調製 電子顕微鏡法
電子顕微鏡法 FIELD EMISSION GUN
FIELD EMISSION GUN
 ムービー
ムービー コントローラー
コントローラー



























 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)