+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-4642 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | 12 Angstrom structure of detergent solubilised LAT1-CD98hc | |||||||||
 マップデータ マップデータ | 12 angstrom structure of LAT1-CD98hc | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 12.0 Å | |||||||||
 データ登録者 データ登録者 | Chiduza GN / Johnson R / Wright GS / Antonyuk S / Muench S / Hasnain SS | |||||||||
 引用 引用 |  ジャーナル: Acta Crystallogr D Struct Biol / 年: 2019 ジャーナル: Acta Crystallogr D Struct Biol / 年: 2019タイトル: LAT1 (SLC7A5) and CD98hc (SLC3A2) complex dynamics revealed by single-particle cryo-EM. 著者: George N Chiduza / Rachel M Johnson / Gareth S A Wright / Svetlana V Antonyuk / Stephen P Muench / S Samar Hasnain /  要旨: Solute carriers are a large class of transporters that play key roles in normal and disease physiology. Among the solute carriers, heteromeric amino-acid transporters (HATs) are unique in their ...Solute carriers are a large class of transporters that play key roles in normal and disease physiology. Among the solute carriers, heteromeric amino-acid transporters (HATs) are unique in their quaternary structure. LAT1-CD98hc, a HAT, transports essential amino acids and drugs across the blood-brain barrier and into cancer cells. It is therefore an important target both biologically and therapeutically. During the course of this work, cryo-EM structures of LAT1-CD98hc in the inward-facing conformation and in either the substrate-bound or apo states were reported to 3.3-3.5 Å resolution [Yan et al. (2019), Nature (London), 568, 127-130]. Here, these structures are analyzed together with our lower resolution cryo-EM structure, and multibody 3D auto-refinement against single-particle cryo-EM data was used to characterize the dynamics of the interaction of CD98hc and LAT1. It is shown that the CD98hc ectodomain and the LAT1 extracellular surface share no substantial interface. This allows the CD98hc ectodomain to have a high degree of movement within the extracellular space. The functional implications of these aspects are discussed together with the structure determination. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_4642.map.gz emd_4642.map.gz | 3.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-4642-v30.xml emd-4642-v30.xml emd-4642.xml emd-4642.xml | 11.7 KB 11.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
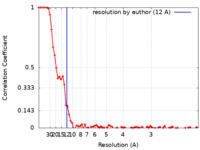
| FSC (解像度算出) |  emd_4642_fsc.xml emd_4642_fsc.xml | 8.4 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_4642.png emd_4642.png | 75.9 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-4642 http://ftp.pdbj.org/pub/emdb/structures/EMD-4642 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4642 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4642 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_4642_validation.pdf.gz emd_4642_validation.pdf.gz | 222.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_4642_full_validation.pdf.gz emd_4642_full_validation.pdf.gz | 221.9 KB | 表示 | |
| XML形式データ |  emd_4642_validation.xml.gz emd_4642_validation.xml.gz | 9.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4642 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4642 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4642 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4642 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_4642.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_4642.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | 12 angstrom structure of LAT1-CD98hc | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.06 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : LAT1-CD98hc
| 全体 | 名称: LAT1-CD98hc |
|---|---|
| 要素 |
|
-超分子 #1: LAT1-CD98hc
| 超分子 | 名称: LAT1-CD98hc / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) / 細胞中の位置: Plasma membrane Homo sapiens (ヒト) / 細胞中の位置: Plasma membrane |
| 分子量 | 理論値: 123 kDa/nm |
-分子 #1: LAT1
| 分子 | 名称: LAT1 / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MAGAGPKRRA LAAPAAEEKE EAREKMLAAK SADGSAPAGE GEGVTLQRNI TLLNGVAIIV GTIIGSGIF VTPTGVLKEA GSPGLALVVW AACGVFSIVG ALCYAELGTT ISKSGGDYAY M LEVYGSLP AFLKLWIELL IIRPSSQYIV ALVFATYLLK PLFPTCPVPE ...文字列: MAGAGPKRRA LAAPAAEEKE EAREKMLAAK SADGSAPAGE GEGVTLQRNI TLLNGVAIIV GTIIGSGIF VTPTGVLKEA GSPGLALVVW AACGVFSIVG ALCYAELGTT ISKSGGDYAY M LEVYGSLP AFLKLWIELL IIRPSSQYIV ALVFATYLLK PLFPTCPVPE EAAKLVACLC VL LLTAVNC YSVKAATRVQ DAFAAAKLLA LALIILLGFV QIGKGDVSNL DPNFSFEGTK LDV GNIVLA LYSGLFAYGG WNYLNFVTEE MINPYRNLPL AIIISLPIVT LVYVLTNLAY FTTL STEQM LSSEAVAVDF GNYHLGVMSW IIPVFVGLSC FGSVNGSLFT SSRLFFVGSR EGHLP SILS MIHPQLLTPV PSLVFTCVMT LLYAFSKDIF SVINFFSFFN WLCVALAIIG MIWLRH RKP ELERPIKVNL ALPVFFILAC LFLIAVSFWK TPVECGIGFT IILSGLPVYF FGVWWKN KP KWLLQGIFST TVLCQKLMQV VPQET |
-分子 #2: CD98hc
| 分子 | 名称: CD98hc / タイプ: protein_or_peptide / ID: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MELQPPEASI AVVSIPRQLP GSHSEAGVQG LSAGDDSELG SHCVAQTGLE LLASGDPLPS ASQNAEMIE TGSDCVTQAG LQLLASSDPP ALASKNAEVT GTMSQDTEVD MKEVELNELE P EKQPMNAA SGAAMSLAGA EKNGLVKIKV AEDEAEAAAA AKFTGLSKEE ...文字列: MELQPPEASI AVVSIPRQLP GSHSEAGVQG LSAGDDSELG SHCVAQTGLE LLASGDPLPS ASQNAEMIE TGSDCVTQAG LQLLASSDPP ALASKNAEVT GTMSQDTEVD MKEVELNELE P EKQPMNAA SGAAMSLAGA EKNGLVKIKV AEDEAEAAAA AKFTGLSKEE LLKVAGSPGW VR TRWALLL LFWLGWLGML AGAVVIIVRA PRCRELPAQK WWHTGALYRI GDLQAFQGHG AGN LAGLKG RLDYLSSLKV KGLVLGPIHK NQKDDVAQTD LLQIDPNFGS KEDFDSLLQS AKKK SIRVI LDLTPNYRGE NSWFSTQVDT VATKVKDALE FWLQAGVDGF QVRDIENLKD ASSFL AEWQ NITKGFSEDR LLIAGTNSSD LQQILSLLES NKDLLLTSSY LSDSGSTGEH TKSLVT QYL NATGNRWCSW SLSQARLLTS FLPAQLLRLY QLMLFTLPGT PVFSYGDEIG LDAAALP GQ PMEAPVMLWD ESSFPDIPGA VSANMTVKGQ SEDPGSLLSL FRRLSDQRSK ERSLLHGD F HAFSAGPGLF SYIRHWDQNE RFLVVLNFGD VGLSAGLQAS DLPASASLPA KADLLLSTQ PGREEGSPLE LERLKLEPHE GLLLRFPYAA |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2.3 mg/mL |
|---|---|
| 緩衝液 | pH: 8 詳細: Buffer: 100 mM Tris-Cl, 300 mM NaCl, 0.01% w/v DDM/LMNG/CHS (15:3:1), pH8 |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277.15 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | 位相板: VOLTA PHASE PLATE |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均電子線量: 4.96 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | PDB ID: Chain - Chain ID: A |
|---|---|
| 精密化 | プロトコル: RIGID BODY FIT |
 ムービー
ムービー コントローラー
コントローラー












 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)