+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of Tulane virus 9-6-17 variant capsid protein VP1 9-14-18 | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | virion capsid / capsid protein / Tulane virus / VIRUS | |||||||||
| 機能・相同性 | Calicivirus coat protein C-terminal / Calicivirus coat protein C-terminal / Calicivirus coat protein / Calicivirus coat protein / virion component / Viral coat protein subunit / host cell cytoplasm / Capsid protein 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Tulane virus (ウイルス) Tulane virus (ウイルス) | |||||||||
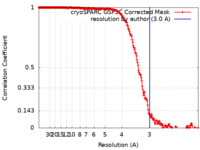
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.0 Å | |||||||||
 データ登録者 データ登録者 | Sun C / Jiang W | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Biomolecules / 年: 2024 ジャーナル: Biomolecules / 年: 2024タイトル: The 2.6 Å Structure of a Tulane Virus Variant with Minor Mutations Leading to Receptor Change. 著者: Chen Sun / Pengwei Huang / Xueyong Xu / Frank S Vago / Kunpeng Li / Thomas Klose / Xi Jason Jiang / Wen Jiang /  要旨: Human noroviruses (HuNoVs) are a major cause of acute gastroenteritis, contributing significantly to annual foodborne illness cases. However, studying these viruses has been challenging due to ...Human noroviruses (HuNoVs) are a major cause of acute gastroenteritis, contributing significantly to annual foodborne illness cases. However, studying these viruses has been challenging due to limitations in tissue culture techniques for over four decades. Tulane virus (TV) has emerged as a crucial surrogate for HuNoVs due to its close resemblance in amino acid composition and the availability of a robust cell culture system. Initially isolated from rhesus macaques in 2008, TV represents a novel belonging to the genus. Its significance lies in sharing the same host cell receptor, histo-blood group antigen (HBGA), as HuNoVs. In this study, we introduce, through cryo-electron microscopy (cryo-EM), the structure of a specific TV variant (the 9-6-17 TV) that has notably lost its ability to bind to its receptor, B-type HBGA-a finding confirmed using an enzyme-linked immunosorbent assay (ELISA). These results offer a profound insight into the genetic modifications occurring in TV that are necessary for adaptation to cell culture environments. This research significantly contributes to advancing our understanding of the genetic changes that are pivotal to successful adaptation, shedding light on fundamental aspects of evolution. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_45963.map.gz emd_45963.map.gz | 408 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-45963-v30.xml emd-45963-v30.xml emd-45963.xml emd-45963.xml | 15.5 KB 15.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_45963_fsc.xml emd_45963_fsc.xml | 19.8 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_45963.png emd_45963.png | 167.3 KB | ||
| マスクデータ |  emd_45963_msk_1.map emd_45963_msk_1.map | 824 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-45963.cif.gz emd-45963.cif.gz | 5.8 KB | ||
| その他 |  emd_45963_half_map_1.map.gz emd_45963_half_map_1.map.gz emd_45963_half_map_2.map.gz emd_45963_half_map_2.map.gz | 750.5 MB 750.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-45963 http://ftp.pdbj.org/pub/emdb/structures/EMD-45963 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-45963 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-45963 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_45963.map.gz / 形式: CCP4 / 大きさ: 824 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_45963.map.gz / 形式: CCP4 / 大きさ: 824 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.035 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_45963_msk_1.map emd_45963_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_45963_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_45963_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Tulane virus
| 全体 | 名称:  Tulane virus (ウイルス) Tulane virus (ウイルス) |
|---|---|
| 要素 |
|
-超分子 #1: Tulane virus
| 超分子 | 名称: Tulane virus / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: all 詳細: The Tulane virus was purified from the LLC-MK2 cell line. NCBI-ID: 512169 / 生物種: Tulane virus / Sci species strain: 9-6-17 / ウイルスタイプ: VIRION / ウイルス・単離状態: STRAIN / ウイルス・エンベロープ: No / ウイルス・中空状態: No |
|---|---|
| 宿主 | 生物種:  |
| 分子量 | 理論値: 57 MDa |
| ウイルス殻 | Shell ID: 1 / 直径: 400.0 Å |
-分子 #1: Capsid protein
| 分子 | 名称: Capsid protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Tulane virus (ウイルス) Tulane virus (ウイルス) |
| 分子量 | 理論値: 57.933172 KDa |
| 配列 | 文字列: MESSKTEQVT GATGITQSTV TAPLPEAVSS LSLAPTVNAL DPWVYLNQTE VPGGTFTVSS ATQPGSVLLE LEISPELNLY TSHLFRMYA GWSGGFSLKL LVAGNAFSAG KLIAAIIPPN IEVPNSAYLL TGFPHEILDF RTADSMEIIA PDIKNIDYHF R GDKLGKLV ...文字列: MESSKTEQVT GATGITQSTV TAPLPEAVSS LSLAPTVNAL DPWVYLNQTE VPGGTFTVSS ATQPGSVLLE LEISPELNLY TSHLFRMYA GWSGGFSLKL LVAGNAFSAG KLIAAIIPPN IEVPNSAYLL TGFPHEILDF RTADSMEIIA PDIKNIDYHF R GDKLGKLV VMVYSPLRST SADFEIEIKL TSAPLPDFKF TMLVPPIQNN ALPIWSIPQA PPYSMVNPRS PLTPVVELYI NS SYATCNH QLGRYTIYQG AIGNSTFNPS GAWTATCTAE AGSVTGHPNW RYALLDLPDN PTFDPTLPPV PRGFCDWGSG VKS GNKQHL VCFTGKKVEG GFQDVDTHMW DYGDNETVGL DNTYQRTIYI KDPSLEKDAQ YLVIPMGVSG AANDDTVQVA PNCY GSWDY APTVAPPLGE QFVWFRSQLP ASKTTTTSGV NSVPVNVNAL MSPDLMCSAY ASGFPLGKVA LLDYVLFGGS VVRQF KLYP EGYMTANTTG SNTGFIIPAD GYFRFNSWVS PSFMISSVVD LNLQTAVVFR UniProtKB: Capsid protein |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 / 詳細: PBS |
|---|---|
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 293.15 K / 装置: GATAN CRYOPLUNGE 3 |
| 詳細 | Purified virus |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 撮影したグリッド数: 1 / 実像数: 969 / 平均電子線量: 23.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: OTHER / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.2 µm / 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 81000 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー
















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)