+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of the Strand displacement Complex (III) of Yeast Mitochondrial DNA polymerase Gamma (MIP1) with downstream DNA | |||||||||
 マップデータ マップデータ | Anisotropic sharpened map of the original map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Mitochondrial DNA Polymerase Gamma / Strand displacement complex / MIP1 / REPLICATION / Helicase independent DNA polymerase / Transferase-DNA complex | |||||||||
| 機能・相同性 | :  機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.6 Å | |||||||||
 データ登録者 データ登録者 | Nayak AR / Sokolova VO / Sillamaa S / Sedmen J / Temiakov D / Zamudio-Ochoa A | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2025 ジャーナル: Nat Commun / 年: 2025タイトル: Structural basis for intrinsic strand displacement activity of mitochondrial DNA polymerase. 著者: Ashok R Nayak / Viktoriia Sokolova / Sirelin Sillamaa / Karl Herbine / Juhan Sedman / Dmitry Temiakov /   要旨: Members of the Pol A family of DNA polymerases, found across all domains of life, utilize various strategies for DNA strand separation during replication. In higher eukaryotes, mitochondrial DNA ...Members of the Pol A family of DNA polymerases, found across all domains of life, utilize various strategies for DNA strand separation during replication. In higher eukaryotes, mitochondrial DNA polymerase γ relies on the replicative helicase TWINKLE, whereas the yeast ortholog, Mip1, can unwind DNA independently. Using Mip1 as a model, we present a series of high-resolution cryo-EM structures that capture the process of DNA strand displacement. Our data reveal previously unidentified structural elements that facilitate the unwinding of the downstream DNA duplex. Yeast cells harboring Mip1 variants defective in strand displacement exhibit impaired oxidative phosphorylation and loss of mtDNA, corroborating the structural observations. This study provides a molecular basis for the intrinsic strand displacement activity of Mip1 and illuminates the distinct unwinding mechanisms utilized by Pol A family DNA polymerases. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_45196.map.gz emd_45196.map.gz | 134.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-45196-v30.xml emd-45196-v30.xml emd-45196.xml emd-45196.xml | 29.5 KB 29.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_45196_fsc.xml emd_45196_fsc.xml | 11.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_45196.png emd_45196.png | 115.4 KB | ||
| Filedesc metadata |  emd-45196.cif.gz emd-45196.cif.gz | 8.4 KB | ||
| その他 |  emd_45196_additional_1.map.gz emd_45196_additional_1.map.gz emd_45196_additional_2.map.gz emd_45196_additional_2.map.gz emd_45196_half_map_1.map.gz emd_45196_half_map_1.map.gz emd_45196_half_map_2.map.gz emd_45196_half_map_2.map.gz | 126 MB 72.7 MB 134.3 MB 134.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-45196 http://ftp.pdbj.org/pub/emdb/structures/EMD-45196 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-45196 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-45196 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_45196_validation.pdf.gz emd_45196_validation.pdf.gz | 1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_45196_full_validation.pdf.gz emd_45196_full_validation.pdf.gz | 1 MB | 表示 | |
| XML形式データ |  emd_45196_validation.xml.gz emd_45196_validation.xml.gz | 19.4 KB | 表示 | |
| CIF形式データ |  emd_45196_validation.cif.gz emd_45196_validation.cif.gz | 25 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-45196 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-45196 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-45196 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-45196 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_45196.map.gz / 形式: CCP4 / 大きさ: 144.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_45196.map.gz / 形式: CCP4 / 大きさ: 144.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Anisotropic sharpened map of the original map | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.7653 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: DeepEM sharpened map
| ファイル | emd_45196_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | DeepEM sharpened map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Original unsharpened map
| ファイル | emd_45196_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Original unsharpened map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half Map B
| ファイル | emd_45196_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half Map B | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half Map A
| ファイル | emd_45196_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half Map A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Cryo-EM map of the Strand displacement Complex (III) of Yeast Mit...
| 全体 | 名称: Cryo-EM map of the Strand displacement Complex (III) of Yeast Mitochondrial DNA polymerase Gamma (MIP1) |
|---|---|
| 要素 |
|
-超分子 #1: Cryo-EM map of the Strand displacement Complex (III) of Yeast Mit...
| 超分子 | 名称: Cryo-EM map of the Strand displacement Complex (III) of Yeast Mitochondrial DNA polymerase Gamma (MIP1) タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#4 詳細: Yeast Mitochondrial DNA polymerase Gamma (MIP1)wild-type assembled on a primer-template and a downstream non-template strand with incoming nucleotide, dATP bound to the polymerase site. |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 142 KDa |
-分子 #1: DNA polymerase gamma
| 分子 | 名称: DNA polymerase gamma / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO / EC番号: DNA-directed DNA polymerase |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 141.745438 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MASTKKNTAE APRINPVGIQ YLGESLQRQV FGSCGGKDEV EQSDKLMELS KKSLKDHGLW GKKTLITDPI SFPLPPLQGR SLDEHFQKI GRFNSEPYKS FCEDKFTEMV ARPAEWLRKP GWVKYVPGMA PVEVAYPDEE LVVFDVETLY NVSDYPTLAT A LSSTAWYL ...文字列: MASTKKNTAE APRINPVGIQ YLGESLQRQV FGSCGGKDEV EQSDKLMELS KKSLKDHGLW GKKTLITDPI SFPLPPLQGR SLDEHFQKI GRFNSEPYKS FCEDKFTEMV ARPAEWLRKP GWVKYVPGMA PVEVAYPDEE LVVFDVETLY NVSDYPTLAT A LSSTAWYL WCSPFICGGD DPAALIPLNT LNKEQVVIGH NVAYDRARVL EEYNFRDSKA FFLDTQSLHI ASFGLCSRQR PM FMKNNKK KEAEVESEVH PEISIEDYDD PWLNVSALNS LKDVAKFHCK IDLDKTDRDF FASTDKSTII ENFQKLVNYC ATD VTATSQ VFDKIFPVFL KKCPHPVSFA GLKSLSKCIL PTKLNDWNDY LNSSESLYQQ SKVQIESKIV QIIKDIALLK DKPD FYLKD PWLSQLDWTT KPLRLTKKGV PAKCQKLPGF PEWYRQLFPS KDTVEPKITI KSRIIPILFK LSWENSPVIW SKESG WCFN VPHEQVETYK AKNYVLADSV SQEEEEIRMN NLGLQCTGVL FKVPHPNGPT FNCTNLLTKS YNHFFEKGVL KSESEL AHQ ALQINSSGSY WMSARERIQS QFVVPNCKFP NEFQSLSAKS SLNNEKTNDL AIIIPKIVPM GTITRRAVEN TWLTASN AK ANRIGSELKT QVKAPPGYCF VGADVDSEEL WIASLVGDSI FNVHGGTAIG WMCLEGTKNE GTDLHTKTAQ ILGCSRNE A KIFNYGRIYG AGAKFASQLL KRFNPSLTDE ETKKIANKLY ENTKGKTKRS KLFKKFWYGG SESILFNKLE SIAEQETPK TPVLGCGITY SLMKKNLRAN SFLPSRINWA IQSSGVDYLH LLCCSMEYII KKYNLEARLC ISIHDEIRFL VSEKDKYRAA MALQISNIW TRAMFCQQMG INELPQNCAF FSQVDIDSVI RKEVNMDCIT PSNKTAIPHG EALDINQLLD KPNSKLGKPS L DIDSKVSQ YAYNYREPVF EEYNKSYTPE FLKYFLAMQV QSDKRDVNRL EDEYLRECTS KEYARDGNTA EYSLLDYIKD VE KGKRTKV RIMGSNFLDG TKNAKADQRI RLPVNMPDYP TLHKIANDSA IPEKQLLENR RKKENRIDDE NKKKLTRKKN TTP MERKYK RVYGGRKAFE AFYECANKPL DYTLETEKQF FNIPIDGVID DVLNDKSNYK KKPSQARTAS SSPIRKTAKA VHSK KLPAR KSSTTNRNLV ELERDITISR EYKLAAALEH HHHHH UniProtKB: UNIPROTKB: A0A8H4BW69 |
-分子 #2: Non-Template DNA
| 分子 | 名称: Non-Template DNA / タイプ: dna / ID: 2 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 7.078555 KDa |
| 配列 | 文字列: (DT)(DT)(DT)(DT)(DG)(DG)(DT)(DA)(DG)(DT) (DC)(DG)(DT)(DG)(DA)(DC)(DT)(DC)(DG)(DA) (DC)(DC)(DG) |
-分子 #3: Primer DNA
| 分子 | 名称: Primer DNA / タイプ: dna / ID: 3 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 7.17261 KDa |
| 配列 | 文字列: (DG)(DA)(DA)(DG)(DA)(DC)(DA)(DG)(DT)(DC) (DT)(DG)(DC)(DG)(DG)(DC)(DG)(DC)(DG)(DC) (DG)(DG)(DG) |
-分子 #4: Template DNA
| 分子 | 名称: Template DNA / タイプ: dna / ID: 4 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 12.854218 KDa |
| 配列 | 文字列: (DC)(DG)(DG)(DT)(DC)(DG)(DA)(DG)(DA)(DG) (DT)(DC)(DA)(DC)(DG)(DA)(DC)(DT)(DA)(DC) (DC)(DC)(DG)(DC)(DG)(DC)(DG)(DC)(DC) (DG)(DC)(DA)(DG)(DA)(DC)(DT)(DG)(DT)(DC) (DT) (DT)(DC) |
-分子 #5: 2'-DEOXYADENOSINE 5'-TRIPHOSPHATE
| 分子 | 名称: 2'-DEOXYADENOSINE 5'-TRIPHOSPHATE / タイプ: ligand / ID: 5 / コピー数: 1 / 式: DTP |
|---|---|
| 分子量 | 理論値: 491.182 Da |
| Chemical component information |  ChemComp-DTP: |
-分子 #6: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 6 / コピー数: 1 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-分子 #7: 3'-DEOXYTHYMIDINE-5'-MONOPHOSPHATE
| 分子 | 名称: 3'-DEOXYTHYMIDINE-5'-MONOPHOSPHATE / タイプ: ligand / ID: 7 / コピー数: 1 / 式: 2DT |
|---|---|
| 分子量 | 理論値: 306.209 Da |
| Chemical component information | 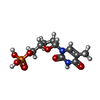 ChemComp-2DT: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.57 mg/mL |
|---|---|
| 緩衝液 | pH: 7.9 / 詳細: 10 mM Tris-HCL, 100 mM NaCL, 10 mM DTT, 5 mM MgCl2 |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
| 詳細 | A 4 uM strand displacement complex complex was assembled with wild-type MIP1 and a DNA scaffold with primer, template, and non-template strands, mixed in equal millimolar ratio. The primer strand was extended by three nucleotides with 1 mM dGTP and chain terminated with 1 mM di-deoxy TTP. 1 mM dATP was added before plunge freezing. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - スリット幅: 10 eV |
| ソフトウェア | 名称: EPU (ver. 3.7) |
| 撮影 | フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) 撮影したグリッド数: 2 / 実像数: 14819 / 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 1.5 µm / 最小 デフォーカス(公称値): 0.5 µm / 倍率(公称値): 105000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | PDB ID: Chain - Residue range: 1-1254 / Chain - Source name: AlphaFold / Chain - Initial model type: in silico model |
|---|---|
| ソフトウェア | 名称:  Coot (ver. 0.9.8.5) Coot (ver. 0.9.8.5) |
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT |
| 得られたモデル |  PDB-9c53: |
 ムービー
ムービー コントローラー
コントローラー










 X (Sec.)
X (Sec.) Y (Row.)
Y (Row.) Z (Col.)
Z (Col.)






















































