+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | INF2 at the Barbed End of F-Actin with Incoming Actin | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Actin / Filament / Elongation / Ends / CYTOSOLIC PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報cytoskeletal motor activator activity / myosin heavy chain binding / tropomyosin binding / regulation of mitochondrial fission / actin filament bundle / troponin I binding / filamentous actin / mesenchyme migration / skeletal muscle myofibril / actin filament bundle assembly ...cytoskeletal motor activator activity / myosin heavy chain binding / tropomyosin binding / regulation of mitochondrial fission / actin filament bundle / troponin I binding / filamentous actin / mesenchyme migration / skeletal muscle myofibril / actin filament bundle assembly / striated muscle thin filament / skeletal muscle thin filament assembly / actin monomer binding / skeletal muscle fiber development / stress fiber / titin binding / actin filament polymerization / filopodium / actin filament / 加水分解酵素; 酸無水物に作用; 酸無水物に作用・細胞または細胞小器官の運動に関与 / small GTPase binding / calcium-dependent protein binding / lamellipodium / actin binding / cell body / hydrolase activity / protein domain specific binding / calcium ion binding / positive regulation of gene expression / perinuclear region of cytoplasm / magnesium ion binding / ATP binding / identical protein binding / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) / Homo sapiens (ヒト) /  | |||||||||
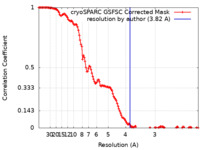
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.82 Å | |||||||||
 データ登録者 データ登録者 | Palmer NJ / Barrie KR / Dominguez R | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2024 ジャーナル: Nature / 年: 2024タイトル: Mechanisms of actin filament severing and elongation by formins. 著者: Nicholas J Palmer / Kyle R Barrie / Roberto Dominguez /  要旨: Humans express 15 formins that play crucial roles in actin-based processes, including cytokinesis, cell motility and mechanotransduction. However, the lack of structures bound to the actin filament ...Humans express 15 formins that play crucial roles in actin-based processes, including cytokinesis, cell motility and mechanotransduction. However, the lack of structures bound to the actin filament (F-actin) has been a major impediment to understanding formin function. Whereas formins are known for their ability to nucleate and elongate F-actin, some formins can additionally depolymerize, sever or bundle F-actin. Two mammalian formins, inverted formin 2 (INF2) and diaphanous 1 (DIA1, encoded by DIAPH1), exemplify this diversity. INF2 shows potent severing activity but elongates weakly whereas DIA1 has potent elongation activity but does not sever. Using cryo-electron microscopy (cryo-EM) we show five structural states of INF2 and two of DIA1 bound to the middle and barbed end of F-actin. INF2 and DIA1 bind differently to these sites, consistent with their distinct activities. The formin-homology 2 and Wiskott-Aldrich syndrome protein-homology 2 (FH2 and WH2, respectively) domains of INF2 are positioned to sever F-actin, whereas DIA1 appears unsuited for severing. These structures also show how profilin-actin is delivered to the fast-growing barbed end, and how this is followed by a transition of the incoming monomer into the F-actin conformation and the release of profilin. Combined, the seven structures presented here provide step-by-step visualization of the mechanisms of F-actin severing and elongation by formins. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_44019.map.gz emd_44019.map.gz | 107.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-44019-v30.xml emd-44019-v30.xml emd-44019.xml emd-44019.xml | 21.3 KB 21.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_44019_fsc.xml emd_44019_fsc.xml | 12.8 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_44019.png emd_44019.png | 20.6 KB | ||
| マスクデータ |  emd_44019_msk_1.map emd_44019_msk_1.map | 216 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-44019.cif.gz emd-44019.cif.gz | 7 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-44019 http://ftp.pdbj.org/pub/emdb/structures/EMD-44019 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-44019 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-44019 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_44019_validation.pdf.gz emd_44019_validation.pdf.gz | 562.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_44019_full_validation.pdf.gz emd_44019_full_validation.pdf.gz | 562.3 KB | 表示 | |
| XML形式データ |  emd_44019_validation.xml.gz emd_44019_validation.xml.gz | 13.8 KB | 表示 | |
| CIF形式データ |  emd_44019_validation.cif.gz emd_44019_validation.cif.gz | 18.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-44019 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-44019 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-44019 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-44019 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  9azqMC  9az4C  9azpC  9b03C  9b0kC  9b27C  9b3dC C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_44019.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_44019.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.08 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_44019_msk_1.map emd_44019_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Actin
| 全体 | 名称: Actin |
|---|---|
| 要素 |
|
-超分子 #1: Actin
| 超分子 | 名称: Actin / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#3 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 264 KDa |
-超分子 #2: INF2 Dimer
| 超分子 | 名称: INF2 Dimer / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #2-#3 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-超分子 #3: Actin Filament
| 超分子 | 名称: Actin Filament / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Actin, alpha skeletal muscle
| 分子 | 名称: Actin, alpha skeletal muscle / タイプ: protein_or_peptide / ID: 1 / コピー数: 7 / 光学異性体: LEVO EC番号: 加水分解酵素; 酸無水物に作用; 酸無水物に作用・細胞または細胞小器官の運動に関与 |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 41.387227 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: TTALVCDNGS GLVKAGFAGD DAPRAVFPSI VGRPRHQGVM VGMGQKDSYV GDEAQSKRGI LTLKYPIE(HIC)G IITNWD DME KIWHHTFYNE LRVAPEEHPT LLTEAPLNPK ANREKMTQIM FETFNVPAMY VAIQAVLSLY ASGRTTGIVL DSGDGVT HN ...文字列: TTALVCDNGS GLVKAGFAGD DAPRAVFPSI VGRPRHQGVM VGMGQKDSYV GDEAQSKRGI LTLKYPIE(HIC)G IITNWD DME KIWHHTFYNE LRVAPEEHPT LLTEAPLNPK ANREKMTQIM FETFNVPAMY VAIQAVLSLY ASGRTTGIVL DSGDGVT HN VPIYEGYALP HAIMRLDLAG RDLTDYLMKI LTERGYSFVT TAEREIVRDI KEKLCYVALD FENEMATAAS SSSLEKSY E LPDGQVITIG NERFRCPETL FQPSFIGMES AGIHETTYNS IMKCDIDIRK DLYANNVMSG GTTMYPGIAD RMQKEITAL APSTMKIKII APPERKYSVW IGGSILASLS TFQQMWITKQ EYDEAGPSIV HRKCF UniProtKB: Actin, alpha skeletal muscle |
-分子 #2: Inverted formin-2
| 分子 | 名称: Inverted formin-2 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 6.870784 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: VPSHRRVNPP TLRMKKLNWQ KLPSNVAREH NSMWASLSSP DAEAVEPDFS SIERLFSFPA UniProtKB: Inverted formin-2 |
-分子 #3: Inverted formin-2
| 分子 | 名称: Inverted formin-2 / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 36.856391 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: KEPKEITFLD AKKSLNLNIF LKQFKCSNEE VAAMIRAGDT TKFDVEVLKQ LLKLLPEKHE IENLRAFTEE RAKLASADHF YLLLLAIPC YQLRIECMLL CEGAAAVLDM VRPKAQLVLA ACESLLTSRQ LPIFCQLILR IGNFLNYGSH TGDADGFKIS T LLKLTETK ...文字列: KEPKEITFLD AKKSLNLNIF LKQFKCSNEE VAAMIRAGDT TKFDVEVLKQ LLKLLPEKHE IENLRAFTEE RAKLASADHF YLLLLAIPC YQLRIECMLL CEGAAAVLDM VRPKAQLVLA ACESLLTSRQ LPIFCQLILR IGNFLNYGSH TGDADGFKIS T LLKLTETK SQQNRVTLLH HVLEEAEKSH PDLLQLPRDL EQPSQAAGIN LEIIRSEASS NLKKLLETER KVSASVAEVQ EQ YTERLQA SISAFRALDE LFEAIEQKQR ELADYLCEDA QQLSLEDTFS TMKAFRDLFL RALKENKDRK EQAAKAERRK QQL AEE UniProtKB: Inverted formin-2 |
-分子 #4: ADENOSINE-5'-DIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-DIPHOSPHATE / タイプ: ligand / ID: 4 / コピー数: 7 / 式: ADP |
|---|---|
| 分子量 | 理論値: 427.201 Da |
| Chemical component information |  ChemComp-ADP: |
-分子 #5: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 5 / コピー数: 7 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1.45 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
| ||||||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 / 支持フィルム - Film thickness: 100 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 120 sec. | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 実像数: 52360 / 平均電子線量: 44.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: SPOT SCAN / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 0.5 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー


































 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)