+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Human OGG1 bound to a 35-bp DNA with an 8-oxoG in the middle | |||||||||
 マップデータ マップデータ | Human OGG1 binding at 8-oxoG on a 35 bp DNA half map 1 | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Human OGG1 binding at 8-oxoG of a 35 bp DNA duplex / HYDROLASE / LYASE-DNA complex | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Defective OGG1 Substrate Binding / Defective OGG1 Substrate Processing / Defective OGG1 Localization / depurination / negative regulation of double-strand break repair via single-strand annealing / oxidized purine nucleobase lesion DNA N-glycosylase activity / base-excision repair, AP site formation / depyrimidination / 8-oxo-7,8-dihydroguanine DNA N-glycosylase activity / Displacement of DNA glycosylase by APEX1 ...Defective OGG1 Substrate Binding / Defective OGG1 Substrate Processing / Defective OGG1 Localization / depurination / negative regulation of double-strand break repair via single-strand annealing / oxidized purine nucleobase lesion DNA N-glycosylase activity / base-excision repair, AP site formation / depyrimidination / 8-oxo-7,8-dihydroguanine DNA N-glycosylase activity / Displacement of DNA glycosylase by APEX1 / positive regulation of gene expression via chromosomal CpG island demethylation / oxidized purine DNA binding / 加水分解酵素; 糖加水分解酵素; N-グリコシル化合物加水分解酵素 / APEX1-Independent Resolution of AP Sites via the Single Nucleotide Replacement Pathway / Recognition and association of DNA glycosylase with site containing an affected purine / Cleavage of the damaged purine / Recognition and association of DNA glycosylase with site containing an affected pyrimidine / Cleavage of the damaged pyrimidine / class I DNA-(apurinic or apyrimidinic site) endonuclease activity / DNA-(apurinic or apyrimidinic site) lyase / cellular response to reactive oxygen species / nucleotide-excision repair / response to radiation / base-excision repair / nuclear matrix / response to oxidative stress / endonuclease activity / microtubule binding / damaged DNA binding / nuclear speck / RNA polymerase II cis-regulatory region sequence-specific DNA binding / mitochondrial matrix / DNA damage response / regulation of DNA-templated transcription / enzyme binding / positive regulation of transcription by RNA polymerase II / protein-containing complex / mitochondrion / DNA binding / nucleoplasm / nucleus / cytosol 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) / synthetic construct (人工物) Homo sapiens (ヒト) / synthetic construct (人工物) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.7 Å | |||||||||
 データ登録者 データ登録者 | You Q / Li H | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Commun Biol / 年: 2024 ジャーナル: Commun Biol / 年: 2024タイトル: Human 8-oxoguanine glycosylase OGG1 binds nucleosome at the dsDNA ends and the super-helical locations. 著者: Qinglong You / Xiang Feng / Yi Cai / Stephen B Baylin / Huilin Li /  要旨: The human glycosylase OGG1 extrudes and excises the oxidized DNA base 8-oxoguanine (8-oxoG) to initiate base excision repair and plays important roles in many pathological conditions such as cancer, ...The human glycosylase OGG1 extrudes and excises the oxidized DNA base 8-oxoguanine (8-oxoG) to initiate base excision repair and plays important roles in many pathological conditions such as cancer, inflammation, and neurodegenerative diseases. Previous structural studies have used a truncated protein and short linear DNA, so it has been unclear how full-length OGG1 operates on longer DNA or on nucleosomes. Here we report cryo-EM structures of human OGG1 bound to a 35-bp long DNA containing an 8-oxoG within an unmethylated Cp-8-oxoG dinucleotide as well as to a nucleosome with an 8-oxoG at super-helical location (SHL)-5. The 8-oxoG in the linear DNA is flipped out by OGG1, consistent with previous crystallographic findings with a 15-bp DNA. OGG1 preferentially binds near dsDNA ends at the nucleosomal entry/exit sites. Such preference may underlie the enzyme's function in DNA double-strand break repair. Unexpectedly, we find that OGG1 bends the nucleosomal entry DNA, flips an undamaged guanine, and binds to internal nucleosomal DNA sites such as SHL-5 and SHL+6. We suggest that the DNA base search mechanism by OGG1 may be chromatin context-dependent and that OGG1 may partner with chromatin remodelers to excise 8-oxoG at the nucleosomal internal sites. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_43607.map.gz emd_43607.map.gz | 3.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-43607-v30.xml emd-43607-v30.xml emd-43607.xml emd-43607.xml | 18.6 KB 18.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_43607.png emd_43607.png | 67.6 KB | ||
| Filedesc metadata |  emd-43607.cif.gz emd-43607.cif.gz | 6.3 KB | ||
| その他 |  emd_43607_half_map_1.map.gz emd_43607_half_map_1.map.gz emd_43607_half_map_2.map.gz emd_43607_half_map_2.map.gz | 23.1 MB 23.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-43607 http://ftp.pdbj.org/pub/emdb/structures/EMD-43607 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-43607 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-43607 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_43607_validation.pdf.gz emd_43607_validation.pdf.gz | 547.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_43607_full_validation.pdf.gz emd_43607_full_validation.pdf.gz | 547.1 KB | 表示 | |
| XML形式データ |  emd_43607_validation.xml.gz emd_43607_validation.xml.gz | 10.5 KB | 表示 | |
| CIF形式データ |  emd_43607_validation.cif.gz emd_43607_validation.cif.gz | 12.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-43607 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-43607 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-43607 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-43607 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8vx4MC  8vx5C  8vx6C M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_43607.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_43607.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Human OGG1 binding at 8-oxoG on a 35 bp DNA half map 1 | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.828 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: Human OGG1 binding at 8-oxoG on a 35 bp DNA
| ファイル | emd_43607_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Human OGG1 binding at 8-oxoG on a 35 bp DNA | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
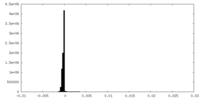
| 密度ヒストグラム |
-ハーフマップ: Human OGG1 binding at 8-oxoG on a 35 bp DNA half map 2
| ファイル | emd_43607_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Human OGG1 binding at 8-oxoG on a 35 bp DNA half map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
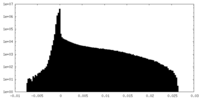
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Complex of Human OGG1 with a 35 bp DNA duplex containing 8-oxoG
| 全体 | 名称: Complex of Human OGG1 with a 35 bp DNA duplex containing 8-oxoG |
|---|---|
| 要素 |
|
-超分子 #1: Complex of Human OGG1 with a 35 bp DNA duplex containing 8-oxoG
| 超分子 | 名称: Complex of Human OGG1 with a 35 bp DNA duplex containing 8-oxoG タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: DNA (35-MER)
| 分子 | 名称: DNA (35-MER) / タイプ: dna / ID: 1 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 10.695851 KDa |
| 配列 | 文字列: (DA)(DT)(DG)(DC)(DC)(DT)(DC)(DG)(DC)(DA) (DG)(DA)(DA)(DT)(DC)(DC)(DC)(8OG)(DC) (DT)(DG)(DC)(DC)(DG)(DA)(DG)(DG)(DC)(DC) (DG)(DC)(DT)(DC)(DA)(DA) |
-分子 #2: DNA (35-MER)
| 分子 | 名称: DNA (35-MER) / タイプ: dna / ID: 2 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 10.861943 KDa |
| 配列 | 文字列: (DT)(DT)(DG)(DA)(DG)(DC)(DG)(DG)(DC)(DC) (DT)(DC)(DG)(DG)(DC)(DA)(DG)(DC)(DG)(DG) (DG)(DA)(DT)(DT)(DC)(DT)(DG)(DC)(DG) (DA)(DG)(DG)(DC)(DA)(DT) |
-分子 #3: N-glycosylase/DNA lyase
| 分子 | 名称: N-glycosylase/DNA lyase / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO EC番号: 加水分解酵素; 糖加水分解酵素; N-グリコシル化合物加水分解酵素 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 43.72002 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MGHHHHHHDY KDHDGDYKDH DIDYKDDDDK ENLYFQGGGG GSDPARALLP RRMGHRTLAS TPALWASIPC PRSELRLDLV LPSGQSFRW REQSPAHWSG VLADQVWTLT QTEEQLHCTV YRGDKSQASR PTPDELEAVR KYFQLDVTLA QLYHHWGSVD S HFQEVAQK ...文字列: MGHHHHHHDY KDHDGDYKDH DIDYKDDDDK ENLYFQGGGG GSDPARALLP RRMGHRTLAS TPALWASIPC PRSELRLDLV LPSGQSFRW REQSPAHWSG VLADQVWTLT QTEEQLHCTV YRGDKSQASR PTPDELEAVR KYFQLDVTLA QLYHHWGSVD S HFQEVAQK FQGVRLLRQD PIECLFSFIC SSNNNIARIT GMVERLCQAF GPRLIQLDDV TYHGFPSLQA LAGPEVEAHL RK LGLGYRA RYVSASARAI LEEQGGLAWL QQLRESSYEE AHKALCILPG VGTQVADCIC LMALDKPQAV PVDVHMWHIA QRD YSWHPT TSQAKGPSPQ TNKELGNFFR SLWGPYAGWA QAVLFSADLR QSRHAQEPPA KRRKGSKGPE G UniProtKB: N-glycosylase/DNA lyase |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 64.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: AB INITIO MODEL |
|---|---|
| 得られたモデル |  PDB-8vx4: |
 ムービー
ムービー コントローラー
コントローラー











 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)