+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | AaegOR10 structure bound to o-cresol | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Mosquito olfactory receptor OR10 / MEMBRANE PROTEIN | |||||||||
| 機能・相同性 | Olfactory receptor, insect / 7tm Odorant receptor / olfactory receptor activity / odorant binding / signal transduction / identical protein binding / plasma membrane / Odorant receptor / Odorant receptor 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |   Apocrypta bakeri (昆虫) Apocrypta bakeri (昆虫) | |||||||||
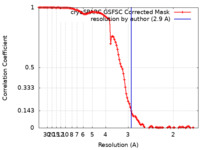
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.9 Å | |||||||||
 データ登録者 データ登録者 | Zhao J / del Marmol J | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Science / 年: 2024 ジャーナル: Science / 年: 2024タイトル: Structural basis of odor sensing by insect heteromeric odorant receptors. 著者: Jiawei Zhao / Andy Q Chen / Jaewook Ryu / Josefina Del Mármol /  要旨: Most insects, including human-targeting mosquitoes, detect odors through odorant-activated ion channel complexes consisting of a divergent odorant-binding subunit (OR) and a conserved co-receptor ...Most insects, including human-targeting mosquitoes, detect odors through odorant-activated ion channel complexes consisting of a divergent odorant-binding subunit (OR) and a conserved co-receptor subunit (Orco). As a basis for understanding how odorants activate these heteromeric receptors, we report here cryo-electron microscopy structures of two different heteromeric odorant receptor complexes containing ORs from disease-vector mosquitos or . These structures reveal an unexpected stoichiometry of one OR to three Orco subunits. Comparison of structures in odorant-bound and unbound states indicates that odorant binding to the sole OR subunit is sufficient to open the channel pore, suggesting a mechanism of OR activation and a conceptual framework for understanding evolution of insect odorant receptor sensitivity. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_42850.map.gz emd_42850.map.gz | 129.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-42850-v30.xml emd-42850-v30.xml emd-42850.xml emd-42850.xml | 16.3 KB 16.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_42850_fsc.xml emd_42850_fsc.xml | 10.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_42850.png emd_42850.png | 170.4 KB | ||
| Filedesc metadata |  emd-42850.cif.gz emd-42850.cif.gz | 5.9 KB | ||
| その他 |  emd_42850_half_map_1.map.gz emd_42850_half_map_1.map.gz emd_42850_half_map_2.map.gz emd_42850_half_map_2.map.gz | 127.2 MB 127.2 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-42850 http://ftp.pdbj.org/pub/emdb/structures/EMD-42850 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-42850 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-42850 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_42850_validation.pdf.gz emd_42850_validation.pdf.gz | 1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_42850_full_validation.pdf.gz emd_42850_full_validation.pdf.gz | 1 MB | 表示 | |
| XML形式データ |  emd_42850_validation.xml.gz emd_42850_validation.xml.gz | 19.5 KB | 表示 | |
| CIF形式データ |  emd_42850_validation.cif.gz emd_42850_validation.cif.gz | 25.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-42850 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-42850 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-42850 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-42850 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_42850.map.gz / 形式: CCP4 / 大きさ: 137.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_42850.map.gz / 形式: CCP4 / 大きさ: 137.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.83 Å | ||||||||||||||||||||||||||||||||||||
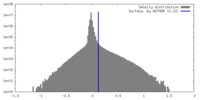
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_42850_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
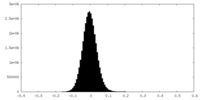
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_42850_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
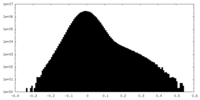
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : AaegOR10 heterotetramer bound to o-cresol
| 全体 | 名称: AaegOR10 heterotetramer bound to o-cresol |
|---|---|
| 要素 |
|
-超分子 #1: AaegOR10 heterotetramer bound to o-cresol
| 超分子 | 名称: AaegOR10 heterotetramer bound to o-cresol / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 |
|---|
-超分子 #2: Aedes aegypti OR10
| 超分子 | 名称: Aedes aegypti OR10 / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  |
-超分子 #3: Apocrypta bakeri Orco
| 超分子 | 名称: Apocrypta bakeri Orco / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #2 |
|---|---|
| 由来(天然) | 生物種:  Apocrypta bakeri (昆虫) Apocrypta bakeri (昆虫) |
-分子 #1: Odorant receptor OR10
| 分子 | 名称: Odorant receptor OR10 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 43.491168 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MASILDCPIV SVNARVWRFW SFVLKHDAMR YISIIPVTVM TFFMFTDLCR SWGNIQELII KAYFAVLYFN AVLRTLILVK DRKLYENFM QGISNVYFEI SHIDDHKIQS LLKSYTVRAR MLSISNLALG AIISTCFVVY PIFTGERGLP YGMFIPGLDS F RSPHYEII ...文字列: MASILDCPIV SVNARVWRFW SFVLKHDAMR YISIIPVTVM TFFMFTDLCR SWGNIQELII KAYFAVLYFN AVLRTLILVK DRKLYENFM QGISNVYFEI SHIDDHKIQS LLKSYTVRAR MLSISNLALG AIISTCFVVY PIFTGERGLP YGMFIPGLDS F RSPHYEII YIVQVVLTFP GCCMYIPFTS FFASTTLFGL VQIKTLQRQL QTFKDNINSQ DKEKVKAKVV KLIEDHKRII TY VSELNSL VTYICFVEFL SFGMMLCALL FLLNVIENHA QIVIVAAYIF MIISQIFAFY WHANEVREES MNLAEAAYSG PWV ELDNSI KKKLLLIILR AQQPLEITVG NVYPMTLEMF QSLLNASYSY FTLLRRVYN UniProtKB: Odorant receptor |
-分子 #2: Odorant receptor Orco
| 分子 | 名称: Odorant receptor Orco / タイプ: protein_or_peptide / ID: 2 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Apocrypta bakeri (昆虫) Apocrypta bakeri (昆虫) |
| 分子量 | 理論値: 53.204953 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MKFKHQGLVA DLLPNIRVMQ GVGHFMFNYY SEGKKFPHRI YCIVTLLLLL LQYGMMAVNL MMESDDVDDL TANTITMLFF LHPIVKMIY FPVRSKIFYK TLAIWNNPNS HPLFAESNAR FHALAITKMR RLLFCVAGAT IFSVISWTGI TFIEDSVKRI T DPETNETT ...文字列: MKFKHQGLVA DLLPNIRVMQ GVGHFMFNYY SEGKKFPHRI YCIVTLLLLL LQYGMMAVNL MMESDDVDDL TANTITMLFF LHPIVKMIY FPVRSKIFYK TLAIWNNPNS HPLFAESNAR FHALAITKMR RLLFCVAGAT IFSVISWTGI TFIEDSVKRI T DPETNETT IIPIPRLMIR TFYPFNAMSG AGHVFALIYQ FYYLVISMAV SNSLDVLFCS WLLFACEQLQ HLKAIMKPLM EL SATLDTV VPNSGELFKA GSADHLRESQ GVQPSGNGDN VLDVDLRGIY SNRQDFTATF RPTAGTTFNG GVGPNGLTKK QEM LVRSAI KYWVERHKHV VRLVTAVGDA YGVALLLHML TTTITLTLLA YQATKVNGVN VYAATVIGYL LYTLGQVFLF CIFG NRLIE ESSSVMEAAY SCHWYDGSEE AKTFVQIVCQ QCQKAMSISG AKFFTVSLDL FASVLGAVVT YFMVLVQLK UniProtKB: Odorant receptor |
-分子 #3: o-cresol
| 分子 | 名称: o-cresol / タイプ: ligand / ID: 3 / コピー数: 1 / 式: JZ0 |
|---|---|
| 分子量 | 理論値: 108.138 Da |
| Chemical component information | 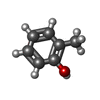 ChemComp-JZ0: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | #0 - Image recording ID: 1 #0 - フィルム・検出器のモデル: GATAN K3 (6k x 4k) #0 - 平均電子線量: 55.2 e/Å2 / #1 - Image recording ID: 2 #1 - フィルム・検出器のモデル: GATAN K3 (6k x 4k) #1 - 平均電子線量: 58.2 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー











 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)