+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of Heterochromatin Protein 1 (HP1) alpha in complex with an H2A.Z nucleosome | |||||||||
 マップデータ マップデータ | primary map, consensus refinement with mask | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | heterochromatin / nucleosome / chromatin / GENE REGULATION / STRUCTURAL PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報chromocenter / histone H3K9me2/3 reader activity / nucleosomal DNA binding / Transcriptional Regulation by E2F6 / : / histone methyltransferase complex / site of DNA damage / histone deacetylase complex / ribonucleoprotein complex binding / pericentric heterochromatin ...chromocenter / histone H3K9me2/3 reader activity / nucleosomal DNA binding / Transcriptional Regulation by E2F6 / : / histone methyltransferase complex / site of DNA damage / histone deacetylase complex / ribonucleoprotein complex binding / pericentric heterochromatin / RNA polymerase II core promoter sequence-specific DNA binding / heterochromatin / transcription repressor complex / SUMOylation of chromatin organization proteins / Regulation of endogenous retroelements by KRAB-ZFP proteins / cellular response to estradiol stimulus / euchromatin / kinetochore / histone deacetylase binding / structural constituent of chromatin / nuclear envelope / nucleosome / heterochromatin formation / nucleosome assembly / Factors involved in megakaryocyte development and platelet production / chromatin organization / protein-macromolecule adaptor activity / DNA-binding transcription factor binding / chromosome, telomeric region / RNA polymerase II cis-regulatory region sequence-specific DNA binding / protein heterodimerization activity / ribonucleoprotein complex / negative regulation of DNA-templated transcription / DNA damage response / chromatin binding / protein-containing complex binding / nucleolus / negative regulation of transcription by RNA polymerase II / positive regulation of transcription by RNA polymerase II / protein-containing complex / DNA binding / nucleoplasm / identical protein binding / nucleus 類似検索 - 分子機能 | |||||||||
| 生物種 |   Homo sapiens (ヒト) / Homo sapiens (ヒト) /  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 6.3 Å | |||||||||
 データ登録者 データ登録者 | Tan D / Sokolova V | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Structure / 年: 2024 ジャーナル: Structure / 年: 2024タイトル: Structural mechanism of HP1⍺-dependent transcriptional repression and chromatin compaction. 著者: Vladyslava Sokolova / Jacob Miratsky / Vladimir Svetlov / Michael Brenowitz / John Vant / Tyler S Lewis / Kelly Dryden / Gahyun Lee / Shayan Sarkar / Evgeny Nudler / Abhishek Singharoy / Dongyan Tan /  要旨: Heterochromatin protein 1 (HP1) plays a central role in establishing and maintaining constitutive heterochromatin. However, the mechanisms underlying HP1-nucleosome interactions and their ...Heterochromatin protein 1 (HP1) plays a central role in establishing and maintaining constitutive heterochromatin. However, the mechanisms underlying HP1-nucleosome interactions and their contributions to heterochromatin functions remain elusive. Here, we present the cryoelectron microscopy (cryo-EM) structure of an HP1α dimer bound to an H2A.Z-nucleosome, revealing two distinct HP1α-nucleosome interfaces. The primary HP1α binding site is located at the N terminus of histone H3, specifically at the trimethylated lysine 9 (K9me3) region, while a secondary binding site is situated near histone H2B, close to nucleosome superhelical location 4 (SHL4). Our biochemical data further demonstrates that HP1α binding influences the dynamics of DNA on the nucleosome. It promotes DNA unwrapping near the nucleosome entry and exit sites while concurrently restricting DNA accessibility in the vicinity of SHL4. Our study offers a model for HP1α-mediated heterochromatin maintenance and gene silencing. It also sheds light on the H3K9me-independent role of HP1 in responding to DNA damage. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_42774.map.gz emd_42774.map.gz | 49.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-42774-v30.xml emd-42774-v30.xml emd-42774.xml emd-42774.xml | 27.1 KB 27.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_42774.png emd_42774.png | 55.2 KB | ||
| Filedesc metadata |  emd-42774.cif.gz emd-42774.cif.gz | 7.6 KB | ||
| その他 |  emd_42774_half_map_1.map.gz emd_42774_half_map_1.map.gz emd_42774_half_map_2.map.gz emd_42774_half_map_2.map.gz | 49.8 MB 49.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-42774 http://ftp.pdbj.org/pub/emdb/structures/EMD-42774 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-42774 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-42774 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_42774_validation.pdf.gz emd_42774_validation.pdf.gz | 846.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_42774_full_validation.pdf.gz emd_42774_full_validation.pdf.gz | 846.2 KB | 表示 | |
| XML形式データ |  emd_42774_validation.xml.gz emd_42774_validation.xml.gz | 12 KB | 表示 | |
| CIF形式データ |  emd_42774_validation.cif.gz emd_42774_validation.cif.gz | 14.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-42774 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-42774 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-42774 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-42774 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_42774.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_42774.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | primary map, consensus refinement with mask | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.08 Å | ||||||||||||||||||||||||||||||||||||
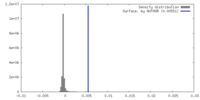
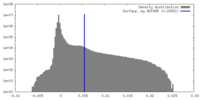
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : HP1alpha dimer bound to H2A.Z nucleosome
| 全体 | 名称: HP1alpha dimer bound to H2A.Z nucleosome |
|---|---|
| 要素 |
|
-超分子 #1: HP1alpha dimer bound to H2A.Z nucleosome
| 超分子 | 名称: HP1alpha dimer bound to H2A.Z nucleosome / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 288 KDa |
-分子 #1: Chromobox protein homolog 5
| 分子 | 名称: Chromobox protein homolog 5 / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 22.651398 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: GSPEFGKKTK RTADSSSSED EEEYVVEKVL DRRVVKGQVE YLLKWKGFSE EHNTWEPEKN LDCPELISEF MKKYKKMKEG ENNKPREKS ESNKRKSNFS NSADDIKSKK KREQSNDIAR GFERGLEPEK IIGATDSCGD LMFLMKWKDT DEADLVLAKE A NVKCPQIV ...文字列: GSPEFGKKTK RTADSSSSED EEEYVVEKVL DRRVVKGQVE YLLKWKGFSE EHNTWEPEKN LDCPELISEF MKKYKKMKEG ENNKPREKS ESNKRKSNFS NSADDIKSKK KREQSNDIAR GFERGLEPEK IIGATDSCGD LMFLMKWKDT DEADLVLAKE A NVKCPQIV IAFYEERLTW HAYPEDAENK EKETAKS UniProtKB: Chromobox protein homolog 5 |
-分子 #2: Histone H3.2
| 分子 | 名称: Histone H3.2 / タイプ: protein_or_peptide / ID: 2 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: |
| 分子量 | 理論値: 15.30393 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: ARTKQTARKS TGGKAPRKQL ATKAARKSAP ATGGVKKPHR YRPGTVALRE IRRYQKSTEL LIRKLPFQRL VREIAQDFKT DLRFQSSAV MALQEASEAY LVALFEDTNL CAIHAKRVTI MPKDIQLARR IRGERA UniProtKB: Histone H3.2 |
-分子 #3: Histone H4
| 分子 | 名称: Histone H4 / タイプ: protein_or_peptide / ID: 3 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: |
| 分子量 | 理論値: 11.263231 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: SGRGKGGKGL GKGGAKRHRK VLRDNIQGIT KPAIRRLARR GGVKRISGLI YEETRGVLKV FLENVIRDAV TYTEHAKRKT VTAMDVVYA LKRQGRTLYG FGG UniProtKB: Histone H4 |
-分子 #4: Histone H2A.Z
| 分子 | 名称: Histone H2A.Z / タイプ: protein_or_peptide / ID: 4 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 13.450601 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: AGGKAGKDSG KAKTKAVSRS QRAGLQFPVG RIHRHLKSRT TSHGRVGATA AVYSAAILEY LTAEVLELAG NASKDLKVKR ITPRHLQLA IRGDEELDSL IKATIAGGGV IPHIHKSLIG KKGQQKTV UniProtKB: Histone H2A.Z |
-分子 #5: Histone H2B 1.1
| 分子 | 名称: Histone H2B 1.1 / タイプ: protein_or_peptide / ID: 5 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: |
| 分子量 | 理論値: 13.848097 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: PEPAKSAPAP KKGSKKAVTK TQKKDGKKRR KTRKESYAIY VYKVLKQVHP DTGISSKAMS IMNSFVNDVF ERIAGEASRL AHYNKRSTI TSREIQTAVR LLLPGELAKH AVSEGTKAVT KYTSAK UniProtKB: Histone H2B 1.1 |
-分子 #6: DNA Widom601 (208bp) strand1
| 分子 | 名称: DNA Widom601 (208bp) strand1 / タイプ: dna / ID: 6 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 64.456039 KDa |
| 配列 | 文字列: (DA)(DC)(DT)(DC)(DC)(DG)(DG)(DC)(DA)(DA) (DG)(DG)(DT)(DC)(DG)(DC)(DT)(DG)(DT)(DT) (DC)(DA)(DA)(DT)(DA)(DC)(DA)(DT)(DG) (DC)(DA)(DC)(DA)(DG)(DG)(DA)(DT)(DG)(DT) (DA) (DT)(DA)(DT)(DA)(DT) ...文字列: (DA)(DC)(DT)(DC)(DC)(DG)(DG)(DC)(DA)(DA) (DG)(DG)(DT)(DC)(DG)(DC)(DT)(DG)(DT)(DT) (DC)(DA)(DA)(DT)(DA)(DC)(DA)(DT)(DG) (DC)(DA)(DC)(DA)(DG)(DG)(DA)(DT)(DG)(DT) (DA) (DT)(DA)(DT)(DA)(DT)(DC)(DT)(DG) (DA)(DC)(DA)(DC)(DG)(DT)(DG)(DC)(DC)(DT) (DG)(DG) (DA)(DG)(DA)(DC)(DT)(DA)(DG) (DG)(DG)(DA)(DG)(DT)(DA)(DA)(DT)(DC)(DC) (DC)(DC)(DT) (DT)(DG)(DG)(DC)(DG)(DG) (DT)(DT)(DA)(DA)(DA)(DA)(DC)(DG)(DC)(DG) (DG)(DG)(DG)(DG) (DA)(DC)(DA)(DG)(DC) (DG)(DC)(DG)(DT)(DA)(DC)(DG)(DT)(DG)(DC) (DG)(DT)(DT)(DT)(DA) (DA)(DG)(DC)(DG) (DG)(DT)(DG)(DC)(DT)(DA)(DG)(DA)(DG)(DC) (DT)(DG)(DT)(DC)(DT)(DA) (DC)(DG)(DA) (DC)(DC)(DA)(DA)(DT)(DT)(DG)(DA)(DG)(DC) (DG)(DG)(DC)(DC)(DT)(DC)(DG) (DG)(DC) (DA)(DC)(DC)(DG)(DG)(DG)(DA)(DT)(DT)(DC) (DT)(DC)(DC)(DA)(DG)(DG)(DG)(DC) (DG) (DG)(DC)(DC)(DG)(DC)(DG)(DT)(DA)(DT)(DA) (DG)(DG)(DG)(DT)(DC)(DC)(DA)(DT)(DC) (DA)(DC)(DA)(DT)(DA)(DA)(DG)(DT) |
-分子 #7: DNA Widom601 (208bp) strand2
| 分子 | 名称: DNA Widom601 (208bp) strand2 / タイプ: dna / ID: 7 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 63.988711 KDa |
| 配列 | 文字列: (DA)(DC)(DT)(DT)(DA)(DT)(DG)(DT)(DG)(DA) (DT)(DG)(DG)(DA)(DC)(DC)(DC)(DT)(DA)(DT) (DA)(DC)(DG)(DC)(DG)(DG)(DC)(DC)(DG) (DC)(DC)(DC)(DT)(DG)(DG)(DA)(DG)(DA)(DA) (DT) (DC)(DC)(DC)(DG)(DG) ...文字列: (DA)(DC)(DT)(DT)(DA)(DT)(DG)(DT)(DG)(DA) (DT)(DG)(DG)(DA)(DC)(DC)(DC)(DT)(DA)(DT) (DA)(DC)(DG)(DC)(DG)(DG)(DC)(DC)(DG) (DC)(DC)(DC)(DT)(DG)(DG)(DA)(DG)(DA)(DA) (DT) (DC)(DC)(DC)(DG)(DG)(DT)(DG)(DC) (DC)(DG)(DA)(DG)(DG)(DC)(DC)(DG)(DC)(DT) (DC)(DA) (DA)(DT)(DT)(DG)(DG)(DT)(DC) (DG)(DT)(DA)(DG)(DA)(DC)(DA)(DG)(DC)(DT) (DC)(DT)(DA) (DG)(DC)(DA)(DC)(DC)(DG) (DC)(DT)(DT)(DA)(DA)(DA)(DC)(DG)(DC)(DA) (DC)(DG)(DT)(DA) (DC)(DG)(DC)(DG)(DC) (DT)(DG)(DT)(DC)(DC)(DC)(DC)(DC)(DG)(DC) (DG)(DT)(DT)(DT)(DT) (DA)(DA)(DC)(DC) (DG)(DC)(DC)(DA)(DA)(DG)(DG)(DG)(DG)(DA) (DT)(DT)(DA)(DC)(DT)(DC) (DC)(DC)(DT) (DA)(DG)(DT)(DC)(DT)(DC)(DC)(DA)(DG)(DG) (DC)(DA)(DC)(DG)(DT)(DG)(DT) (DC)(DA) (DG)(DA)(DT)(DA)(DT)(DA)(DT)(DA)(DC)(DA) (DT)(DC)(DC)(DT)(DG)(DT)(DG)(DC) (DA) (DT)(DG)(DT)(DA)(DT)(DT)(DG)(DA)(DA)(DC) (DA)(DG)(DC)(DG)(DA)(DC)(DC)(DT)(DT) (DG)(DC)(DC)(DG)(DG)(DA)(DG)(DT) |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 39.2 K / 装置: FEI VITROBOT MARK III |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 10 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 倍率(補正後): 81000 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.25 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー














 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)