+ Open data
Open data
- Basic information
Basic information
| Entry |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| Title | structure of the K9me3-H2A.Z nucleosome | |||||||||
 Map data Map data | ||||||||||
 Sample Sample |
| |||||||||
 Keywords Keywords | heterochromatin / nucleosome / chromatin / GENE REGULATION / STRUCTURAL PROTEIN | |||||||||
| Biological species |   | |||||||||
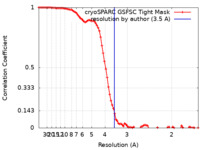
| Method | single particle reconstruction / cryo EM / Resolution: 3.5 Å | |||||||||
 Authors Authors | Tan D | |||||||||
| Funding support |  United States, 1 items United States, 1 items
| |||||||||
 Citation Citation |  Journal: Structure / Year: 2024 Journal: Structure / Year: 2024Title: Structural mechanism of HP1⍺-dependent transcriptional repression and chromatin compaction. Authors: Vladyslava Sokolova / Jacob Miratsky / Vladimir Svetlov / Michael Brenowitz / John Vant / Tyler S Lewis / Kelly Dryden / Gahyun Lee / Shayan Sarkar / Evgeny Nudler / Abhishek Singharoy / Dongyan Tan /  Abstract: Heterochromatin protein 1 (HP1) plays a central role in establishing and maintaining constitutive heterochromatin. However, the mechanisms underlying HP1-nucleosome interactions and their ...Heterochromatin protein 1 (HP1) plays a central role in establishing and maintaining constitutive heterochromatin. However, the mechanisms underlying HP1-nucleosome interactions and their contributions to heterochromatin functions remain elusive. Here, we present the cryoelectron microscopy (cryo-EM) structure of an HP1α dimer bound to an H2A.Z-nucleosome, revealing two distinct HP1α-nucleosome interfaces. The primary HP1α binding site is located at the N terminus of histone H3, specifically at the trimethylated lysine 9 (K9me3) region, while a secondary binding site is situated near histone H2B, close to nucleosome superhelical location 4 (SHL4). Our biochemical data further demonstrates that HP1α binding influences the dynamics of DNA on the nucleosome. It promotes DNA unwrapping near the nucleosome entry and exit sites while concurrently restricting DNA accessibility in the vicinity of SHL4. Our study offers a model for HP1α-mediated heterochromatin maintenance and gene silencing. It also sheds light on the H3K9me-independent role of HP1 in responding to DNA damage. | |||||||||
| History |
|
- Structure visualization
Structure visualization
| Supplemental images |
|---|
- Downloads & links
Downloads & links
-EMDB archive
| Map data |  emd_42773.map.gz emd_42773.map.gz | 59.8 MB |  EMDB map data format EMDB map data format | |
|---|---|---|---|---|
| Header (meta data) |  emd-42773-v30.xml emd-42773-v30.xml emd-42773.xml emd-42773.xml | 19.3 KB 19.3 KB | Display Display |  EMDB header EMDB header |
| FSC (resolution estimation) |  emd_42773_fsc.xml emd_42773_fsc.xml | 9.6 KB | Display |  FSC data file FSC data file |
| Images |  emd_42773.png emd_42773.png | 82.6 KB | ||
| Filedesc metadata |  emd-42773.cif.gz emd-42773.cif.gz | 5.7 KB | ||
| Others |  emd_42773_half_map_1.map.gz emd_42773_half_map_1.map.gz emd_42773_half_map_2.map.gz emd_42773_half_map_2.map.gz | 59.4 MB 59.4 MB | ||
| Archive directory |  http://ftp.pdbj.org/pub/emdb/structures/EMD-42773 http://ftp.pdbj.org/pub/emdb/structures/EMD-42773 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-42773 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-42773 | HTTPS FTP |
-Related structure data
- Links
Links
| EMDB pages |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- Map
Map
| File |  Download / File: emd_42773.map.gz / Format: CCP4 / Size: 64 MB / Type: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) Download / File: emd_42773.map.gz / Format: CCP4 / Size: 64 MB / Type: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Projections & slices | Image control
Images are generated by Spider. | ||||||||||||||||||||||||||||||||||||
| Voxel size | X=Y=Z: 0.825 Å | ||||||||||||||||||||||||||||||||||||
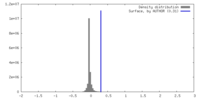
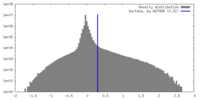
| Density |
| ||||||||||||||||||||||||||||||||||||
| Symmetry | Space group: 1 | ||||||||||||||||||||||||||||||||||||
| Details | EMDB XML:
|
-Supplemental data
- Sample components
Sample components
-Entire : HP1alpha dimer bound to H2A.Z nucleosome
| Entire | Name: HP1alpha dimer bound to H2A.Z nucleosome |
|---|---|
| Components |
|
-Supramolecule #1: HP1alpha dimer bound to H2A.Z nucleosome
| Supramolecule | Name: HP1alpha dimer bound to H2A.Z nucleosome / type: complex / ID: 1 / Parent: 0 / Macromolecule list: #1, #3-#6, #10-#11 |
|---|---|
| Source (natural) | Organism:  |
| Molecular weight | Theoretical: 288 MDa |
-Macromolecule #1: Histone H3, K9me3
| Macromolecule | Name: Histone H3, K9me3 / type: protein_or_peptide / ID: 1 / Enantiomer: LEVO |
|---|---|
| Source (natural) | Organism: |
| Recombinant expression | Organism:  |
| Sequence | String: ARTKQTARK(me3) STGGKAPRKQ LATKAARKSA PATGGVKKPH RYRPGTVALR EIRRYQKSTE L LIRKLPFQ RLVREIAQDF KTDLRFQSSA VMALQEASEA YLVALFEDTN LAAIHAKRVT IM PKDIQLA RRIRGERA |
-Macromolecule #2: Histone H4
| Macromolecule | Name: Histone H4 / type: protein_or_peptide / ID: 2 / Enantiomer: LEVO |
|---|---|
| Source (natural) | Organism: |
| Recombinant expression | Organism:  |
| Sequence | String: MSGRGKGGKG LGKGGAKRHR KVLRDNIQGI TKPAIRRLAR RGGVKRISGL IYEETRGVLK VFLENVIRD AVTYTEHAKR KTVTAMDVVY ALKRQGRTLY GFGG |
-Macromolecule #3: Histone H2A.Z
| Macromolecule | Name: Histone H2A.Z / type: protein_or_peptide / ID: 3 / Enantiomer: LEVO |
|---|---|
| Source (natural) | Organism:  |
| Recombinant expression | Organism:  |
| Sequence | String: MAGGKAGKDS GKAKTKAVSR SQRAGLQFPV GRIHRHLKSR TTSHGRVGAT AAVYSAAILE YLTAEVLEL AGNASKDLKV KRITPRHLQL AIRGDEELDS LIKATIAGGG VIPHIHKSLI G KKGQQKTV |
-Macromolecule #4: Histone H2B
| Macromolecule | Name: Histone H2B / type: protein_or_peptide / ID: 4 / Enantiomer: LEVO |
|---|---|
| Source (natural) | Organism: |
| Recombinant expression | Organism:  |
| Sequence | String: MPEPAKSAPA PKKGSKKAVT KTQKKDGKKR RKSRKESYAI YVYKVLKQVH PDTGISSKAM SIMNSFVND VFERIAGEAS RLAHYNKRST ITSREIQTAV RLLLPGELAK HAVSEGTKAV T KYTSAK |
-Macromolecule #5: Widom601 (208bp) strand1
| Macromolecule | Name: Widom601 (208bp) strand1 / type: dna / ID: 5 / Classification: DNA |
|---|---|
| Source (natural) | Organism: synthetic construct (others) |
| Sequence | String: ACTCCGGCAA GGTCGCTGTT CAATACATGC ACAGGATGTA TATATCTGAC ACGTGCCTGG AGACTAGGGA GTAATCCCCT TGGCGGTTAA AACGCGGGGG ACAGCGCGTA CGTGCGTTTA AGCGGTGCTA GAGCTGTCTA CGACCAATTG AGCGGCCTCG GCACCGGGAT ...String: ACTCCGGCAA GGTCGCTGTT CAATACATGC ACAGGATGTA TATATCTGAC ACGTGCCTGG AGACTAGGGA GTAATCCCCT TGGCGGTTAA AACGCGGGGG ACAGCGCGTA CGTGCGTTTA AGCGGTGCTA GAGCTGTCTA CGACCAATTG AGCGGCCTCG GCACCGGGAT TCTCCAGGGC GGCCGCGTAT AGGGTCCATC ACATAAGT |
-Macromolecule #6: Widom601 (208bp) strand2
| Macromolecule | Name: Widom601 (208bp) strand2 / type: dna / ID: 6 / Classification: DNA |
|---|---|
| Source (natural) | Organism: synthetic construct (others) |
| Sequence | String: ACTTATGTGA TGGACCCTAT ACGCGGCCGC CCTGGAGAAT CCCGGTGCCG AGGCCGCTCA ATTGGTCGTA GACAGCTCTA GCACCGCTTA AACGCACGTA CGCGCTGTCC CCCGCGTTTT AACCGCCAAG GGGATTACTC CCTAGTCTCC AGGCACGTGT CAGATATATA ...String: ACTTATGTGA TGGACCCTAT ACGCGGCCGC CCTGGAGAAT CCCGGTGCCG AGGCCGCTCA ATTGGTCGTA GACAGCTCTA GCACCGCTTA AACGCACGTA CGCGCTGTCC CCCGCGTTTT AACCGCCAAG GGGATTACTC CCTAGTCTCC AGGCACGTGT CAGATATATA CATCCTGTGC ATGTATTGAA CAGCGACCTT GCCGGAGT |
-Experimental details
-Structure determination
| Method | cryo EM |
|---|---|
 Processing Processing | single particle reconstruction |
| Aggregation state | particle |
- Sample preparation
Sample preparation
| Buffer | pH: 7.5 |
|---|---|
| Vitrification | Cryogen name: ETHANE / Chamber humidity: 100 % / Chamber temperature: 39.2 K / Instrument: FEI VITROBOT MARK III |
- Electron microscopy
Electron microscopy
| Microscope | FEI TITAN KRIOS |
|---|---|
| Specialist optics | Energy filter - Name: GIF Bioquantum / Energy filter - Slit width: 10 eV |
| Image recording | Film or detector model: GATAN K3 (6k x 4k) / Average electron dose: 50.0 e/Å2 |
| Electron beam | Acceleration voltage: 300 kV / Electron source:  FIELD EMISSION GUN FIELD EMISSION GUN |
| Electron optics | C2 aperture diameter: 100.0 µm / Calibrated magnification: 81000 / Illumination mode: FLOOD BEAM / Imaging mode: BRIGHT FIELD / Cs: 2.7 mm / Nominal defocus max: 2.25 µm / Nominal defocus min: 1.0 µm |
| Sample stage | Specimen holder model: FEI TITAN KRIOS AUTOGRID HOLDER / Cooling holder cryogen: NITROGEN |
| Experimental equipment |  Model: Titan Krios / Image courtesy: FEI Company |
+ Image processing
Image processing
-Atomic model buiding 1
| Initial model | PDB ID: Chain - Source name: AlphaFold / Chain - Initial model type: in silico model |
|---|---|
| Refinement | Protocol: FLEXIBLE FIT / Target criteria: Cross-correlation coefficient |
 Movie
Movie Controller
Controller








 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)