+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | KIF1A[1-393] ADP bound in complex with a microtubule | ||||||||||||||||||
 マップデータ マップデータ | Primary map, locally refined on the central asymmetric unit | ||||||||||||||||||
 試料 試料 |
| ||||||||||||||||||
 キーワード キーワード | KIF1A / kinesin / motility / microtubule / tubulin / MOTOR PROTEIN | ||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報neuronal dense core vesicle membrane / plus-end-directed kinesin ATPase / dense core granule cytoskeletal transport / anterograde neuronal dense core vesicle transport / retrograde neuronal dense core vesicle transport / regulation of dendritic spine development / regulation of dendritic spine morphogenesis / anterograde axonal transport / plus-end-directed microtubule motor activity / Microtubule-dependent trafficking of connexons from Golgi to the plasma membrane ...neuronal dense core vesicle membrane / plus-end-directed kinesin ATPase / dense core granule cytoskeletal transport / anterograde neuronal dense core vesicle transport / retrograde neuronal dense core vesicle transport / regulation of dendritic spine development / regulation of dendritic spine morphogenesis / anterograde axonal transport / plus-end-directed microtubule motor activity / Microtubule-dependent trafficking of connexons from Golgi to the plasma membrane / Resolution of Sister Chromatid Cohesion / Hedgehog 'off' state / Cilium Assembly / Intraflagellar transport / COPI-dependent Golgi-to-ER retrograde traffic / Mitotic Prometaphase / Carboxyterminal post-translational modifications of tubulin / RHOH GTPase cycle / EML4 and NUDC in mitotic spindle formation / Sealing of the nuclear envelope (NE) by ESCRT-III / Kinesins / PKR-mediated signaling / Separation of Sister Chromatids / The role of GTSE1 in G2/M progression after G2 checkpoint / Aggrephagy / Kinesins / RHO GTPases activate IQGAPs / RHO GTPases Activate Formins / HSP90 chaperone cycle for steroid hormone receptors (SHR) in the presence of ligand / kinesin complex / MHC class II antigen presentation / Recruitment of NuMA to mitotic centrosomes / COPI-mediated anterograde transport / COPI-dependent Golgi-to-ER retrograde traffic / cytoskeletal motor activity / neuronal dense core vesicle / microtubule-based process / vesicle-mediated transport / axon cytoplasm / structural constituent of cytoskeleton / microtubule cytoskeleton organization / mitotic cell cycle / microtubule cytoskeleton / microtubule binding / 加水分解酵素; 酸無水物に作用; GTPに作用・細胞または細胞小器官の運動に関与 / microtubule / axon / GTPase activity / synapse / dendrite / GTP binding / perinuclear region of cytoplasm / ATP hydrolysis activity / ATP binding / metal ion binding / identical protein binding / cytoplasm 類似検索 - 分子機能 | ||||||||||||||||||
| 生物種 |  Homo sapiens (ヒト) / Homo sapiens (ヒト) /  | ||||||||||||||||||
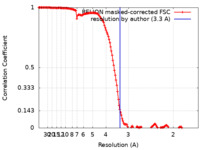
| 手法 | らせん対称体再構成法 / クライオ電子顕微鏡法 / 解像度: 3.3 Å | ||||||||||||||||||
 データ登録者 データ登録者 | Benoit MPMH / Rao L / Asenjo AB / Gennerich A / Sosa H | ||||||||||||||||||
| 資金援助 |  米国, 5件 米国, 5件
| ||||||||||||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2024 ジャーナル: Nat Commun / 年: 2024タイトル: Cryo-EM unveils kinesin KIF1A's processivity mechanism and the impact of its pathogenic variant P305L. 著者: Matthieu P M H Benoit / Lu Rao / Ana B Asenjo / Arne Gennerich / Hernando Sosa /  要旨: Mutations in the microtubule-associated motor protein KIF1A lead to severe neurological conditions known as KIF1A-associated neurological disorders (KAND). Despite insights into its molecular ...Mutations in the microtubule-associated motor protein KIF1A lead to severe neurological conditions known as KIF1A-associated neurological disorders (KAND). Despite insights into its molecular mechanism, high-resolution structures of KIF1A-microtubule complexes remain undefined. Here, we present 2.7-3.5 Å resolution structures of dimeric microtubule-bound KIF1A, including the pathogenic P305L mutant, across various nucleotide states. Our structures reveal that KIF1A binds microtubules in one- and two-heads-bound configurations, with both heads exhibiting distinct conformations with tight inter-head connection. Notably, KIF1A's class-specific loop 12 (K-loop) forms electrostatic interactions with the C-terminal tails of both α- and β-tubulin. The P305L mutation does not disrupt these interactions but alters loop-12's conformation, impairing strong microtubule-binding. Structure-function analysis reveals the K-loop and head-head coordination as major determinants of KIF1A's superprocessive motility. Our findings advance the understanding of KIF1A's molecular mechanism and provide a basis for developing structure-guided therapeutics against KAND. | ||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_42547.map.gz emd_42547.map.gz | 33.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-42547-v30.xml emd-42547-v30.xml emd-42547.xml emd-42547.xml | 25.3 KB 25.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_42547_fsc.xml emd_42547_fsc.xml | 14.8 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_42547.png emd_42547.png | 119.9 KB | ||
| マスクデータ |  emd_42547_msk_1.map emd_42547_msk_1.map emd_42547_msk_2.map emd_42547_msk_2.map emd_42547_msk_3.map emd_42547_msk_3.map emd_42547_msk_4.map emd_42547_msk_4.map | 274.6 MB 274.6 MB 274.6 MB 274.6 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-42547.cif.gz emd-42547.cif.gz | 7.5 KB | ||
| その他 |  emd_42547_additional_1.map.gz emd_42547_additional_1.map.gz emd_42547_additional_2.map.gz emd_42547_additional_2.map.gz emd_42547_half_map_1.map.gz emd_42547_half_map_1.map.gz emd_42547_half_map_2.map.gz emd_42547_half_map_2.map.gz | 239.9 MB 241 MB 219.9 MB 220 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-42547 http://ftp.pdbj.org/pub/emdb/structures/EMD-42547 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-42547 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-42547 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_42547_validation.pdf.gz emd_42547_validation.pdf.gz | 1.2 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_42547_full_validation.pdf.gz emd_42547_full_validation.pdf.gz | 1.2 MB | 表示 | |
| XML形式データ |  emd_42547_validation.xml.gz emd_42547_validation.xml.gz | 21.9 KB | 表示 | |
| CIF形式データ |  emd_42547_validation.cif.gz emd_42547_validation.cif.gz | 29 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-42547 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-42547 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-42547 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-42547 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8utrMC  8utnC  8utoC  8utpC  8utqC  8utsC  8uttC  8utuC  8utvC  8utwC  8utyC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_42547.map.gz / 形式: CCP4 / 大きさ: 274.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_42547.map.gz / 形式: CCP4 / 大きさ: 274.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Primary map, locally refined on the central asymmetric unit | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.844 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_42547_msk_1.map emd_42547_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
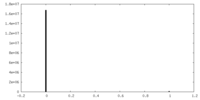
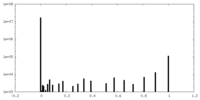
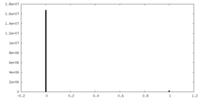
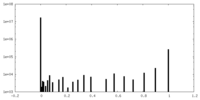
| 密度ヒストグラム |
-マスク #2
| ファイル |  emd_42547_msk_2.map emd_42547_msk_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-マスク #3
| ファイル |  emd_42547_msk_3.map emd_42547_msk_3.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-マスク #4
| ファイル |  emd_42547_msk_4.map emd_42547_msk_4.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Map low pass filtered to 6 A
| ファイル | emd_42547_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Map low pass filtered to 6 A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Map low pass filtered to 20 A
| ファイル | emd_42547_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Map low pass filtered to 20 A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map from the gold Standard refinement
| ファイル | emd_42547_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map from the gold Standard refinement | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map from the gold Standard refinement
| ファイル | emd_42547_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map from the gold Standard refinement | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : KIF1A[1-393] ADP in complex with a 15R microtubule
| 全体 | 名称: KIF1A[1-393] ADP in complex with a 15R microtubule |
|---|---|
| 要素 |
|
-超分子 #1: KIF1A[1-393] ADP in complex with a 15R microtubule
| 超分子 | 名称: KIF1A[1-393] ADP in complex with a 15R microtubule / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#3 |
|---|
-分子 #1: Kinesin-like protein KIF1A
| 分子 | 名称: Kinesin-like protein KIF1A / タイプ: protein_or_peptide / ID: 1 詳細: "linker" residues are comprised of a leucine zipper based on S. cerevisiae GCN4, which is followed by the C-terminal strep-tag コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 49.288438 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MAGASVKVAV RVRPFNSREM SRDSKCIIQM SGSTTTIVNP KQPKETPKSF SFDYSYWSHT SPEDINYASQ KQVYRDIGEE MLQHAFEGY NVCIFAYGQT GAGKSYTMMG KQEKDQQGII PQLCEDLFSR INDTTNDNMS YSVEVSYMEI YCERVRDLLN P KNKGNLRV ...文字列: MAGASVKVAV RVRPFNSREM SRDSKCIIQM SGSTTTIVNP KQPKETPKSF SFDYSYWSHT SPEDINYASQ KQVYRDIGEE MLQHAFEGY NVCIFAYGQT GAGKSYTMMG KQEKDQQGII PQLCEDLFSR INDTTNDNMS YSVEVSYMEI YCERVRDLLN P KNKGNLRV REHPLLGPYV EDLSKLAVTS YNDIQDLMDS GNKARTVAAT NMNETSSRSH AVFNIIFTQK RHDAETNITT EK VSKISLV DLAGSERADS TGAKGTRLKE GANINKSLTT LGKVISALAE MDSGPNKNKK KKKTDFIPYR DSVLTWLLRE NLG GNSRTA MVAALSPADI NYDETLSTLR YADRAKQIRC NAVINEDPNN KLIRELKDEV TRLRDLLYAQ GLGDITDGAG VKQL EDKVE ELASKNYHLE NEVARLKKLV EFTSAWSHPQ FEK UniProtKB: Kinesin-like protein KIF1A |
-分子 #2: Tubulin alpha-1B chain
| 分子 | 名称: Tubulin alpha-1B chain / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 50.204445 KDa |
| 配列 | 文字列: MRECISIHVG QAGVQIGNAC WELYCLEHGI QPDGQMPSDK TIGGGDDSFN TFFSETGAGK HVPRAVFVDL EPTVIDEVRT GTYRQLFHP EQLITGKEDA ANNYARGHYT IGKEIIDLVL DRIRKLADQC TGLQGFLVFH SFGGGTGSGF TSLLMERLSV D YGKKSKLE ...文字列: MRECISIHVG QAGVQIGNAC WELYCLEHGI QPDGQMPSDK TIGGGDDSFN TFFSETGAGK HVPRAVFVDL EPTVIDEVRT GTYRQLFHP EQLITGKEDA ANNYARGHYT IGKEIIDLVL DRIRKLADQC TGLQGFLVFH SFGGGTGSGF TSLLMERLSV D YGKKSKLE FSIYPAPQVS TAVVEPYNSI LTTHTTLEHS DCAFMVDNEA IYDICRRNLD IERPTYTNLN RLISQIVSSI TA SLRFDGA LNVDLTEFQT NLVPYPRIHF PLATYAPVIS AEKAYHEQLS VAEITNACFE PANQMVKCDP RHGKYMACCL LYR GDVVPK DVNAAIATIK TKRSIQFVDW CPTGFKVGIN YQPPTVVPGG DLAKVQRAVC MLSNTTAIAE AWARLDHKFD LMYA KRAFV HWYVGEGMEE GEFSEAREDM AALEKDYEEV GVDSVEGEGE EEGEEY UniProtKB: Tubulin alpha-1B chain |
-分子 #3: Tubulin beta-2B chain
| 分子 | 名称: Tubulin beta-2B chain / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 49.999887 KDa |
| 配列 | 文字列: MREIVHIQAG QCGNQIGAKF WEVISDEHGI DPTGSYHGDS DLQLERINVY YNEATGNKYV PRAILVDLEP GTMDSVRSGP FGQIFRPDN FVFGQSGAGN NWAKGHYTEG AELVDSVLDV VRKESESCDC LQGFQLTHSL GGGTGSGMGT LLISKIREEY P DRIMNTFS ...文字列: MREIVHIQAG QCGNQIGAKF WEVISDEHGI DPTGSYHGDS DLQLERINVY YNEATGNKYV PRAILVDLEP GTMDSVRSGP FGQIFRPDN FVFGQSGAGN NWAKGHYTEG AELVDSVLDV VRKESESCDC LQGFQLTHSL GGGTGSGMGT LLISKIREEY P DRIMNTFS VMPSPKVSDT VVEPYNATLS VHQLVENTDE TYCIDNEALY DICFRTLKLT TPTYGDLNHL VSATMSGVTT CL RFPGQLN ADLRKLAVNM VPFPRLHFFM PGFAPLTSRG SQQYRALTVP ELTQQMFDSK NMMAACDPRH GRYLTVAAIF RGR MSMKEV DEQMLNVQNK NSSYFVEWIP NNVKTAVCDI PPRGLKMSAT FIGNSTAIQE LFKRISEQFT AMFRRKAFLH WYTG EGMDE MEFTEAESNM NDLVSEYQQY QDATADEQGE FEEEEGEDEA UniProtKB: Tubulin beta chain |
-分子 #4: ADENOSINE-5'-DIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-DIPHOSPHATE / タイプ: ligand / ID: 4 / コピー数: 1 / 式: ADP |
|---|---|
| 分子量 | 理論値: 427.201 Da |
| Chemical component information |  ChemComp-ADP: |
-分子 #5: GUANOSINE-5'-TRIPHOSPHATE
| 分子 | 名称: GUANOSINE-5'-TRIPHOSPHATE / タイプ: ligand / ID: 5 / コピー数: 1 / 式: GTP |
|---|---|
| 分子量 | 理論値: 523.18 Da |
| Chemical component information |  ChemComp-GTP: |
-分子 #6: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 6 / コピー数: 1 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-分子 #7: GUANOSINE-5'-DIPHOSPHATE
| 分子 | 名称: GUANOSINE-5'-DIPHOSPHATE / タイプ: ligand / ID: 7 / コピー数: 1 / 式: GDP |
|---|---|
| 分子量 | 理論値: 443.201 Da |
| Chemical component information |  ChemComp-GDP: |
-分子 #8: TAXOL
| 分子 | 名称: TAXOL / タイプ: ligand / ID: 8 / コピー数: 1 / 式: TA1 |
|---|---|
| 分子量 | 理論値: 853.906 Da |
| Chemical component information |  ChemComp-TA1: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | filament |
- 試料調製
試料調製
| 緩衝液 | pH: 6.8 構成要素:
| ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| グリッド | モデル: UltrAuFoil / 材質: GOLD / 前処理 - タイプ: PLASMA CLEANING | ||||||||||||
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 67.5 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 0.8 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー























 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)