+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Major interface of Streptococcal surface enolase dimer from AP53 group A streptococcus bound to a lipid vesicle | |||||||||
 マップデータ マップデータ | SEN dimer map on the surface of lipid major dimer interface | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Glycolysis / plasminogen binder / membrane protein / LYASE | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報phosphopyruvate hydratase / phosphopyruvate hydratase complex / phosphopyruvate hydratase activity / peptidoglycan-based cell wall / glycolytic process / magnesium ion binding / cell surface / extracellular region 類似検索 - 分子機能 | |||||||||
| 生物種 |  Streptococcus pyogenes (化膿レンサ球菌) Streptococcus pyogenes (化膿レンサ球菌) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.8 Å | |||||||||
 データ登録者 データ登録者 | Tjia-Fleck S / Readnour BM / Castellino FJ | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Publishedタイトル: Streptococcus surface alpha enolase exposed dimers were found to be the active form on lipid surface that binds to human plasminogen 著者: Tjia-Fleck S / Readnour BM / Castellino FJ | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_42435.map.gz emd_42435.map.gz | 35.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-42435-v30.xml emd-42435-v30.xml emd-42435.xml emd-42435.xml | 17.8 KB 17.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_42435_fsc.xml emd_42435_fsc.xml | 7.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_42435.png emd_42435.png | 92.8 KB | ||
| Filedesc metadata |  emd-42435.cif.gz emd-42435.cif.gz | 5.8 KB | ||
| その他 |  emd_42435_additional_1.map.gz emd_42435_additional_1.map.gz emd_42435_half_map_1.map.gz emd_42435_half_map_1.map.gz emd_42435_half_map_2.map.gz emd_42435_half_map_2.map.gz | 35.2 MB 34.7 MB 34.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-42435 http://ftp.pdbj.org/pub/emdb/structures/EMD-42435 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-42435 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-42435 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_42435_validation.pdf.gz emd_42435_validation.pdf.gz | 726.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_42435_full_validation.pdf.gz emd_42435_full_validation.pdf.gz | 726.1 KB | 表示 | |
| XML形式データ |  emd_42435_validation.xml.gz emd_42435_validation.xml.gz | 14.4 KB | 表示 | |
| CIF形式データ |  emd_42435_validation.cif.gz emd_42435_validation.cif.gz | 18.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-42435 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-42435 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-42435 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-42435 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8uopMC  8uoyC  8uq6C M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_42435.map.gz / 形式: CCP4 / 大きさ: 37.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_42435.map.gz / 形式: CCP4 / 大きさ: 37.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | SEN dimer map on the surface of lipid major dimer interface | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.078 Å | ||||||||||||||||||||||||||||||||||||
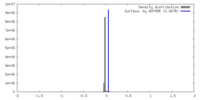
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: SEN dimer map on the surface of lipid combined dimer interfaces
| ファイル | emd_42435_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | SEN dimer map on the surface of lipid combined dimer interfaces | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
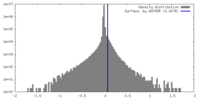
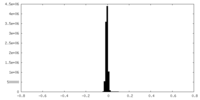
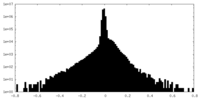
| 密度ヒストグラム |
-ハーフマップ: SEN dimer half map on the surface of lipid major dimer interface B
| ファイル | emd_42435_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | SEN dimer half map on the surface of lipid major dimer interface B | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
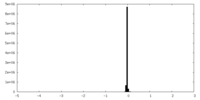
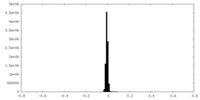
| 密度ヒストグラム |
-ハーフマップ: SEN dimer half map on the surface of lipid major dimer interface A
| ファイル | emd_42435_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | SEN dimer half map on the surface of lipid major dimer interface A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
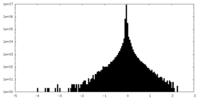
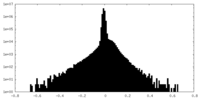
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Streptococcal surface enolase major interface embedded into the s...
| 全体 | 名称: Streptococcal surface enolase major interface embedded into the surface of a lipid vesicle |
|---|---|
| 要素 |
|
-超分子 #1: Streptococcal surface enolase major interface embedded into the s...
| 超分子 | 名称: Streptococcal surface enolase major interface embedded into the surface of a lipid vesicle タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Streptococcus pyogenes (化膿レンサ球菌) Streptococcus pyogenes (化膿レンサ球菌) |
| 分子量 | 理論値: 95 KDa |
-分子 #1: Enolase
| 分子 | 名称: Enolase / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO / EC番号: phosphopyruvate hydratase |
|---|---|
| 由来(天然) | 生物種:  Streptococcus pyogenes (化膿レンサ球菌) Streptococcus pyogenes (化膿レンサ球菌) |
| 分子量 | 理論値: 47.543281 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: HMSIITDVYA REVLDSRGNP TLEVEVYTES GAFGRGMVPS GASTGEHEAV ELRDGDKSRY LGLGTQKAVD NVNNIIAEAI IGYDVRDQQ AIDRAMIALD GTPNKGKLGA NAILGVSIAV ARAAADYLEV PLYTYLGGFN TKVLPTPMMN IINGGSHSDA P IAFQEFMI ...文字列: HMSIITDVYA REVLDSRGNP TLEVEVYTES GAFGRGMVPS GASTGEHEAV ELRDGDKSRY LGLGTQKAVD NVNNIIAEAI IGYDVRDQQ AIDRAMIALD GTPNKGKLGA NAILGVSIAV ARAAADYLEV PLYTYLGGFN TKVLPTPMMN IINGGSHSDA P IAFQEFMI MPVGAPTFKE GLRWGAEVFH ALKKILKERG LVTAVGDEGG FAPKFEGTED GVETILKAIE AAGYEAGENG IM IGFDCAS SEFYDKERKV YDYTKFEGEG AAVRTSAEQV DYLEELVNKY PIITIEDGMD ENDWDGWKVL TERLGKRVQL VGD DFFVTN TEYLARGIKE NAANSILIKV NQIGTLTETF EAIEMAKEAG YTAVVSHRSG ETEDSTIADI AVATNAGQIK TGSL SRTDR IAKYNQLLRI EDQLGEVAQY KGIKSFYNLK K UniProtKB: Enolase |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| グリッド | 材質: GOLD / メッシュ: 400 / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 298 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: SPOT SCAN / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 29.0 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 81000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー









 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)