+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Caenorhabditis elegans Otopetrin 8 (CeOtop8) in pH 8.0 | ||||||||||||
 マップデータ マップデータ | |||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | Proton channel / Otop / C.elegans / Otopetrin / MEMBRANE PROTEIN | ||||||||||||
| 機能・相同性 | Otopetrin / Otopetrin / proton channel activity / proton transmembrane transport / membrane / plasma membrane / Otopetrin-2 機能・相同性情報 機能・相同性情報 | ||||||||||||
| 生物種 |  | ||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.02 Å | ||||||||||||
 データ登録者 データ登録者 | Gan N / Jiang Y | ||||||||||||
| 資金援助 |  米国, 3件 米国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2024 ジャーナル: Nat Commun / 年: 2024タイトル: Structural mechanism of proton conduction in otopetrin proton channel. 著者: Ninghai Gan / Weizhong Zeng / Yan Han / Qingfeng Chen / Youxing Jiang /   要旨: The otopetrin (OTOP) proteins were recently characterized as extracellular proton-activated proton channels. Several recent OTOP channel structures demonstrated that the channels form a dimer with ...The otopetrin (OTOP) proteins were recently characterized as extracellular proton-activated proton channels. Several recent OTOP channel structures demonstrated that the channels form a dimer with each subunit adopting a double-barrel architecture. However, the structural mechanisms underlying some basic functional properties of the OTOP channels remain unresolved, including extracellular pH activation, proton conducting pathway, and rapid desensitization. In this study, we performed structural and functional characterization of the Caenorhabditis elegans OTOP8 (CeOTOP8) and mouse OTOP2 (mOTOP2) and illuminated a set of conformational changes related to the proton-conducting process in OTOP. The structures of CeOTOP8 reveal the conformational change at the N-terminal part of TM12 that renders the channel in a transiently proton-transferring state, elucidating an inter-barrel, Glu/His-bridged proton passage within each subunit. The structures of mOTOP2 reveal the conformational change at the N-terminal part of TM6 that exposes the central glutamate to the extracellular solution for protonation. In addition, the structural comparison between CeOTOP8 and mOTOP2, along with the structure-based mutagenesis, demonstrates that an inter-subunit movement at the OTOP channel dimer interface plays a central role in regulating channel activity. Combining the structural information from both channels, we propose a working model describing the multi-step conformational changes during the proton conducting process. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_42213.map.gz emd_42213.map.gz | 75.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-42213-v30.xml emd-42213-v30.xml emd-42213.xml emd-42213.xml | 16.2 KB 16.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_42213_fsc.xml emd_42213_fsc.xml | 10.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_42213.png emd_42213.png | 33.5 KB | ||
| Filedesc metadata |  emd-42213.cif.gz emd-42213.cif.gz | 6 KB | ||
| その他 |  emd_42213_half_map_1.map.gz emd_42213_half_map_1.map.gz emd_42213_half_map_2.map.gz emd_42213_half_map_2.map.gz | 69.8 MB 69.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-42213 http://ftp.pdbj.org/pub/emdb/structures/EMD-42213 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-42213 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-42213 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_42213_validation.pdf.gz emd_42213_validation.pdf.gz | 996.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_42213_full_validation.pdf.gz emd_42213_full_validation.pdf.gz | 995.9 KB | 表示 | |
| XML形式データ |  emd_42213_validation.xml.gz emd_42213_validation.xml.gz | 17.6 KB | 表示 | |
| CIF形式データ |  emd_42213_validation.cif.gz emd_42213_validation.cif.gz | 23.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-42213 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-42213 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-42213 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-42213 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_42213.map.gz / 形式: CCP4 / 大きさ: 91.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_42213.map.gz / 形式: CCP4 / 大きさ: 91.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.83 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #1
| ファイル | emd_42213_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
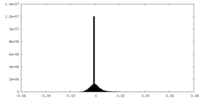
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_42213_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
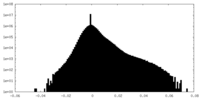
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : CeOTOP8
| 全体 | 名称: CeOTOP8 |
|---|---|
| 要素 |
|
-超分子 #1: CeOTOP8
| 超分子 | 名称: CeOTOP8 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Otopetrin-2
| 分子 | 名称: Otopetrin-2 / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 69.791508 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MLMVDEQLKI GATPMYSPSH NPIVIADDME KGGSDSDSLP SSPTHHHELY RRPWIHEPRA TNFFVRLITS LYALILTIIS LVVEVSPTW RTDMWLAETI FYISMYGVGI LFFAYCYIFI IYPGPYNQLI SVLRKYKIIK NSEVWFIMQS QHNGEGAGTL Y LRLGALFF ...文字列: MLMVDEQLKI GATPMYSPSH NPIVIADDME KGGSDSDSLP SSPTHHHELY RRPWIHEPRA TNFFVRLITS LYALILTIIS LVVEVSPTW RTDMWLAETI FYISMYGVGI LFFAYCYIFI IYPGPYNQLI SVLRKYKIIK NSEVWFIMQS QHNGEGAGTL Y LRLGALFF GSVGIVLFGL ELFLCIENVA CKKVAIAKMI VAIVFTFIQM HFIFCNSKIT VNSSRKIVAF GMMHLISVNL WT WFRFVLA KQEAKAHKKA QLKQTFRKYY SSSSSSSSEE IHELISAVLN STLNNTPATK TMEPVASRLF ALEHFGDVAT FLT TCIVEY SLIGAAIMFI LWKSIGQNNH QQSNSGKRKV KMRIDCSSSS TGLFAGIIFL IGSLVSMGMY TIFESLRNSS GAQL VFGIV DLSLFSIALG ACIIGLWRMR HLQYRLHAHG EVIDEILLII GLIGEILYCA VGIDVFITCR RSADLTVSAL PAFVF VIRM IQVVVQAAFI LTTSRLRCLS KYSMKYKPGK EIITFLLVSN VTLFVFHTFE GMKSSFGFSS KAATQYNYII YAVGPL LVF YRFHSSACLA EIWKHTYSTK SNEYDHEHMS LSDSNLTAIT PISDIKEIPS PQLLKH UniProtKB: Otopetrin-2 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: OTHER / 撮影モード: DIFFRACTION / 最大 デフォーカス(公称値): 2.2 µm / 最小 デフォーカス(公称値): 0.9 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)