[日本語] English
 万見
万見- EMDB-42033: RNA priming complex of Human Polymerase-Alpha-Primase (Conformation 1) -
+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | RNA priming complex of Human Polymerase-Alpha-Primase (Conformation 1) | ||||||||||||
 マップデータ マップデータ | |||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | Polymerase / Primase / Polymerase Alpha / REPLICATION | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報DNA primase AEP / ribonucleotide binding / DNA replication initiation / Telomere C-strand synthesis initiation / DNA/RNA hybrid binding / Inhibition of replication initiation of damaged DNA by RB1/E2F1 / regulation of type I interferon production / alpha DNA polymerase:primase complex / Polymerase switching / Processive synthesis on the lagging strand ...DNA primase AEP / ribonucleotide binding / DNA replication initiation / Telomere C-strand synthesis initiation / DNA/RNA hybrid binding / Inhibition of replication initiation of damaged DNA by RB1/E2F1 / regulation of type I interferon production / alpha DNA polymerase:primase complex / Polymerase switching / Processive synthesis on the lagging strand / lagging strand elongation / Removal of the Flap Intermediate / Polymerase switching on the C-strand of the telomere / mitotic DNA replication initiation / DNA replication, synthesis of primer / DNA strand elongation involved in DNA replication / DNA synthesis involved in DNA repair / leading strand elongation / G1/S-Specific Transcription / DNA replication origin binding / DNA replication initiation / Activation of the pre-replicative complex / Defective pyroptosis / double-strand break repair via nonhomologous end joining / nuclear matrix / protein import into nucleus / DNA-directed RNA polymerase activity / nuclear envelope / single-stranded DNA binding / 4 iron, 4 sulfur cluster binding / DNA-directed DNA polymerase / DNA-directed DNA polymerase activity / DNA replication / ciliary basal body / DNA repair / nucleotide binding / intracellular membrane-bounded organelle / chromatin binding / protein kinase binding / chromatin / nucleolus / magnesium ion binding / DNA binding / zinc ion binding / nucleoplasm / metal ion binding / nucleus / membrane / cytosol 類似検索 - 分子機能 | ||||||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||||||||
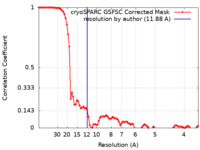
| 手法 | 単粒子再構成法 / ネガティブ染色法 / 解像度: 11.88 Å | ||||||||||||
 データ登録者 データ登録者 | Cordoba JJ / Chazin WJ | ||||||||||||
| 資金援助 |  米国, 3件 米国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: J Mol Biol / 年: 2023 ジャーナル: J Mol Biol / 年: 2023タイトル: Flexibility and Distributive Synthesis Regulate RNA Priming and Handoff in Human DNA Polymerase α-Primase. 著者: John J Cordoba / Elwood A Mullins / Lauren E Salay / Brandt F Eichman / Walter J Chazin /  要旨: DNA replication in eukaryotes relies on the synthesis of a ∼30-nucleotide RNA/DNA primer strand through the dual action of the heterotetrameric polymerase α-primase (pol-prim) enzyme. Synthesis of ...DNA replication in eukaryotes relies on the synthesis of a ∼30-nucleotide RNA/DNA primer strand through the dual action of the heterotetrameric polymerase α-primase (pol-prim) enzyme. Synthesis of the 7-10-nucleotide RNA primer is regulated by the C-terminal domain of the primase regulatory subunit (PRIM2C) and is followed by intramolecular handoff of the primer to pol α for extension by ∼20 nucleotides of DNA. Here, we provide evidence that RNA primer synthesis is governed by a combination of the high affinity and flexible linkage of the PRIM2C domain and the surprisingly low affinity of the primase catalytic domain (PRIM1) for substrate. Using a combination of small angle X-ray scattering and electron microscopy, we found significant variability in the organization of PRIM2C and PRIM1 in the absence and presence of substrate, and that the population of structures with both PRIM2C and PRIM1 in a configuration aligned for synthesis is low. Crosslinking was used to visualize the orientation of PRIM2C and PRIM1 when engaged by substrate as observed by electron microscopy. Microscale thermophoresis was used to measure substrate affinities for a series of pol-prim constructs, which showed that the PRIM1 catalytic domain does not bind the template or emergent RNA-primed templates with appreciable affinity. Together, these findings support a model of RNA primer synthesis in which generation of the nascent RNA strand and handoff of the RNA-primed template from primase to polymerase α is mediated by the high degree of inter-domain flexibility of pol-prim, the ready dissociation of PRIM1 from its substrate, and the much higher affinity of the POLA1cat domain of polymerase α for full-length RNA-primed templates. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
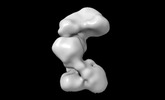
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_42033.map.gz emd_42033.map.gz | 31.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-42033-v30.xml emd-42033-v30.xml emd-42033.xml emd-42033.xml | 21.5 KB 21.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_42033_fsc.xml emd_42033_fsc.xml | 8.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_42033.png emd_42033.png | 18.5 KB | ||
| Filedesc metadata |  emd-42033.cif.gz emd-42033.cif.gz | 6.4 KB | ||
| その他 |  emd_42033_half_map_1.map.gz emd_42033_half_map_1.map.gz emd_42033_half_map_2.map.gz emd_42033_half_map_2.map.gz | 59.4 MB 59.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-42033 http://ftp.pdbj.org/pub/emdb/structures/EMD-42033 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-42033 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-42033 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_42033_validation.pdf.gz emd_42033_validation.pdf.gz | 688.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_42033_full_validation.pdf.gz emd_42033_full_validation.pdf.gz | 687.9 KB | 表示 | |
| XML形式データ |  emd_42033_validation.xml.gz emd_42033_validation.xml.gz | 16.7 KB | 表示 | |
| CIF形式データ |  emd_42033_validation.cif.gz emd_42033_validation.cif.gz | 21.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-42033 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-42033 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-42033 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-42033 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_42033.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_42033.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.79015 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #1
| ファイル | emd_42033_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
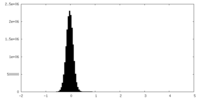
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_42033_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
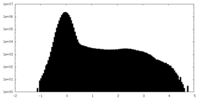
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Ternary complex of polymerase alpha-primase bound to an RNA prime...
| 全体 | 名称: Ternary complex of polymerase alpha-primase bound to an RNA primed-template substrate. |
|---|---|
| 要素 |
|
-超分子 #1: Ternary complex of polymerase alpha-primase bound to an RNA prime...
| 超分子 | 名称: Ternary complex of polymerase alpha-primase bound to an RNA primed-template substrate. タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all / 詳細: Complex stabilized by BS3 crosslinking. |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 200 KDa |
-分子 #1: DNA polymerase alpha subunit B
| 分子 | 名称: DNA polymerase alpha subunit B / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  |
| 配列 | 文字列: GSATPSQKYN SRSNRGEVVT SFGLAQGV S WSGRGGAGNI SLKVLGCPEA LTGSYKSMFQ KLPDIREVLT CKIEELGSEL KEHYKIEAF TPLLAPAQEP VTLLGQIGCD SNGKLNNKSV ILEGDREHSS GAQIPVDLSE LKEYSLFPGQ VVIMEGINT TGRKLVATKL ...文字列: GSATPSQKYN SRSNRGEVVT SFGLAQGV S WSGRGGAGNI SLKVLGCPEA LTGSYKSMFQ KLPDIREVLT CKIEELGSEL KEHYKIEAF TPLLAPAQEP VTLLGQIGCD SNGKLNNKSV ILEGDREHSS GAQIPVDLSE LKEYSLFPGQ VVIMEGINT TGRKLVATKL YEGVPLPFYQ PTEEDADFEQ SMVLVACGPY TTSDSITYDP L LDLIAVIN HDRPDVCILF GPFLDAKHEQ VENCLLTSPF EDIFKQCLRT IIEGTRSSGS HL VFVPSLR DVHHEPVYPQ PPFSYSDLSR EDKKQVQFVS EPCSLSINGV IFGLTSTDLL FHL GAEEIS SSSGTSDRFS RILKHILTQR SYYPLYPPQE DMAIDYESFY VYAQLPVTPD VLII PSELR YFVKDVLGCV CVNPGRLTKG QVGGTFARLY LRRPAADGAE RQSPCIAVQV VRI UniProtKB: DNA polymerase alpha subunit B |
-分子 #2: DNA polymerase alpha catalytic subunit
| 分子 | 名称: DNA polymerase alpha catalytic subunit / タイプ: protein_or_peptide / ID: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MAQLTDEEKY RDCERFKCPC PTCGTENIYD NVFDGSGTDM EPSLYRCSNI DCKASPL TF TVQLSNKLIM DIRRFIKKYY DGWLICEEPT CRNRTRHLPL QFSRTGPLCP ACMKATLQ P EYSDKSLYTQ LCFYRYIFDA ECALEKLTTD HEKDKLKKQF FTPKVLQDYR ...文字列: MAQLTDEEKY RDCERFKCPC PTCGTENIYD NVFDGSGTDM EPSLYRCSNI DCKASPL TF TVQLSNKLIM DIRRFIKKYY DGWLICEEPT CRNRTRHLPL QFSRTGPLCP ACMKATLQ P EYSDKSLYTQ LCFYRYIFDA ECALEKLTTD HEKDKLKKQF FTPKVLQDYR KLKNTAEQF LSRSGYSEVN LSKLFAGCAV KS UniProtKB: DNA polymerase alpha catalytic subunit |
-分子 #3: DNA primase large subunit
| 分子 | 名称: DNA primase large subunit / タイプ: protein_or_peptide / ID: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MEFSGRKWRK LRLAGDQRNA SYPHCLQFYL QPPSENISLI EFENLAIDRV KLLKSVENLG VSYVKGTEQ YQSKLESELR KLKFSYRENL EDEYEPRRRD HISHFILRLA YCQSEELRRW F IQQEMDLL RFRFSILPKD KIQDFLKDSQ LQFEAISDEE KTLREQEIVA ...文字列: MEFSGRKWRK LRLAGDQRNA SYPHCLQFYL QPPSENISLI EFENLAIDRV KLLKSVENLG VSYVKGTEQ YQSKLESELR KLKFSYRENL EDEYEPRRRD HISHFILRLA YCQSEELRRW F IQQEMDLL RFRFSILPKD KIQDFLKDSQ LQFEAISDEE KTLREQEIVA SSPSLSGLKL GF ESIYKIP FADALDLFRG RKVYLEDGFA YVPLKDIVAI ILNEFRAKLS KALALTARSL PAV QSDERL QPLLNHLSHS YTGQDYSTQG NVGKISLDQI DLLSTKSFPP CMRQLHKALR ENHH LRHGG RMQYGLFLKG IGLTLEQALQ FWKQEFIKGK MDPDKFDKGY SYNIRHSFGK EGKRT DYTP FSCLKIILSN PPSQGDYHGC PFRHSDPELL KQKLQSYKIS PGGISQILDL VKGTHY QVA CQKYFEMIHN VDDCGFSLNH PNQFFCESQR ILNGGKDIKK EPIQPETPQP KPSVQKT KD ASSALASLNS SLEMDMEGLE DYFSEDS UniProtKB: DNA primase large subunit |
-分子 #4: DNA primase small subunit
| 分子 | 名称: DNA primase small subunit / タイプ: protein_or_peptide / ID: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  |
| 配列 | 文字列: GSMETFDPTE LPELLKLYYR RLFPYSQYYR WLNYGGVIKN YFQHREFSFT LKDDIYIRYQ SF NNQSDLE KEMQKMNPYK IDIGAVYSHR PNQHNTVKLG AFQAQEKELV FDIDMTDYDD VRR CCSSAD ICPKCWTLMT MAIRIIDRAL KEDFGFKHRL WVYSGRRGVH ...文字列: GSMETFDPTE LPELLKLYYR RLFPYSQYYR WLNYGGVIKN YFQHREFSFT LKDDIYIRYQ SF NNQSDLE KEMQKMNPYK IDIGAVYSHR PNQHNTVKLG AFQAQEKELV FDIDMTDYDD VRR CCSSAD ICPKCWTLMT MAIRIIDRAL KEDFGFKHRL WVYSGRRGVH CWVCDESVRK LSSA VRSGI VEYLSLVKGG QDVKKKVHLS EKIHPFIRKS INIIKKYFEE YALVNQDILE NKESW DKIL ALVPETIHDE LQQSFQKSHN SLQRWEHLKK VASRYQNNIK NDKYGPWLEW EIMLQY CFP RLDINVSKGI NHLLKSPFSV HPKTGRISVP IDLQKVDQFD PFTVPTISFI CRELDAI ST NEEEKEENEA ESDVKHRTRD YKKTSLAPYV KVFEHFLENL DKSRKGELLK KSDLQKDF UniProtKB: DNA primase small subunit |
-分子 #5: RNA priming substrate - primer
| 分子 | 名称: RNA priming substrate - primer / タイプ: rna / ID: 5 / 詳細: 5' triphosphorylated |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: GGAUACUG |
-分子 #6: RNA priming substrate - template
| 分子 | 名称: RNA priming substrate - template / タイプ: dna / ID: 6 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: GTATGTATGT CAGTATCCTG TATGTATGA |
-実験情報
-構造解析
| 手法 | ネガティブ染色法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.009 mg/mL | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| |||||||||||||||
| 染色 | タイプ: NEGATIVE / 材質: Uranyl Formate 詳細: 2.5 uL sample was applied to grid for 1 minute, then blotted with filter paper, washed for 5 seconds with DI water, blotted, washed for 5 seconds in stain, blotted, then stained for 90 seconds. | |||||||||||||||
| グリッド | モデル: Quantifoil / 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: CONTINUOUS / 支持フィルム - Film thickness: 5 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 180 sec. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI 20 |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN ULTRASCAN 4000 (4k x 4k) デジタル化 - サイズ - 横: 4096 pixel / デジタル化 - サイズ - 縦: 4096 pixel / 撮影したグリッド数: 1 / 実像数: 1083 / 平均電子線量: 20.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: SPOT SCAN / 撮影モード: BRIGHT FIELD / Cs: 1.2 mm / 最大 デフォーカス(公称値): 1.5 µm / 最小 デフォーカス(公称値): 1.5 µm |
 ムービー
ムービー コントローラー
コントローラー















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)