+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Locally refined (symmetry based) cryo-EM map of the dimer of the SPARTA oligomer with guide RNA and target DNA | |||||||||
 マップデータ マップデータ | lcocally refined map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | RNA / DNA / Argonaute / TIR / immune system / RNA BINDING PROTEIN | |||||||||
| 生物種 |  Thermoflavifilum thermophilum (バクテリア) Thermoflavifilum thermophilum (バクテリア) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.15 Å | |||||||||
 データ登録者 データ登録者 | Malik R / Kottur J / Aggarwal AK | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2024 ジャーナル: Nat Commun / 年: 2024タイトル: Nucleic acid mediated activation of a short prokaryotic Argonaute immune system. 著者: Jithesh Kottur / Radhika Malik / Aneel K Aggarwal /   要旨: A short prokaryotic Argonaute (pAgo) TIR-APAZ (SPARTA) defense system, activated by invading DNA to unleash its TIR domain for NAD(P) hydrolysis, was recently identified in bacteria. We report the ...A short prokaryotic Argonaute (pAgo) TIR-APAZ (SPARTA) defense system, activated by invading DNA to unleash its TIR domain for NAD(P) hydrolysis, was recently identified in bacteria. We report the crystal structure of SPARTA heterodimer in the absence of guide-RNA/target-ssDNA (2.66 Å) and a cryo-EM structure of the SPARTA oligomer (tetramer of heterodimers) bound to guide-RNA/target-ssDNA at nominal 3.15-3.35 Å resolution. The crystal structure provides a high-resolution view of SPARTA, revealing the APAZ domain as equivalent to the N, L1, and L2 regions of long pAgos and the MID domain containing a unique insertion (insert57). Cryo-EM structure reveals regions of the PIWI (loop10-9) and APAZ (helix αN) domains that reconfigure for nucleic-acid binding and decrypts regions/residues that reorganize to expose a positively charged pocket for higher-order assembly. The TIR domains amass in a parallel-strands arrangement for catalysis. We visualize SPARTA before and after RNA/ssDNA binding and uncover the basis of its active assembly leading to abortive infection. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_41945.map.gz emd_41945.map.gz | 398.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-41945-v30.xml emd-41945-v30.xml emd-41945.xml emd-41945.xml | 14.7 KB 14.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_41945.png emd_41945.png | 88.2 KB | ||
| Filedesc metadata |  emd-41945.cif.gz emd-41945.cif.gz | 5.1 KB | ||
| その他 |  emd_41945_half_map_1.map.gz emd_41945_half_map_1.map.gz emd_41945_half_map_2.map.gz emd_41945_half_map_2.map.gz | 390.7 MB 390.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-41945 http://ftp.pdbj.org/pub/emdb/structures/EMD-41945 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-41945 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-41945 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_41945_validation.pdf.gz emd_41945_validation.pdf.gz | 958.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_41945_full_validation.pdf.gz emd_41945_full_validation.pdf.gz | 958.3 KB | 表示 | |
| XML形式データ |  emd_41945_validation.xml.gz emd_41945_validation.xml.gz | 17.9 KB | 表示 | |
| CIF形式データ |  emd_41945_validation.cif.gz emd_41945_validation.cif.gz | 21.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41945 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41945 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41945 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41945 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_41945.map.gz / 形式: CCP4 / 大きさ: 421.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_41945.map.gz / 形式: CCP4 / 大きさ: 421.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | lcocally refined map | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.083 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #1
| ファイル | emd_41945_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
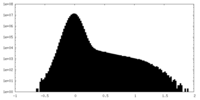
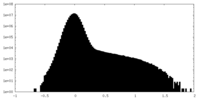
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_41945_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
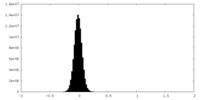
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : SPARTA complex with gRNA/tDNA
| 全体 | 名称: SPARTA complex with gRNA/tDNA |
|---|---|
| 要素 |
|
-超分子 #1: SPARTA complex with gRNA/tDNA
| 超分子 | 名称: SPARTA complex with gRNA/tDNA / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Thermoflavifilum thermophilum (バクテリア) Thermoflavifilum thermophilum (バクテリア) |
-分子 #1: Ago
| 分子 | 名称: Ago / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Thermoflavifilum thermophilum (バクテリア) Thermoflavifilum thermophilum (バクテリア) |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MKELIYIEEP SILFAHGQKC TDPRDGLALF GPLNQIYGIK SGVVGTQKGL QIFKSYLDKI QKPIYNHNN ITRPMFPGFE AVFGCKWESQ NIVFKEITDE EIRRYLFNAS THKRTYDLVT L FNDKIITA NKNDEERVDV WFVIVPEEIY KYCRPNSVLP NELVQTKSLI ...文字列: MKELIYIEEP SILFAHGQKC TDPRDGLALF GPLNQIYGIK SGVVGTQKGL QIFKSYLDKI QKPIYNHNN ITRPMFPGFE AVFGCKWESQ NIVFKEITDE EIRRYLFNAS THKRTYDLVT L FNDKIITA NKNDEERVDV WFVIVPEEIY KYCRPNSVLP NELVQTKSLI SKSKAKSFRY TP TLFEEFN KKLKEVEKEA KTYNYDAQFH DQLKARLLEH TIPTQILRES TLAWRDFKNT FGA PIRDFS KIEGHLAWTI STAAYYKAGG KPWKLGDIRP GVCYLGLVYK KIEKSKNPQN ACCA AQMFL DNGDGTVFKG EVGPWYNPEK GEYHLKPKEA KALLTQALES YKEQNKSYPK EVFIH ARTR FNDEEWNAFN EVTPKNTNLV GVTITKSKPL KLYKTEGAFP IMRGNAYIVD EKKAFL WTL GFVPKLQSTL SMEVPNPIFI EINKGEAEIQ QVLKDILALT KLNYNACIYA DGEPVTL RF ANKIGEILTA STEIKTPPLA FKYYI |
-分子 #2: TIR-APAZ
| 分子 | 名称: TIR-APAZ / タイプ: protein_or_peptide / ID: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Thermoflavifilum thermophilum (バクテリア) Thermoflavifilum thermophilum (バクテリア) |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MRNKIFISHA TPEDDDFTRW LSLKLIGLGY EVWCDILFLD KGVDFWSTIE KEIRENTCKF LIVSSTAGN KREGVLKELA VATKVKKHLQ DDMFIIPLAI DENLSYDDIN IEIVRLNAID F KKSWAKGL QDLLDAFEKQ NVPKKPPDHS KSNLLYQQIF LHDKQAIEKE ...文字列: MRNKIFISHA TPEDDDFTRW LSLKLIGLGY EVWCDILFLD KGVDFWSTIE KEIRENTCKF LIVSSTAGN KREGVLKELA VATKVKKHLQ DDMFIIPLAI DENLSYDDIN IEIVRLNAID F KKSWAKGL QDLLDAFEKQ NVPKKPPDHS KSNLLYQQIF LHDKQAIEKE ETYDSNWFPI IS FPNELRF HRYDWRLPKQ FDVRTLAFPA IRYKEYLCTF AWEYDFIHQL PKTETYNGQE SIR ISTSDI LSGRYDTDFI RNYECQRLIV QLINKAFELR MKDKNVREYQ MSKTFAYWIE KGKL EKDKF EKIKLVGKQK NKYWHFGISA AGKLYPSPVL MVSSHIIFTM DGINLIKSKS IQHSS RRKQ GKNWWNDKWR EKLLAFIRFL SDDQNAIYLN VGSEEKILIS NKPLKFFGKM SYVTPS EVT LEEESVLADI NNFEEDTEDL DELEDIE |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 糖包埋 | 材質: Vitreous ice |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 51.11 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 0.8 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: NONE |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 3.15 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 238432 |
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
 ムービー
ムービー コントローラー
コントローラー










 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)