+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | S. cerevisiae Pex1/Pex6 with 1 mM ATP | ||||||||||||
 マップデータ マップデータ | |||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | Peroxisome AAA-ATPase unfoldase / MOTOR PROTEIN | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報protein import into peroxisome matrix, receptor recycling / protein import into peroxisome matrix / protein transporter activity / peroxisomal membrane / transporter complex / ATPase complex / protein unfolding / 加水分解酵素; 酸無水物に作用; 酸無水物に作用・細胞または細胞小器官の運動に関与 / peroxisome / ATP hydrolysis activity ...protein import into peroxisome matrix, receptor recycling / protein import into peroxisome matrix / protein transporter activity / peroxisomal membrane / transporter complex / ATPase complex / protein unfolding / 加水分解酵素; 酸無水物に作用; 酸無水物に作用・細胞または細胞小器官の運動に関与 / peroxisome / ATP hydrolysis activity / ATP binding / cytosol 類似検索 - 分子機能 | ||||||||||||
| 生物種 |  | ||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.89 Å | ||||||||||||
 データ登録者 データ登録者 | Gardner BM / Richardson CD / Martin A / Lander GC / Chowdhury S | ||||||||||||
| 資金援助 |  米国, 3件 米国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: J Biol Chem / 年: 2024 ジャーナル: J Biol Chem / 年: 2024タイトル: The N1 domain of the peroxisomal AAA-ATPase Pex6 is required for Pex15 binding and proper assembly with Pex1. 著者: Bashir A Ali / Ryan M Judy / Saikat Chowdhury / Nicole K Jacobsen / Dominic T Castanzo / Kaili L Carr / Chris D Richardson / Gabriel C Lander / Andreas Martin / Brooke M Gardner /   要旨: The heterohexameric ATPases associated with diverse cellular activities (AAA)-ATPase Pex1/Pex6 is essential for the formation and maintenance of peroxisomes. Pex1/Pex6, similar to other AAA-ATPases, ...The heterohexameric ATPases associated with diverse cellular activities (AAA)-ATPase Pex1/Pex6 is essential for the formation and maintenance of peroxisomes. Pex1/Pex6, similar to other AAA-ATPases, uses the energy from ATP hydrolysis to mechanically thread substrate proteins through its central pore, thereby unfolding them. In related AAA-ATPase motors, substrates are recruited through binding to the motor's N-terminal domains or N terminally bound cofactors. Here, we use structural and biochemical techniques to characterize the function of the N1 domain in Pex6 from budding yeast, Saccharomyces cerevisiae. We found that although Pex1/ΔN1-Pex6 is an active ATPase in vitro, it does not support Pex1/Pex6 function at the peroxisome in vivo. An X-ray crystal structure of the isolated Pex6 N1 domain shows that the Pex6 N1 domain shares the same fold as the N-terminal domains of PEX1, CDC48, and NSF, despite poor sequence conservation. Integrating this structure with a cryo-EM reconstruction of Pex1/Pex6, AlphaFold2 predictions, and biochemical assays shows that Pex6 N1 mediates binding to both the peroxisomal membrane tether Pex15 and an extended loop from the D2 ATPase domain of Pex1 that influences Pex1/Pex6 heterohexamer stability. Given the direct interactions with both Pex15 and the D2 ATPase domains, the Pex6 N1 domain is poised to coordinate binding of cofactors and substrates with Pex1/Pex6 ATPase activity. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_41788.map.gz emd_41788.map.gz | 41.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-41788-v30.xml emd-41788-v30.xml emd-41788.xml emd-41788.xml | 20.7 KB 20.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_41788.png emd_41788.png | 54 KB | ||
| Filedesc metadata |  emd-41788.cif.gz emd-41788.cif.gz | 7.3 KB | ||
| その他 |  emd_41788_half_map_1.map.gz emd_41788_half_map_1.map.gz emd_41788_half_map_2.map.gz emd_41788_half_map_2.map.gz | 77.8 MB 77.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-41788 http://ftp.pdbj.org/pub/emdb/structures/EMD-41788 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-41788 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-41788 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_41788_validation.pdf.gz emd_41788_validation.pdf.gz | 1.2 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_41788_full_validation.pdf.gz emd_41788_full_validation.pdf.gz | 1.2 MB | 表示 | |
| XML形式データ |  emd_41788_validation.xml.gz emd_41788_validation.xml.gz | 12.9 KB | 表示 | |
| CIF形式データ |  emd_41788_validation.cif.gz emd_41788_validation.cif.gz | 15 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41788 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41788 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41788 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41788 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8u0vMC  8u0xC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_41788.map.gz / 形式: CCP4 / 大きさ: 83.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_41788.map.gz / 形式: CCP4 / 大きさ: 83.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.15 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #1
| ファイル | emd_41788_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
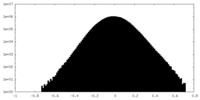
| 投影像・断面図 |
| ||||||||||||
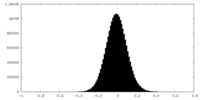
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_41788_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
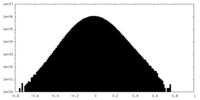
| 投影像・断面図 |
| ||||||||||||
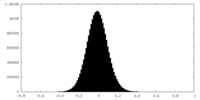
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Pex1/Pex6 AAA-ATPase
| 全体 | 名称: Pex1/Pex6 AAA-ATPase |
|---|---|
| 要素 |
|
-超分子 #1: Pex1/Pex6 AAA-ATPase
| 超分子 | 名称: Pex1/Pex6 AAA-ATPase / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 637 KDa |
-分子 #1: Peroxisomal ATPase PEX1
| 分子 | 名称: Peroxisomal ATPase PEX1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO EC番号: 加水分解酵素; 酸無水物に作用; 酸無水物に作用・細胞または細胞小器官の運動に関与 |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 118.653977 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MTTTKRLKFE NLRIQFSNAI VGNFLRLPHS IINVLESTNY AIQEFGIAVH SHNSDIPIVH LGWDGHDSGS SENVVLINPV LATVYDLNQ KSPLVDLYIQ RYDHTHLATE VYVTPETSDD WEIIDANAMR FQNGEILHQT RIVTPGETLI CYLEGIVTKF K IDRVEPSM ...文字列: MTTTKRLKFE NLRIQFSNAI VGNFLRLPHS IINVLESTNY AIQEFGIAVH SHNSDIPIVH LGWDGHDSGS SENVVLINPV LATVYDLNQ KSPLVDLYIQ RYDHTHLATE VYVTPETSDD WEIIDANAMR FQNGEILHQT RIVTPGETLI CYLEGIVTKF K IDRVEPSM KSARITDGSL VVVAPKVNKT RLVKAEYGHS NKTILKNGAI QLLKKVILRS TVCKMDFPKD NLFVVYISDG AQ LPSQKGY ASIVKCSLRQ SKKSDSDNKS VGIPSKKIGV FIKCDSQIPE NHIALSSHLW DAFFTHPMNG AKIKLEFLQM NQA NIISGR NATVNIKYFG KDVPTKSGDQ YSKLLGGSLL TNNLILPTEQ IIIEIKKGES EQQLCNLNEI SNESVQWKVT QMGK EEVKD IIERHLPKHY HVKETGEVSR TSKDEDDFIT VNSIKKEMVN YLTSPIIATP AIILDGKQGI GKTRLLKELI NEVEK DHHI FVKYADCETL HETSNLDKTQ KLIMEWCSFC YWYGPSLIVL DNVEALFGKP QANDGDPSNN GQWDNASKLL NFFINQ VTK IFNKDNKRIR VLFSGKQKTQ INPLLFDKHF VSETWSLRAP DKHARAKLLE YFFSKNQIMK LNRDLQFSDL SLETEGF SP LDLEIFTEKI FYDLQLERDC DNVVTRELFS KSLSAFTPSA LRGVKLTKET NIKWGDIGAL ANAKDVLLET LEWPTKYE P IFVNCPLRLR SGILLYGYPG CGKTLLASAV AQQCGLNFIS VKGPEILNKF IGASEQNIRE LFERAQSVKP CILFFDEFD SIAPKRGHDS TGVTDRVVNQ LLTQMDGAEG LDGVYILAAT SRPDLIDSAL LRPGRLDKSV ICNIPTESER LDILQAIVNS KDKDTGQKK FALEKNADLK LIAEKTAGFS GADLQGLCYN AYLKSVHRWL SAADQSEVVP GNDNIEYFSI NEHGRREENR L RLKTLLQQ DVVHETKTST SAASELTAVV TINDLLEACQ ETKPSISTSE LVKLRGIYDR FQKDRNGEMP NGENSIDIGS RL SLMGSSD YKDDDDK UniProtKB: Peroxisomal ATPase PEX1 |
-分子 #2: Peroxisomal ATPase PEX6
| 分子 | 名称: Peroxisomal ATPase PEX6 / タイプ: protein_or_peptide / ID: 2 / コピー数: 3 / 光学異性体: LEVO EC番号: 加水分解酵素; 酸無水物に作用; 酸無水物に作用・細胞または細胞小器官の運動に関与 |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 117.306758 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MGSSHHHHHH SQDPMKASLT FSLSGIYAPC SISRDIYLEY GDKKAECLYG TIRLPQYGPG CTPGKIVHCV LDDSLPFCSI VVPSKLFGF MPTQPTMDFC YFEPILDNVV PVLDSVTFLI NEQLYSKLMD LPQEMQQIQF LHYKYNINSM ETVVHSRDIL T SGLCQILN ...文字列: MGSSHHHHHH SQDPMKASLT FSLSGIYAPC SISRDIYLEY GDKKAECLYG TIRLPQYGPG CTPGKIVHCV LDDSLPFCSI VVPSKLFGF MPTQPTMDFC YFEPILDNVV PVLDSVTFLI NEQLYSKLMD LPQEMQQIQF LHYKYNINSM ETVVHSRDIL T SGLCQILN CSPFPQGLVD FTETQLILVN DTEQKLSALK YANEDEEYAL PKIGTNSALS IDLESLPCTI SRDLLRPAPH IN DDNSIYA FTDAETLLRL DVTSGSFITV SNMGCVRLVK LFVLLLPNGF KKRTIYAPPK IIASFPDCSV VTISKSNIGH TDI PIANQV FISRVGGWLQ SQKCFQNIIL TTLKKFFSES KRILCQNDLI PIAFDSSMAD LNIAEENDES DDEDELGQYY KNDS LVWFF VTSAELDCFS KDNSHFIIDP NRTKLITTNI TNRRPLPLSR SNLQRYYGFA ETFYYDLHIF PYVRQLVNIL ETSFN CSQR GITLNASVLL HSTTNNVGKA TMVRFASKYL GIHLLEIDCL SLTSNSRQLD STSKIIGYIR AKCENVLPYA SPAVIF LAH LDSILLDVNA NQDPEAIKLQ KSINFEMSKL LDDFTFKFPG TTFVGSVNNI DNVPSSFRSH MRFEILVPVP SEAQRLR IF QWYLSSHELN RDVQQKVPVS YMDNISFSSL SSYSAGLTPL DIKSIVETAR MTATARFYQE SKKCGWLPQS ILITQEDL S KATSKARNEF SVSIGAPQIP NVTWDDIGGI DFVKGEILDT IDMPLKHPEL FTSGMKKRSG ILFYGPPGTG KTLMAKAIA TNFSLNFFSV KGPELLNMYI GESEANVRRV FQKAREAKPC VIFFDEIDSV APKRGNQGDS GGVMDRIVSQ LLAELDGMST DADGVFVIG ATNRPDLLDE ALLRPGRFDK LLYLGIPDTD TKQLNILEAL TRKFVLDNDV KLIELAKLCP FNYTGADFYA L CSDAMLNA MSRIARMVEK KVSQHNELTG ENISTRRWFD KIATKEDTKV VVKMEDFLKA QEQLTPSVSR AELNHYEAVR AN FEGA UniProtKB: Peroxisomal ATPase PEX6 |
-分子 #3: ADENOSINE-5'-TRIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-TRIPHOSPHATE / タイプ: ligand / ID: 3 / コピー数: 10 / 式: ATP |
|---|---|
| 分子量 | 理論値: 507.181 Da |
| Chemical component information |  ChemComp-ATP: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 3.5 mg/mL | ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.6 構成要素:
| ||||||||||||||||||||||||
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 温度 | 最低: 83.0 K / 最高: 83.0 K |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 平均露光時間: 12.0 sec. / 平均電子線量: 54.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 倍率(補正後): 43478 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 1.8 µm / 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 43478 |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | Chain - Source name: AlphaFold / Chain - Initial model type: in silico model |
|---|---|
| 得られたモデル |  PDB-8u0v: |
 ムービー
ムービー コントローラー
コントローラー







 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)