+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of bovine concentrative nucleoside transporter 3 in complex with GS-441524, consensus reconstruction | |||||||||
 マップデータ マップデータ | full map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | membrane protein / transporter / nucleoside / TRANSPORT PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Ribavirin ADME / purine-specific nucleoside:sodium symporter activity / Transport of nucleosides and free purine and pyrimidine bases across the plasma membrane / pyrimidine nucleoside transport / pyrimidine- and adenosine-specific:sodium symporter activity / purine nucleoside transmembrane transport / uridine transmembrane transporter activity / Azathioprine ADME / brush border membrane / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.31 Å | |||||||||
 データ登録者 データ登録者 | Wright NJ / Lee S-Y | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Chem Biol / 年: 2024 ジャーナル: Nat Chem Biol / 年: 2024タイトル: Antiviral drug recognition and elevator-type transport motions of CNT3. 著者: Nicholas J Wright / Feng Zhang / Yang Suo / Lingyang Kong / Ying Yin / Justin G Fedor / Kedar Sharma / Mario J Borgnia / Wonpil Im / Seok-Yong Lee /  要旨: Nucleoside analogs have broad clinical utility as antiviral drugs. Key to their systemic distribution and cellular entry are human nucleoside transporters. Here, we establish that the human ...Nucleoside analogs have broad clinical utility as antiviral drugs. Key to their systemic distribution and cellular entry are human nucleoside transporters. Here, we establish that the human concentrative nucleoside transporter 3 (CNT3) interacts with antiviral drugs used in the treatment of coronavirus infections. We report high-resolution single-particle cryo-electron microscopy structures of bovine CNT3 complexed with antiviral nucleosides N-hydroxycytidine, PSI-6206, GS-441524 and ribavirin, all in inward-facing states. Notably, we found that the orally bioavailable antiviral molnupiravir arrests CNT3 in four distinct conformations, allowing us to capture cryo-electron microscopy structures of drug-loaded outward-facing and drug-loaded intermediate states. Our studies uncover the conformational trajectory of CNT3 during membrane transport of a nucleoside analog antiviral drug, yield new insights into the role of interactions between the transport and the scaffold domains in elevator-like domain movements during drug translocation, and provide insights into the design of nucleoside analog antiviral prodrugs with improved oral bioavailability. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_41732.map.gz emd_41732.map.gz | 59.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-41732-v30.xml emd-41732-v30.xml emd-41732.xml emd-41732.xml | 18.9 KB 18.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_41732.png emd_41732.png | 83.7 KB | ||
| Filedesc metadata |  emd-41732.cif.gz emd-41732.cif.gz | 6.6 KB | ||
| その他 |  emd_41732_half_map_1.map.gz emd_41732_half_map_1.map.gz emd_41732_half_map_2.map.gz emd_41732_half_map_2.map.gz | 59.5 MB 59.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-41732 http://ftp.pdbj.org/pub/emdb/structures/EMD-41732 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-41732 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-41732 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8tz3MC  8tz1C  8tz2C  8tz4C  8tz5C  8tz6C  8tz7C  8tz8C  8tz9C  8tzaC  8tzdC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_41732.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_41732.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | full map | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.08 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: half map 1
| ファイル | emd_41732_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
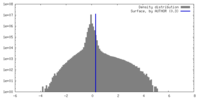
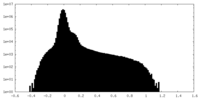
| 密度ヒストグラム |
-ハーフマップ: half map 2
| ファイル | emd_41732_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
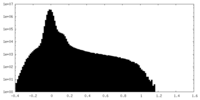
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : bCNT3 trimer
| 全体 | 名称: bCNT3 trimer |
|---|---|
| 要素 |
|
-超分子 #1: bCNT3 trimer
| 超分子 | 名称: bCNT3 trimer / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 232 KDa |
-分子 #1: Sodium/nucleoside cotransporter
| 分子 | 名称: Sodium/nucleoside cotransporter / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 79.376773 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MDYKDDDDKL EATMAMSSKI SVELQRVAAL PAQGCSNTGF QNDEDGFENQ NPSGNDHSLR NRVVQNREHE NGKQVEEHIT IGQDSLRKD EEEEDDQETH RKGCLERMCG RMSDFCREHK TTLRYIIWGI LIAGYLALVI AACVMNFHRA LPLFVITVVA I FFVVWDHL ...文字列: MDYKDDDDKL EATMAMSSKI SVELQRVAAL PAQGCSNTGF QNDEDGFENQ NPSGNDHSLR NRVVQNREHE NGKQVEEHIT IGQDSLRKD EEEEDDQETH RKGCLERMCG RMSDFCREHK TTLRYIIWGI LIAGYLALVI AACVMNFHRA LPLFVITVVA I FFVVWDHL MAKYESQIAR FLSPGQRLLD SHWFWLKWVI WGCLILGVIL WLVFDTAKLG QQQLVSFGGL IIYTSLTFLF SK HPTKVYW RPVFWGIGLQ FLLGLLILRT EPGFMAFDWL GKQVQTFLGY SDAGASFVFG EKYTDHFFAF KVLPIVIFFS TVM SMLYYL GLMQWIIRKV GWVMLVTMGT SPVESVVASG NIFIGQTESP LLVRPYLPYV TKSELHAIMT AGFSTIAGSV LGAY ISFGV SSSHLLTASV MSAPAALAIS KLFWPETETP KINLKNAMKM ESGDSRNLLE AATQGASSSI SLVANIAVNL IAFLA LLSF MNSALSWLGN MFDYPQLSFE VICSYVFMPF AFMMGVDWQD SFMVAKLIGY KTFFNEFVAY QQLSKLISLR QVGGPK FVD GVQQYMSMRS EAISTYALCG FANFGSLGIV IGGLTSMAPS RKRDITAGAM RALIAGTIAC FLTACIAGML TNTPVDI NC HHILENAFNS GLVRNTTNVV SCCQGLLSSA VVKGPGEVIP TGNHSLYSLK NCCNLLNTPT LNCSWIPNVL SNS UniProtKB: Sodium/nucleoside cotransporter |
-分子 #2: SODIUM ION
| 分子 | 名称: SODIUM ION / タイプ: ligand / ID: 2 / コピー数: 6 |
|---|---|
| 分子量 | 理論値: 22.99 Da |
-分子 #3: 1-palmitoyl-2-oleoyl-sn-glycero-3-phosphocholine
| 分子 | 名称: 1-palmitoyl-2-oleoyl-sn-glycero-3-phosphocholine / タイプ: ligand / ID: 3 / コピー数: 12 / 式: LBN |
|---|---|
| 分子量 | 理論値: 760.076 Da |
| Chemical component information |  ChemComp-LBN: |
-分子 #4: water
| 分子 | 名称: water / タイプ: ligand / ID: 4 / コピー数: 15 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 8 mg/mL | ||||||
|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 / 構成要素:
| ||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / チャンバー内温度: 277 K / 装置: LEICA EM GP |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) デジタル化 - サイズ - 横: 5760 pixel / デジタル化 - サイズ - 縦: 4092 pixel / 撮影したグリッド数: 1 / 実像数: 12707 / 平均露光時間: 2.4 sec. / 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 1.8 µm / 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 81000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー


























 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)