+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Automethylated PRC2 dimer bound to nucleosome | |||||||||
 マップデータ マップデータ | Automethylated PRC2 dimer bound to nucleosome 3DFlex reconstruction locally filtered | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Histone methyl transferase / gene repression / epigenetics / chromatin / GENE REGULATION | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報regulation of kidney development / hepatocyte homeostasis / cellular response to trichostatin A / regulation of gliogenesis / negative regulation of striated muscle cell differentiation / [histone H3]-lysine27 N-trimethyltransferase / sex chromatin / negative regulation of keratinocyte differentiation / random inactivation of X chromosome / CAF-1 complex ...regulation of kidney development / hepatocyte homeostasis / cellular response to trichostatin A / regulation of gliogenesis / negative regulation of striated muscle cell differentiation / [histone H3]-lysine27 N-trimethyltransferase / sex chromatin / negative regulation of keratinocyte differentiation / random inactivation of X chromosome / CAF-1 complex / histone H3K27 trimethyltransferase activity / negative regulation of retinoic acid receptor signaling pathway / regulatory ncRNA-mediated heterochromatin formation / skeletal muscle satellite cell maintenance involved in skeletal muscle regeneration / response to tetrachloromethane / cerebellar cortex development / primary miRNA binding / histone H3K27 methyltransferase activity / facultative heterochromatin formation / positive regulation of cell cycle G1/S phase transition / NURF complex / NuRD complex / regulation of cell fate specification / negative regulation of stem cell population maintenance / DNA replication-dependent chromatin assembly / ESC/E(Z) complex / chromatin silencing complex / Transcription of E2F targets under negative control by p107 (RBL1) and p130 (RBL2) in complex with HDAC1 / regulation of stem cell differentiation / RSC-type complex / protein-lysine N-methyltransferase activity / cardiac muscle hypertrophy in response to stress / negative regulation of stem cell differentiation / Transcription of E2F targets under negative control by DREAM complex / pronucleus / Polo-like kinase mediated events / synaptic transmission, GABAergic / positive regulation of dendrite development / histone H3 methyltransferase activity / lncRNA binding / negative regulation of G1/S transition of mitotic cell cycle / negative regulation of gene expression, epigenetic / G1 to G0 transition / spinal cord development / positive regulation of stem cell population maintenance / ATPase complex / G1/S-Specific Transcription / histone methyltransferase activity / Sin3-type complex / oligodendrocyte differentiation / negative regulation of transcription elongation by RNA polymerase II / Transcriptional Regulation by E2F6 / RNA Polymerase I Transcription Initiation / negative regulation of cell differentiation / histone deacetylase complex / G0 and Early G1 / subtelomeric heterochromatin formation / negative regulation of cytokine production involved in inflammatory response / RNA polymerase II core promoter sequence-specific DNA binding / pericentric heterochromatin / ribonucleoprotein complex binding / Cyclin E associated events during G1/S transition / Transcriptional regulation of brown and beige adipocyte differentiation by EBF2 / positive regulation of epithelial to mesenchymal transition / Cyclin A:Cdk2-associated events at S phase entry / keratinocyte differentiation / Deposition of new CENPA-containing nucleosomes at the centromere / enzyme activator activity / Regulation of TP53 Activity through Acetylation / protein localization to chromatin / methylated histone binding / B cell differentiation / SUMOylation of chromatin organization proteins / transcription corepressor binding / ERCC6 (CSB) and EHMT2 (G9a) positively regulate rRNA expression / positive regulation of GTPase activity / negative regulation of cell migration / PRC2 methylates histones and DNA / Regulation of PTEN gene transcription / stem cell differentiation / Defective pyroptosis / HDACs deacetylate histones / liver regeneration / promoter-specific chromatin binding / hippocampus development / transcription coregulator activity / negative regulation of transforming growth factor beta receptor signaling pathway / positive regulation of MAP kinase activity / protein modification process / positive regulation of protein serine/threonine kinase activity / brain development / regulation of circadian rhythm / heterochromatin formation / chromatin DNA binding / PKMTs methylate histone lysines / histone deacetylase binding / Activation of anterior HOX genes in hindbrain development during early embryogenesis / HCMV Early Events / cellular response to hydrogen peroxide / G1/S transition of mitotic cell cycle 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) / Homo sapiens (ヒト) / | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 6.2 Å | |||||||||
 データ登録者 データ登録者 | Sauer PV / Pavlenko E / Nogales E / Poepsel S | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Mol Cell / 年: 2024 ジャーナル: Mol Cell / 年: 2024タイトル: Activation of automethylated PRC2 by dimerization on chromatin. 著者: Paul V Sauer / Egor Pavlenko / Trinity Cookis / Linda C Zirden / Juliane Renn / Ankush Singhal / Pascal Hunold / Michaela N Hoehne-Wiechmann / Olivia van Ray / Farnusch Kaschani / Markus ...著者: Paul V Sauer / Egor Pavlenko / Trinity Cookis / Linda C Zirden / Juliane Renn / Ankush Singhal / Pascal Hunold / Michaela N Hoehne-Wiechmann / Olivia van Ray / Farnusch Kaschani / Markus Kaiser / Robert Hänsel-Hertsch / Karissa Y Sanbonmatsu / Eva Nogales / Simon Poepsel /   要旨: Polycomb repressive complex 2 (PRC2) is an epigenetic regulator that trimethylates lysine 27 of histone 3 (H3K27me3) and is essential for embryonic development and cellular differentiation. H3K27me3 ...Polycomb repressive complex 2 (PRC2) is an epigenetic regulator that trimethylates lysine 27 of histone 3 (H3K27me3) and is essential for embryonic development and cellular differentiation. H3K27me3 is associated with transcriptionally repressed chromatin and is established when PRC2 is allosterically activated upon methyl-lysine binding by the regulatory subunit EED. Automethylation of the catalytic subunit enhancer of zeste homolog 2 (EZH2) stimulates its activity by an unknown mechanism. Here, we show that human PRC2 forms a dimer on chromatin in which an inactive, automethylated PRC2 protomer is the allosteric activator of a second PRC2 that is poised to methylate H3 of a substrate nucleosome. Functional assays support our model of allosteric trans-autoactivation via EED, suggesting a previously unknown mechanism mediating context-dependent activation of PRC2. Our work showcases the molecular mechanism of auto-modification-coupled dimerization in the regulation of chromatin-modifying complexes. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_41110.map.gz emd_41110.map.gz | 1.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-41110-v30.xml emd-41110-v30.xml emd-41110.xml emd-41110.xml | 46.2 KB 46.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_41110.png emd_41110.png | 41.5 KB | ||
| マスクデータ |  emd_41110_msk_1.map emd_41110_msk_1.map | 30.5 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-41110.cif.gz emd-41110.cif.gz | 9.5 KB | ||
| その他 |  emd_41110_additional_1.map.gz emd_41110_additional_1.map.gz emd_41110_additional_2.map.gz emd_41110_additional_2.map.gz emd_41110_additional_3.map.gz emd_41110_additional_3.map.gz emd_41110_additional_4.map.gz emd_41110_additional_4.map.gz emd_41110_additional_5.map.gz emd_41110_additional_5.map.gz emd_41110_additional_6.map.gz emd_41110_additional_6.map.gz emd_41110_half_map_1.map.gz emd_41110_half_map_1.map.gz emd_41110_half_map_2.map.gz emd_41110_half_map_2.map.gz | 67.8 MB 1.2 MB 1.2 MB 1.2 MB 474.5 KB 1.1 MB 127.2 MB 127.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-41110 http://ftp.pdbj.org/pub/emdb/structures/EMD-41110 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-41110 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-41110 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_41110_validation.pdf.gz emd_41110_validation.pdf.gz | 766.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_41110_full_validation.pdf.gz emd_41110_full_validation.pdf.gz | 766.1 KB | 表示 | |
| XML形式データ |  emd_41110_validation.xml.gz emd_41110_validation.xml.gz | 13.4 KB | 表示 | |
| CIF形式データ |  emd_41110_validation.cif.gz emd_41110_validation.cif.gz | 15.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41110 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41110 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41110 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41110 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_41110.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_41110.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Automethylated PRC2 dimer bound to nucleosome 3DFlex reconstruction locally filtered | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.3 Å | ||||||||||||||||||||||||||||||||||||
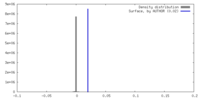
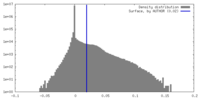
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_41110_msk_1.map emd_41110_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
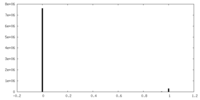
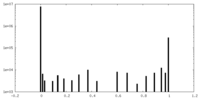
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
+全体 : Automethylated PRC2 dimer bound to nucleosome
+超分子 #1: Automethylated PRC2 dimer bound to nucleosome
+超分子 #2: Nucleosome
+超分子 #3: Proximal PRC2
+超分子 #4: Distal PRC2
+分子 #1: Polycomb protein SUZ12
+分子 #2: Histone-lysine N-methyltransferase EZH2
+分子 #3: Polycomb protein EED
+分子 #4: Histone-binding protein RBBP4
+分子 #5: Zinc finger protein AEBP2
+分子 #6: Histone H3.2
+分子 #8: Histone H4
+分子 #9: Histone H2A type 1
+分子 #10: Histone H2B 1.1
+分子 #12: activating methylated peptide
+分子 #7: DNA (226-MER)
+分子 #11: DNA (226-MER)
+分子 #13: S-ADENOSYL-L-HOMOCYSTEINE
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.8 |
|---|---|
| グリッド | 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 詳細: Streptavidin affinity grid |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / 平均電子線量: 40.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 3.5 µm / 最小 デフォーカス(公称値): 1.5 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: EMDB MAP EMDB ID: |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 6.2 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 34000 |
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT |
|---|---|
| 得られたモデル |  PDB-8t9g: |
 ムービー
ムービー コントローラー
コントローラー





























 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)
























 Trichoplusia ni (イラクサキンウワバ)
Trichoplusia ni (イラクサキンウワバ)


