[English] 日本語
 Yorodumi
Yorodumi- EMDB-40624: Chlorella virus Hyaluronan Synthase bound to GlcA extended GlcNAc... -
+ Open data
Open data
- Basic information
Basic information
| Entry |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| Title | Chlorella virus Hyaluronan Synthase bound to GlcA extended GlcNAc primer and UDP | |||||||||
 Map data Map data | Chlorella virus Hyaluronan Synthase bound to GlcA extended GlcNAc primer and UDP | |||||||||
 Sample Sample |
| |||||||||
 Keywords Keywords | hyaluronic acid / hyaluronan / HA / HAS / glycosyltransferase / GT / membrane protein / nanobody / n-acetylglucosamine / glucuronic acid / TRANSFERASE | |||||||||
| Function / homology | hyaluronan synthase activity / Glycosyltransferase like family 2 / hyaluronan biosynthetic process / Nucleotide-diphospho-sugar transferases / plasma membrane / Hyaluronan synthase Function and homology information Function and homology information | |||||||||
| Biological species |   Paramecium bursaria Chlorella virus 1 / Paramecium bursaria Chlorella virus 1 /  Paramecium bursaria Chlorella virus CZ-2 / Paramecium bursaria Chlorella virus CZ-2 /  | |||||||||
| Method | single particle reconstruction / cryo EM / Resolution: 3.0 Å | |||||||||
 Authors Authors | Stephens Z / Zimmer J | |||||||||
| Funding support |  United States, 2 items United States, 2 items
| |||||||||
 Citation Citation |  Journal: Nat Struct Mol Biol / Year: 2025 Journal: Nat Struct Mol Biol / Year: 2025Title: Structural insights into translocation and tailored synthesis of hyaluronan. Authors: Ireneusz Górniak / Zachery Stephens / Satchal K Erramilli / Tomasz Gawda / Anthony A Kossiakoff / Jochen Zimmer /  Abstract: Hyaluronan (HA) is an essential component of the vertebrate extracellular matrix. It is a heteropolysaccharide of N-acetylglucosamine (GlcNAc) and glucuronic acid (GlcA) reaching several megadaltons ...Hyaluronan (HA) is an essential component of the vertebrate extracellular matrix. It is a heteropolysaccharide of N-acetylglucosamine (GlcNAc) and glucuronic acid (GlcA) reaching several megadaltons in healthy tissues. HA is synthesized and translocated in a coupled reaction by HA synthase (HAS). Here, structural snapshots of HAS provide insights into HA biosynthesis, from substrate recognition to HA elongation and translocation. We monitor the extension of a GlcNAc primer with GlcA, reveal the coordination of the uridine diphosphate product by a conserved gating loop and capture the opening of a translocation channel to coordinate a translocating HA polymer. Furthermore, we identify channel-lining residues that modulate HA product lengths. Integrating structural and biochemical analyses suggests an avenue for polysaccharide engineering based on finely tuned enzymatic activity and HA coordination. | |||||||||
| History |
|
- Structure visualization
Structure visualization
| Supplemental images |
|---|
- Downloads & links
Downloads & links
-EMDB archive
| Map data |  emd_40624.map.gz emd_40624.map.gz | 59.6 MB |  EMDB map data format EMDB map data format | |
|---|---|---|---|---|
| Header (meta data) |  emd-40624-v30.xml emd-40624-v30.xml emd-40624.xml emd-40624.xml | 18.1 KB 18.1 KB | Display Display |  EMDB header EMDB header |
| Images |  emd_40624.png emd_40624.png | 32.5 KB | ||
| Filedesc metadata |  emd-40624.cif.gz emd-40624.cif.gz | 7 KB | ||
| Others |  emd_40624_half_map_1.map.gz emd_40624_half_map_1.map.gz emd_40624_half_map_2.map.gz emd_40624_half_map_2.map.gz | 59.1 MB 59.1 MB | ||
| Archive directory |  http://ftp.pdbj.org/pub/emdb/structures/EMD-40624 http://ftp.pdbj.org/pub/emdb/structures/EMD-40624 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-40624 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-40624 | HTTPS FTP |
-Related structure data
| Related structure data |  8sneMC  8smmC  8smnC  8smpC  8sncC  8sndC M: atomic model generated by this map C: citing same article ( |
|---|---|
| Similar structure data | Similarity search - Function & homology  F&H Search F&H Search |
- Links
Links
| EMDB pages |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| Related items in Molecule of the Month |
- Map
Map
| File |  Download / File: emd_40624.map.gz / Format: CCP4 / Size: 64 MB / Type: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) Download / File: emd_40624.map.gz / Format: CCP4 / Size: 64 MB / Type: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Annotation | Chlorella virus Hyaluronan Synthase bound to GlcA extended GlcNAc primer and UDP | ||||||||||||||||||||||||||||||||||||
| Projections & slices | Image control
Images are generated by Spider. | ||||||||||||||||||||||||||||||||||||
| Voxel size | X=Y=Z: 1.08 Å | ||||||||||||||||||||||||||||||||||||
| Density |
| ||||||||||||||||||||||||||||||||||||
| Symmetry | Space group: 1 | ||||||||||||||||||||||||||||||||||||
| Details | EMDB XML:
|
-Supplemental data
-Half map: Half Map 1
| File | emd_40624_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Annotation | Half Map 1 | ||||||||||||
| Projections & Slices |
| ||||||||||||
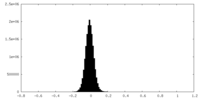
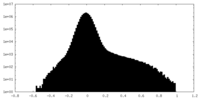
| Density Histograms |
-Half map: Half Map 2
| File | emd_40624_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Annotation | Half Map 2 | ||||||||||||
| Projections & Slices |
| ||||||||||||
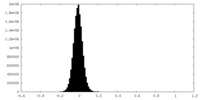
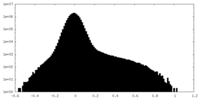
| Density Histograms |
- Sample components
Sample components
-Entire : Ternary complex of Chlorella virus hyaluronan synthase bound to n...
| Entire | Name: Ternary complex of Chlorella virus hyaluronan synthase bound to nanobodies 872 and 886 |
|---|---|
| Components |
|
-Supramolecule #1: Ternary complex of Chlorella virus hyaluronan synthase bound to n...
| Supramolecule | Name: Ternary complex of Chlorella virus hyaluronan synthase bound to nanobodies 872 and 886 type: complex / ID: 1 / Parent: 0 / Macromolecule list: #1-#3 |
|---|---|
| Source (natural) | Organism:   Paramecium bursaria Chlorella virus 1 Paramecium bursaria Chlorella virus 1 |
| Molecular weight | Theoretical: 95.3 kDa/nm |
-Macromolecule #1: Hyaluronan synthase
| Macromolecule | Name: Hyaluronan synthase / type: protein_or_peptide / ID: 1 / Number of copies: 1 / Enantiomer: LEVO |
|---|---|
| Source (natural) | Organism:  Paramecium bursaria Chlorella virus CZ-2 Paramecium bursaria Chlorella virus CZ-2 |
| Molecular weight | Theoretical: 65.890062 KDa |
| Recombinant expression | Organism:  |
| Sequence | String: MGTSWRTIVS ANLFAVGGAL LMLAPAIVGY VFQWNIGVSA VWGISVYGVF VLGFYIAQIV FSEFNRMRLS DWISLRPDNW NATRVAVII AGYREDPFMF KKCLESVRDS EYGNVARLIC VIDGDEEEDL KMAEIYKQVY NDNVKKPGVV LCESENKNGS T IDSDVSKN ...String: MGTSWRTIVS ANLFAVGGAL LMLAPAIVGY VFQWNIGVSA VWGISVYGVF VLGFYIAQIV FSEFNRMRLS DWISLRPDNW NATRVAVII AGYREDPFMF KKCLESVRDS EYGNVARLIC VIDGDEEEDL KMAEIYKQVY NDNVKKPGVV LCESENKNGS T IDSDVSKN ICILQPHRGK RESLYTGFQL ASMDPSVHAV VLIDSDTVLE KNAILEVVYP LSCDPNIKAV AGECKIWNTD TI LSMLVSW RYFSAFNVER GAQSLWKTVQ CVGGPLGAYT IDIINEIKDP WITQTFLGNK CTYGDDRRLT NEVLMRGKKI VYT PFAVGW SDSPTNVMRY IVQQTRWSKS WCREIWYTLG SAWKHGFSGI YLAFECMYQI MYFFLVMYLF SYIAIKADIR AQTA TVLVS TLVTIIKSSY LALRAKNLKA FYFVLYTYVY FFCMIPARIT AMFTMFDIAW GTRGGNAKMT IGARVWLWAK QFLIT YMWW AGVLAAGVYS IVDNWYFDWA DIQYRFALVG ICSYLVFVSI VLVIYLIGKI TTWNYTPLQK ELIEERYLHN ASENAP EVL EHHHHHHHHH H UniProtKB: Hyaluronan synthase |
-Macromolecule #2: Nanobody 872
| Macromolecule | Name: Nanobody 872 / type: protein_or_peptide / ID: 2 / Number of copies: 1 / Enantiomer: LEVO |
|---|---|
| Source (natural) | Organism:  |
| Molecular weight | Theoretical: 14.783248 KDa |
| Recombinant expression | Organism:  |
| Sequence | String: QVQLVESGGG LVQAGGSLKV SCAASGRAFK TYRMAWFRQA PGKEREFVSG ISALETTYYA DSVKGRFTIS RDNTKNTVSL QMDSLKPED TAVYYCAARR YGGTDYTTTG SYDYWGQGTQ VTVSSHHHHH HEPEA |
-Macromolecule #3: Nanobody 886
| Macromolecule | Name: Nanobody 886 / type: protein_or_peptide / ID: 3 / Number of copies: 1 / Enantiomer: LEVO |
|---|---|
| Source (natural) | Organism:  |
| Molecular weight | Theoretical: 14.737267 KDa |
| Recombinant expression | Organism:  |
| Sequence | String: QVQLVESGGG SVQPGESLRL SCQASGRIVD VNDMAWYRQA PGKQRELVAR IARGGSTHYG DSAWGRFTIS RDNTRNTVYL QMTSLNVED TAVYYCNGEV KVGTRLSPFR TYWGRGTQVT VSSHHHHHHE PEA |
-Macromolecule #5: URIDINE-5'-DIPHOSPHATE
| Macromolecule | Name: URIDINE-5'-DIPHOSPHATE / type: ligand / ID: 5 / Number of copies: 1 / Formula: UDP |
|---|---|
| Molecular weight | Theoretical: 404.161 Da |
| Chemical component information |  ChemComp-UDP: |
-Macromolecule #6: CHOLESTEROL HEMISUCCINATE
| Macromolecule | Name: CHOLESTEROL HEMISUCCINATE / type: ligand / ID: 6 / Number of copies: 1 / Formula: Y01 |
|---|---|
| Molecular weight | Theoretical: 486.726 Da |
| Chemical component information |  ChemComp-Y01: |
-Macromolecule #7: 1,2-Distearoyl-sn-glycerophosphoethanolamine
| Macromolecule | Name: 1,2-Distearoyl-sn-glycerophosphoethanolamine / type: ligand / ID: 7 / Number of copies: 1 / Formula: 3PE |
|---|---|
| Molecular weight | Theoretical: 748.065 Da |
| Chemical component information |  ChemComp-3PE: |
-Macromolecule #8: MANGANESE (II) ION
| Macromolecule | Name: MANGANESE (II) ION / type: ligand / ID: 8 / Number of copies: 2 / Formula: MN |
|---|---|
| Molecular weight | Theoretical: 54.938 Da |
-Experimental details
-Structure determination
| Method | cryo EM |
|---|---|
 Processing Processing | single particle reconstruction |
| Aggregation state | particle |
- Sample preparation
Sample preparation
| Buffer | pH: 7.5 |
|---|---|
| Vitrification | Cryogen name: ETHANE |
- Electron microscopy
Electron microscopy
| Microscope | FEI TITAN KRIOS |
|---|---|
| Image recording | Film or detector model: GATAN K3 (6k x 4k) / Average electron dose: 50.0 e/Å2 |
| Electron beam | Acceleration voltage: 300 kV / Electron source:  FIELD EMISSION GUN FIELD EMISSION GUN |
| Electron optics | C2 aperture diameter: 100.0 µm / Illumination mode: FLOOD BEAM / Imaging mode: BRIGHT FIELD / Cs: 2.7 mm / Nominal defocus max: 2.0 µm / Nominal defocus min: 1.0 µm / Nominal magnification: 81000 |
| Sample stage | Cooling holder cryogen: NITROGEN |
| Experimental equipment |  Model: Titan Krios / Image courtesy: FEI Company |
- Image processing
Image processing
| Startup model | Type of model: PDB ENTRY PDB model - PDB ID: |
|---|---|
| Final reconstruction | Resolution.type: BY AUTHOR / Resolution: 3.0 Å / Resolution method: FSC 0.143 CUT-OFF / Number images used: 220556 |
| Initial angle assignment | Type: MAXIMUM LIKELIHOOD |
| Final angle assignment | Type: MAXIMUM LIKELIHOOD |
 Movie
Movie Controller
Controller










 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)