+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-4046 | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of the bacterial sex F pilus (12.5 Angstrom rise) | ||||||||||||
 マップデータ マップデータ | |||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | F-pilus Conjugation Type IV secretion system Phospholipid / Protein fibril | ||||||||||||
| 機能・相同性 | TraA / TraA / extracellular region / plasma membrane / Pilin / Pilin 機能・相同性情報 機能・相同性情報 | ||||||||||||
| 生物種 |  | ||||||||||||
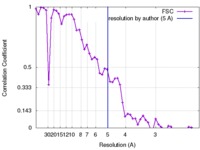
| 手法 | らせん対称体再構成法 / クライオ電子顕微鏡法 / 解像度: 5.0 Å | ||||||||||||
 データ登録者 データ登録者 | Costa TRD / Ilangovan I | ||||||||||||
| 資金援助 |  英国, 英国,  米国, 3件 米国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Cell / 年: 2016 ジャーナル: Cell / 年: 2016タイトル: Structure of the Bacterial Sex F Pilus Reveals an Assembly of a Stoichiometric Protein-Phospholipid Complex. 著者: Tiago R D Costa / Aravindan Ilangovan / Marta Ukleja / Adam Redzej / Joanne M Santini / Terry K Smith / Edward H Egelman / Gabriel Waksman /   要旨: Conjugative pili are widespread bacterial appendages that play important roles in horizontal gene transfer, in spread of antibiotic resistance genes, and as sites of phage attachment. Among ...Conjugative pili are widespread bacterial appendages that play important roles in horizontal gene transfer, in spread of antibiotic resistance genes, and as sites of phage attachment. Among conjugative pili, the F "sex" pilus encoded by the F plasmid is the best functionally characterized, and it is also historically the most important, as the discovery of F-plasmid-mediated conjugation ushered in the era of molecular biology and genetics. Yet, its structure is unknown. Here, we present atomic models of two F family pili, the F and pED208 pili, generated from cryoelectron microscopy reconstructions at 5.0 and 3.6 Å resolution, respectively. These structures reveal that conjugative pili are assemblies of stoichiometric protein-phospholipid units. We further demonstrate that each pilus type binds preferentially to particular phospholipids. These structures provide the molecular basis for F pilus assembly and also shed light on the remarkable properties of conjugative pili in bacterial secretion and phage infection. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_4046.map.gz emd_4046.map.gz | 2.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-4046-v30.xml emd-4046-v30.xml emd-4046.xml emd-4046.xml | 10.6 KB 10.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_4046_fsc.xml emd_4046_fsc.xml | 4.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_4046.png emd_4046.png | 305 KB | ||
| Filedesc metadata |  emd-4046.cif.gz emd-4046.cif.gz | 4.8 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-4046 http://ftp.pdbj.org/pub/emdb/structures/EMD-4046 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4046 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4046 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_4046_validation.pdf.gz emd_4046_validation.pdf.gz | 257.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_4046_full_validation.pdf.gz emd_4046_full_validation.pdf.gz | 257 KB | 表示 | |
| XML形式データ |  emd_4046_validation.xml.gz emd_4046_validation.xml.gz | 7.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4046 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4046 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4046 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4046 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_4046.map.gz / 形式: CCP4 / 大きさ: 16 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_4046.map.gz / 形式: CCP4 / 大きさ: 16 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 これらの図は立方格子座標系で作成されたものです | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.1 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : F-pilus filament made by an assembly of a stoichiometric protein-...
| 全体 | 名称: F-pilus filament made by an assembly of a stoichiometric protein-phospholipid complex |
|---|---|
| 要素 |
|
-超分子 #1: F-pilus filament made by an assembly of a stoichiometric protein-...
| 超分子 | 名称: F-pilus filament made by an assembly of a stoichiometric protein-phospholipid complex タイプ: organelle_or_cellular_component / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 7.3 kDa/nm |
-分子 #1: Pilin
| 分子 | 名称: Pilin / タイプ: protein_or_peptide / ID: 1 / コピー数: 75 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 6.831216 KDa |
| 配列 | 文字列: QDLMASGNTT VKATFGKDSS VVKWVVLAEV LVGAVMYMMT KNVKFLAGFA IISVFIAVGM AVVGL UniProtKB: Pilin |
-分子 #2: [(2~{S})-3-[[(2~{R})-2,3-bis(oxidanyl)propoxy]-oxidanyl-phosphory...
| 分子 | 名称: [(2~{S})-3-[[(2~{R})-2,3-bis(oxidanyl)propoxy]-oxidanyl-phosphoryl]oxy-2-hexadec-9-enoyloxy-propyl] hexadecanoate タイプ: ligand / ID: 2 / コピー数: 75 / 式: 6V6 |
|---|---|
| 分子量 | 理論値: 720.954 Da |
| Chemical component information |  ChemComp-6V6: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | helical array |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| グリッド | モデル: Lacey / 材質: COPPER / メッシュ: 400 |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI POLARA 300 |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均電子線量: 1.67 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Tecnai Polara / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 精密化 | プロトコル: AB INITIO MODEL |
|---|---|
| 得られたモデル |  PDB-5lfb: |
 ムービー
ムービー コントローラー
コントローラー














 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)