+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | SARS-CoV-2 DMV nsp3-4 pore complex (consensus-pore, C6 symmetry) | |||||||||
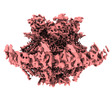
 マップデータ マップデータ | local resolution map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Double membrane vesicle / pore complex / nsp3 / nsp4 / RNA transport / VIRAL PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報protein guanylyltransferase activity / RNA endonuclease activity producing 3'-phosphomonoesters, hydrolytic mechanism / mRNA guanylyltransferase activity / 5'-3' RNA helicase activity / 付加脱離酵素(リアーゼ); P-Oリアーゼ類; - / Assembly of the SARS-CoV-2 Replication-Transcription Complex (RTC) / symbiont-mediated suppression of host cytoplasmic pattern recognition receptor signaling pathway via inhibition of TBK1 activity / Maturation of replicase proteins / ISG15-specific peptidase activity / TRAF3-dependent IRF activation pathway ...protein guanylyltransferase activity / RNA endonuclease activity producing 3'-phosphomonoesters, hydrolytic mechanism / mRNA guanylyltransferase activity / 5'-3' RNA helicase activity / 付加脱離酵素(リアーゼ); P-Oリアーゼ類; - / Assembly of the SARS-CoV-2 Replication-Transcription Complex (RTC) / symbiont-mediated suppression of host cytoplasmic pattern recognition receptor signaling pathway via inhibition of TBK1 activity / Maturation of replicase proteins / ISG15-specific peptidase activity / TRAF3-dependent IRF activation pathway / Transcription of SARS-CoV-2 sgRNAs / Translation of Replicase and Assembly of the Replication Transcription Complex / snRNP Assembly / Replication of the SARS-CoV-2 genome / 加水分解酵素; エステル加水分解酵素; 5'-リン酸モノエステル産生エキソリボヌクレアーゼ / double membrane vesicle viral factory outer membrane / host cell endoplasmic reticulum-Golgi intermediate compartment / SARS coronavirus main proteinase / 5'-3' DNA helicase activity / 3'-5'-RNA exonuclease activity / host cell endosome / symbiont-mediated degradation of host mRNA / mRNA guanylyltransferase / symbiont-mediated suppression of host ISG15-protein conjugation / G-quadruplex RNA binding / symbiont-mediated suppression of host toll-like receptor signaling pathway / symbiont-mediated suppression of host cytoplasmic pattern recognition receptor signaling pathway via inhibition of IRF3 activity / omega peptidase activity / mRNA (guanine-N7)-methyltransferase / SARS-CoV-2 modulates host translation machinery / methyltransferase cap1 / host cell Golgi apparatus / symbiont-mediated suppression of host NF-kappaB cascade / symbiont-mediated perturbation of host ubiquitin-like protein modification / DNA helicase / methyltransferase cap1 activity / ubiquitinyl hydrolase 1 / mRNA 5'-cap (guanine-N7-)-methyltransferase activity / cysteine-type deubiquitinase activity / 加水分解酵素; プロテアーゼ; ペプチド結合加水分解酵素; システインプロテアーゼ / single-stranded RNA binding / host cell perinuclear region of cytoplasm / regulation of autophagy / viral protein processing / lyase activity / host cell endoplasmic reticulum membrane / RNA helicase / symbiont-mediated suppression of host type I interferon-mediated signaling pathway / symbiont-mediated suppression of host gene expression / copper ion binding / viral translational frameshifting / symbiont-mediated activation of host autophagy / RNA-directed RNA polymerase / cysteine-type endopeptidase activity / viral RNA genome replication / RNA-directed RNA polymerase activity / lipid binding / DNA-templated transcription / SARS-CoV-2 activates/modulates innate and adaptive immune responses / host cell nucleus / ATP hydrolysis activity / proteolysis / RNA binding / zinc ion binding / ATP binding / membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
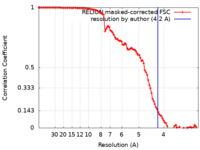
| 手法 | サブトモグラム平均法 / クライオ電子顕微鏡法 / 解像度: 4.2 Å | |||||||||
 データ登録者 データ登録者 | Huang YX / Zhong LJ / Zhang WX / Ni T | |||||||||
| 資金援助 |  香港, 1件 香港, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2024 ジャーナル: Nature / 年: 2024タイトル: Molecular architecture of coronavirus double-membrane vesicle pore complex. 著者: Yixin Huang / Tongyun Wang / Lijie Zhong / Wenxin Zhang / Yu Zhang / Xiulian Yu / Shuofeng Yuan / Tao Ni /  要旨: Coronaviruses remodel the intracellular host membranes during replication, forming double-membrane vesicles (DMVs) to accommodate viral RNA synthesis and modifications. SARS-CoV-2 non-structural ...Coronaviruses remodel the intracellular host membranes during replication, forming double-membrane vesicles (DMVs) to accommodate viral RNA synthesis and modifications. SARS-CoV-2 non-structural protein 3 (nsp3) and nsp4 are the minimal viral components required to induce DMV formation and to form a double-membrane-spanning pore, essential for the transport of newly synthesized viral RNAs. The mechanism of DMV pore complex formation remains unknown. Here we describe the molecular architecture of the SARS-CoV-2 nsp3-nsp4 pore complex, as resolved by cryogenic electron tomography and subtomogram averaging in isolated DMVs. The structures uncover an unexpected stoichiometry and topology of the nsp3-nsp4 pore complex comprising 12 copies each of nsp3 and nsp4, organized in 4 concentric stacking hexamer rings, mimicking a miniature nuclear pore complex. The transmembrane domains are interdigitated to create a high local curvature at the double-membrane junction, coupling double-membrane reorganization with pore formation. The ectodomains form extensive contacts in a pseudo-12-fold symmetry, belting the pore complex from the intermembrane space. A central positively charged ring of arginine residues coordinates the putative RNA translocation, essential for virus replication. Our work establishes a framework for understanding DMV pore formation and RNA translocation, providing a structural basis for the development of new antiviral strategies to combat coronavirus infection. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_39109.map.gz emd_39109.map.gz | 51.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-39109-v30.xml emd-39109-v30.xml emd-39109.xml emd-39109.xml | 21.2 KB 21.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_39109_fsc.xml emd_39109_fsc.xml | 10.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_39109.png emd_39109.png | 117.9 KB | ||
| マスクデータ |  emd_39109_msk_1.map emd_39109_msk_1.map | 91.1 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-39109.cif.gz emd-39109.cif.gz | 7.8 KB | ||
| その他 |  emd_39109_half_map_1.map.gz emd_39109_half_map_1.map.gz emd_39109_half_map_2.map.gz emd_39109_half_map_2.map.gz | 43.4 MB 43.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-39109 http://ftp.pdbj.org/pub/emdb/structures/EMD-39109 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-39109 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-39109 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_39109_validation.pdf.gz emd_39109_validation.pdf.gz | 846.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_39109_full_validation.pdf.gz emd_39109_full_validation.pdf.gz | 846 KB | 表示 | |
| XML形式データ |  emd_39109_validation.xml.gz emd_39109_validation.xml.gz | 17.7 KB | 表示 | |
| CIF形式データ |  emd_39109_validation.cif.gz emd_39109_validation.cif.gz | 23.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-39109 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-39109 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-39109 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-39109 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8yb5MC  8yaxC  8yb7C M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_39109.map.gz / 形式: CCP4 / 大きさ: 91.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_39109.map.gz / 形式: CCP4 / 大きさ: 91.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | local resolution map | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.571 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_39109_msk_1.map emd_39109_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_39109_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_39109_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : SARS-CoV-2 DMV nsp3-4 pore complex
| 全体 | 名称: SARS-CoV-2 DMV nsp3-4 pore complex |
|---|---|
| 要素 |
|
-超分子 #1: SARS-CoV-2 DMV nsp3-4 pore complex
| 超分子 | 名称: SARS-CoV-2 DMV nsp3-4 pore complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Papain-like protease nsp3
| 分子 | 名称: Papain-like protease nsp3 / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO / EC番号: ubiquitinyl hydrolase 1 |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 217.471188 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: APTKVTFGDD TVIEVQGYKS VNITFELDER IDKVLNEKCS AYTVELGTEV NEFACVVADA VIKTLQPVSE LLTPLGIDLD EWSMATYYL FDESGEFKLA SHMYCSFYPP DEDEEEGDCE EEEFEPSTQY EYGTEDDYQG KPLEFGATSA ALQPEEEQEE D WLDDDSQQ ...文字列: APTKVTFGDD TVIEVQGYKS VNITFELDER IDKVLNEKCS AYTVELGTEV NEFACVVADA VIKTLQPVSE LLTPLGIDLD EWSMATYYL FDESGEFKLA SHMYCSFYPP DEDEEEGDCE EEEFEPSTQY EYGTEDDYQG KPLEFGATSA ALQPEEEQEE D WLDDDSQQ TVGQQDGSED NQTTTIQTIV EVQPQLEMEL TPVVQTIEVN SFSGYLKLTD NVYIKNADIV EEAKKVKPTV VV NAANVYL KHGGGVAGAL NKATNNAMQV ESDDYIATNG PLKVGGSCVL SGHNLAKHCL HVVGPNVNKG EDIQLLKSAY ENF NQHEVL LAPLLSAGIF GADPIHSLRV CVDTVRTNVY LAVFDKNLYD KLVSSFLEMK SEKQVEQKIA EIPKEEVKPF ITES KPSVE QRKQDDKKIK ACVEEVTTTL EETKFLTENL LLYIDINGNL HPDSATLVSD IDITFLKKDA PYIVGDVVQE GVLTA VVIP TKKAGGTTEM LAKALRKVPT DNYITTYPGQ GLNGYTVEEA KTVLKKCKSA FYILPSIISN EKQEILGTVS WNLREM LAH AEETRKLMPV CVETKAIVST IQRKYKGIKI QEGVVDYGAR FYFYTSKTTV ASLINTLNDL NETLVTMPLG YVTHGLN LE EAARYMRSLK VPATVSVSSP DAVTAYNGYL TSSSKTPEEH FIETISLAGS YKDWSYSGQS TQLGIEFLKR GDKSVYYT S NPTTFHLDGE VITFDNLKTL LSLREVRTIK VFTTVDNINL HTQVVDMSMT YGQQFGPTYL DGADVTKIKP HNSHEGKTF YVLPNDDTLR VEAFEYYHTT DPSFLGRYMS ALNHTKKWKY PQVNGLTSIK WADNNCYLAT ALLTLQQIEL KFNPPALQDA YYRARAGEA ANFCALILAY CNKTVGELGD VRETMSYLFQ HANLDSCKRV LNVVCKTCGQ QQTTLKGVEA VMYMGTLSYE Q FKKGVQIP CTCGKQATKY LVQQESPFVM MSAPPAQYEL KHGTFTCASE YTGNYQCGHY KHITSKETLY CIDGALLTKS SE YKGPITD VFYKENSYTT TIKPVTYKLD GVVCTEIDPK LDNYYKKDNS YFTEQPIDLV PNQPYPNASF DNFKFVCDNI KFA DDLNQL TGYKKPASRE LKVTFFPDLN GDVVAIDYKH YTPSFKKGAK LLHKPIVWHV NNATNKATYK PNTWCIRCLW STKP VETSN SFDVLKSEDA QGMDNLACED LKPVSEEVVE NPTIQKDVLE CNVKTTEVVG DIILKPANNS LKITEEVGHT DLMAA YVDN SSLTIKKPNE LSRVLGLKTL ATHGLAAVNS VPWDTIANYA KPFLNKVVST TTNIVTRCLN RVCTNYMPYF FTLLLQ LCT FTRSTNSRIK ASMPTTIAKN TVKSVGKFCL EASFNYLKSP NFSKLINIII WFLLLSVCLG SLIYSTAALG VLMSNLG MP SYCTGYREGY LNSTNVTIAT YCTGSIPCSV CLSGLDSLDT YPSLETIQIT ISSFKWDLTA FGLVAEWFLA YILFTRFF Y VLGLAAIMQL FFSYFAVHFI SNSWLMWLII NLVQMAPISA MVRMYIFFAS FYYVWKSYVH VVDGCNSSTC MMCYKRNRA TRVECTTIVN GVRRSFYVYA NGGKGFCKLH NWNCVNCDTF CAGSTFISDE VARDLSLQFK RPINPTDQSS YIVDSVTVKN GSIHLYFDK AGQKTYERHS LSHFVNLDNL RANNTKGSLP INVIVFDGKS KCEESSAKSA SVYYSQLMCQ PILLLDQALV S DVGDSAEV AVKMFDAYVN TFSSTFNVPM EKLKTLVATA EAELAKNVSL DNVLSTFISA ARQGFVDSDV ETKDVVECLK LS HQSDIEV TGDSCNNYML TYNKVENMTP RDLGACIDCS ARHINAQVAK SHNIALIWNV KDFMSLSEQL RKQIRSAAKK NNL PFKLTC ATTRQVVNVV TTKIALKGG UniProtKB: Replicase polyprotein 1ab |
-分子 #2: Non-structural protein 4
| 分子 | 名称: Non-structural protein 4 / タイプ: protein_or_peptide / ID: 2 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 56.229582 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: KIVNNWLKQL IKVTLVFLFV AAIFYLITPV HVMSKHTDFS SEIIGYKAID GGVTRDIAST DTCFANKHAD FDTWFSQRGG SYTNDKACP LIAAVITREV GFVVPGLPGT ILRTTNGDFL HFLPRVFSAV GNICYTPSKL IEYTDFATSA CVLAAECTIF K DASGKPVP ...文字列: KIVNNWLKQL IKVTLVFLFV AAIFYLITPV HVMSKHTDFS SEIIGYKAID GGVTRDIAST DTCFANKHAD FDTWFSQRGG SYTNDKACP LIAAVITREV GFVVPGLPGT ILRTTNGDFL HFLPRVFSAV GNICYTPSKL IEYTDFATSA CVLAAECTIF K DASGKPVP YCYDTNVLEG SVAYESLRPD TRYVLMDGSI IQFPNTYLEG SVRVVTTFDS EYCRHGTCER SEAGVCVSTS GR WVLNNDY YRSLPGVFCG VDAVNLLTNM FTPLIQPIGA LDISASIVAG GIVAIVVTCL AYYFMRFRRA FGEYSHVVAF NTL LFLMSF TVLCLTPVYS FLPGVYSVIY LYLTFYLTND VSFLAHIQWM VMFTPLVPFW ITIAYIICIS TKHFYWFFSN YLKR RVVFN GVSFSTFEEA ALCTFLLNKE MYLKLRSDVL LPLTQYNRYL ALYNKYKYFS GAMDTTSYRE AACCHLAKAL NDFSN SGSD VLYQPPQTSI TSAVLQ UniProtKB: Replicase polyprotein 1ab |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | サブトモグラム平均法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 構成要素:
詳細: 150mM NaCl, 10mM Tris-HCl, 1mM EDTA | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| グリッド | モデル: EMS Lacey Carbon / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 45 sec. / 前処理 - 雰囲気: AIR | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / チャンバー内温度: 277.15 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| ソフトウェア | 名称: SerialEM |
| 撮影 | フィルム・検出器のモデル: TFS FALCON 4i (4k x 4k) 平均電子線量: 3.0 e/Å2 詳細: The tilt-series were acquired using Thermofisher Krios equipped with a Falcon 4i camera and Selectris energy filter. A dose-symmetric scheme (group of 2) was used, with a tilt range of -51 ...詳細: The tilt-series were acquired using Thermofisher Krios equipped with a Falcon 4i camera and Selectris energy filter. A dose-symmetric scheme (group of 2) was used, with a tilt range of -51 degree to 51 degree (or -60 to 60) at 3 degree increments and an exposure dose of 3 e-/A2 per image. The total dose was 105 or 123 e-/A2. |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 6.0 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 81000 |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | Chain - Source name: AlphaFold / Chain - Initial model type: in silico model |
|---|---|
| ソフトウェア | 名称:  UCSF Chimera (ver. 1.16) UCSF Chimera (ver. 1.16) |
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT |
| 得られたモデル |  PDB-8yb5: |
 ムービー
ムービー コントローラー
コントローラー

















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)