+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-3907 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | The electron crystallography structure of the cAMP-bound potassium channel MloK1 (PCO-refined) | |||||||||
 マップデータ マップデータ | PCO (Projective ConstraintOptimization) - refined 3D volume of MloK1 with cAMP | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | MloK1 / MlotiK1 / potassium channel / CNBD / cytoplasmic domains / PCO refinement / MEMBRANE PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報intracellularly cyclic nucleotide-activated monoatomic cation channel activity / potassium channel activity / cAMP binding / protein-containing complex binding / identical protein binding / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  Mesorhizobium loti (根粒菌) / Mesorhizobium loti (根粒菌) /  Mesorhizobium loti MAFF303099 (根粒菌) Mesorhizobium loti MAFF303099 (根粒菌) | |||||||||
| 手法 | 電子線結晶学 / クライオ電子顕微鏡法 / 解像度: 4.5 Å | |||||||||
 データ登録者 データ登録者 | Kowal J / Biyani N | |||||||||
| 資金援助 |  スイス, 1件 スイス, 1件
| |||||||||
 引用 引用 |  ジャーナル: Structure / 年: 2018 ジャーナル: Structure / 年: 2018タイトル: High-Resolution Cryoelectron Microscopy Structure of the Cyclic Nucleotide-Modulated Potassium Channel MloK1 in a Lipid Bilayer. 著者: Julia Kowal / Nikhil Biyani / Mohamed Chami / Sebastian Scherer / Andrzej J Rzepiela / Paul Baumgartner / Vikrant Upadhyay / Crina M Nimigean / Henning Stahlberg /   要旨: Eukaryotic cyclic nucleotide-modulated channels perform their diverse physiological roles by opening and closing their pores to ions in response to cyclic nucleotide binding. We here present a ...Eukaryotic cyclic nucleotide-modulated channels perform their diverse physiological roles by opening and closing their pores to ions in response to cyclic nucleotide binding. We here present a structural model for the cyclic nucleotide-modulated potassium channel homolog from Mesorhizobium loti, MloK1, determined from 2D crystals in the presence of lipids. Even though crystals diffract electrons to only ∼10 Å, using cryoelectron microscopy (cryo-EM) and recently developed computational methods, we have determined a 3D map of full-length MloK1 in the presence of cyclic AMP (cAMP) at ∼4.5 Å isotropic 3D resolution. The structure provides a clear picture of the arrangement of the cyclic nucleotide-binding domains with respect to both the pore and the putative voltage sensor domains when cAMP is bound, and reveals a potential gating mechanism in the context of the lipid-embedded channel. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_3907.map.gz emd_3907.map.gz | 8.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-3907-v30.xml emd-3907-v30.xml emd-3907.xml emd-3907.xml | 19.8 KB 19.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_3907_1.png emd_3907_1.png emd_3907_2.png emd_3907_2.png | 207.5 KB 184.7 KB | ||
| Filedesc metadata |  emd-3907.cif.gz emd-3907.cif.gz | 7.2 KB | ||
| その他 |  emd_3907_additional.map.gz emd_3907_additional.map.gz | 6.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-3907 http://ftp.pdbj.org/pub/emdb/structures/EMD-3907 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3907 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3907 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_3907_validation.pdf.gz emd_3907_validation.pdf.gz | 551.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_3907_full_validation.pdf.gz emd_3907_full_validation.pdf.gz | 551.4 KB | 表示 | |
| XML形式データ |  emd_3907_validation.xml.gz emd_3907_validation.xml.gz | 5 KB | 表示 | |
| CIF形式データ |  emd_3907_validation.cif.gz emd_3907_validation.cif.gz | 5.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3907 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3907 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3907 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3907 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  6eo1MC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_3907.map.gz / 形式: CCP4 / 大きさ: 9.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_3907.map.gz / 形式: CCP4 / 大きさ: 9.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | PCO (Projective ConstraintOptimization) - refined 3D volume of MloK1 with cAMP | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 これらの図は立方格子座標系で作成されたものです | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X: 0.955 Å / Y: 0.955 Å / Z: 1.045 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
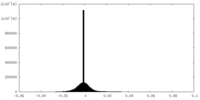
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: Back-projected 3D volume of MloK1 with cAMP
| ファイル | emd_3907_additional.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Back-projected 3D volume of MloK1 with cAMP | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
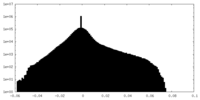
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : MloK1 tetramer
| 全体 | 名称: MloK1 tetramer |
|---|---|
| 要素 |
|
-超分子 #1: MloK1 tetramer
| 超分子 | 名称: MloK1 tetramer / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 詳細: Cyclic nucleotide-modulated potassium channel in the presence of cAMP ligand, reconstituted into 2D lipid membrane crystals. |
|---|---|
| 由来(天然) | 生物種:  Mesorhizobium loti (根粒菌) / 器官: Membrane Mesorhizobium loti (根粒菌) / 器官: Membrane |
| 分子量 | 理論値: 148 KDa |
-分子 #1: Cyclic nucleotide-gated potassium channel mll3241
| 分子 | 名称: Cyclic nucleotide-gated potassium channel mll3241 / タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Mesorhizobium loti MAFF303099 (根粒菌) / 器官: Membrane Mesorhizobium loti MAFF303099 (根粒菌) / 器官: Membrane |
| 分子量 | 理論値: 37.766297 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSVLPFLRIY APLNAVLAAP GLLAVAALTI PDMSGRSRLA LAALLAVIWG AYLLQLAATL LKRRAGVVRD RTPKIAIDVL AVLVPLAAF LLDGSPDWSL YCAVWLLKPL RDSTFFPVLG RVLANEARNL IGVTTLFGVV LFAVALAAYV IERDIQPEKF G SIPQAMWW ...文字列: MSVLPFLRIY APLNAVLAAP GLLAVAALTI PDMSGRSRLA LAALLAVIWG AYLLQLAATL LKRRAGVVRD RTPKIAIDVL AVLVPLAAF LLDGSPDWSL YCAVWLLKPL RDSTFFPVLG RVLANEARNL IGVTTLFGVV LFAVALAAYV IERDIQPEKF G SIPQAMWW AVVTLSTTGY GDTIPQSFAG RVLAGAVMMS GIGIFGLWAG ILATGFYQEV RRGDFVRNWQ LVAAVPLFQK LG PAVLVEI VRALRARTVP AGAVICRIGE PGDRMFFVVE GSVSVATPNP VELGPGAFFG EMALISGEPR SATVSAATTV SLL SLHSAD FQMLCSSSPE IAEIFRKTAL ERRGAAASA UniProtKB: Cyclic nucleotide-gated potassium channel mll3241 |
-分子 #2: POTASSIUM ION
| 分子 | 名称: POTASSIUM ION / タイプ: ligand / ID: 2 / コピー数: 2 / 式: K |
|---|---|
| 分子量 | 理論値: 39.098 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 電子線結晶学 |
| 試料の集合状態 | 2D array |
- 試料調製
試料調製
| 濃度 | 0.7 mg/mL |
|---|---|
| 緩衝液 | pH: 7.6 詳細: 20 mM KCl, 20 mM Tris-HCl pH 7.6, 1 mM BaCl2, 1 mM EDTA, 0.2 mM cAMP |
| グリッド | モデル: Quantifoil R3.5/1 / 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: CONTINUOUS / 支持フィルム - Film thickness: 3 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. / 前処理 - 雰囲気: AIR |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 90 % / チャンバー内温度: 293 K / 装置: FEI VITROBOT MARK IV / 詳細: 3.5 second-blotting. |
| 結晶化 | 脂質・タンパク質比: 0.8 / 脂質混合液: E.coli polar lipids / 装置: dialysis buttons / 雰囲気: dialysis buffer / 温度: 293.0 K / 時間: 5.0 DAY 詳細: DM solubilized MloK1 sample was mixed with E. coli polar lipid extract (Avanti Polar Lipids) at a lipid to protein ratio of 0.8 and dialyzed against detergent free buffer. 2D crystals of the ...詳細: DM solubilized MloK1 sample was mixed with E. coli polar lipid extract (Avanti Polar Lipids) at a lipid to protein ratio of 0.8 and dialyzed against detergent free buffer. 2D crystals of the lipid embedded protein were obtained within 5 days. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Quantum LS エネルギーフィルター - エネルギー下限: 0 eV エネルギーフィルター - エネルギー上限: 20 eV |
| 詳細 | pixel size 1.3 A/pix |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 撮影したグリッド数: 30 / 実像数: 346 / 平均露光時間: 16.0 sec. / 平均電子線量: 45.0 e/Å2 詳細: Each image was dose-fractionated in 40 frames (16 sec in total, 0.4-sec frames). The dose rate was set to ~5 counts/sec/physical-pixel (~2.8 e-/s/A2)leading to a total dose of ~45 e-/A2. Pixel size was 1.3A/pix. |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 4.3 µm / 最小 デフォーカス(公称値): 0.75 µm / 倍率(公称値): 50000 / カメラ長: 800 mm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN / 傾斜角度: 0.0, 55.0 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | PDB ID: Chain - Chain ID: A / Chain - Residue range: 1-355 / Chain - Source name: PDB / Chain - Initial model type: experimental model |
|---|---|
| 詳細 | The initial model was obtained using Modeller (Sali and Blundell, 1993); in particular, the missing fragments were generated for the previously published PDB 4CHV model. This starting model was refined using the Rosetta for cryo-EM package (DiMaio et al., 2015). The symmetry of the channel was restrained during optimization runs (performed following the package tutorial (Wang and DiMaio,2015)). A model with a high fit score to the cryo-EM map and a low energy, as defined by the Rosetta force field, was selected from 100 Rosetta models generated and refined further. Several rounds of manual refinement with Coot (Emsley et al.,2010) and global optimization with Phenix (real_space_refine method (Afonine et al., 2013)) were carried out. Secondary structure constraints were imposed to stabilize the fold of helices and b-sheets during the global optimization. |
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT / 当てはまり具合の基準: fit energy |
| 得られたモデル |  PDB-6eo1: |
 ムービー
ムービー コントローラー
コントローラー







 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)