+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of ATP-DNA-MuB filaments | |||||||||
 マップデータ マップデータ | CryoEM structure of ATP-DNA-MuB Filaments in Bacteriophage Mu DNA Transposition | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | VIRAL PROTEIN-DNA COMPLEX | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報DNA transposition / viral DNA genome replication / 加水分解酵素; 酸無水物に作用; リン含有酸無水物に作用 / DNA integration / host cell cytoplasm / DNA replication / ATP hydrolysis activity / DNA binding / ATP binding / metal ion binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  Escherichia phage Mu (ファージ) Escherichia phage Mu (ファージ) | |||||||||
| 手法 | らせん対称体再構成法 / クライオ電子顕微鏡法 / 解像度: 3.4 Å | |||||||||
 データ登録者 データ登録者 | Zhao X / Zhang K / Li S | |||||||||
| 資金援助 | 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2024 ジャーナル: Nat Commun / 年: 2024タイトル: Elucidating the Architectural dynamics of MuB filaments in bacteriophage Mu DNA transposition. 著者: Xiaolong Zhao / Yongxiang Gao / Qingguo Gong / Kaiming Zhang / Shanshan Li /  要旨: MuB is a non-specific DNA-binding protein and AAA+ ATPase that significantly influences the DNA transposition process of bacteriophage Mu, especially in target DNA selection for transposition. While ...MuB is a non-specific DNA-binding protein and AAA+ ATPase that significantly influences the DNA transposition process of bacteriophage Mu, especially in target DNA selection for transposition. While studies have established the ATP-dependent formation of MuB filament as pivotal to this process, the high-resolution structure of a full-length MuB protomer and the underlying molecular mechanisms governing its oligomerization remain elusive. Here, we use cryo-EM to obtain a 3.4-Å resolution structure of the ATP(+)-DNA(+)-MuB helical filament, which encapsulates the DNA substrate within its axial channel. The structure categorizes MuB within the initiator clade of the AAA+ protein family and precisely locates the ATP and DNA binding sites. Further investigation into the oligomeric states of MuB show the existence of various forms of the filament. These findings lead to a mechanistic model where MuB forms opposite helical filaments along the DNA, exposing potential target sites on the bare DNA and then recruiting MuA, which stimulates MuB's ATPase activity and disrupts the previously formed helical structure. When this happens, MuB generates larger ring structures and dissociates from the DNA. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_38695.map.gz emd_38695.map.gz | 70.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-38695-v30.xml emd-38695-v30.xml emd-38695.xml emd-38695.xml | 20.5 KB 20.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_38695.png emd_38695.png | 61.8 KB | ||
| Filedesc metadata |  emd-38695.cif.gz emd-38695.cif.gz | 6.1 KB | ||
| その他 |  emd_38695_additional_1.map.gz emd_38695_additional_1.map.gz emd_38695_half_map_1.map.gz emd_38695_half_map_1.map.gz emd_38695_half_map_2.map.gz emd_38695_half_map_2.map.gz | 136.5 MB 134.3 MB 134.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-38695 http://ftp.pdbj.org/pub/emdb/structures/EMD-38695 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-38695 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-38695 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_38695_validation.pdf.gz emd_38695_validation.pdf.gz | 1.1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_38695_full_validation.pdf.gz emd_38695_full_validation.pdf.gz | 1.1 MB | 表示 | |
| XML形式データ |  emd_38695_validation.xml.gz emd_38695_validation.xml.gz | 14.5 KB | 表示 | |
| CIF形式データ |  emd_38695_validation.cif.gz emd_38695_validation.cif.gz | 17.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-38695 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-38695 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-38695 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-38695 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8xvbMC  8xvcC  8xvdC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_38695.map.gz / 形式: CCP4 / 大きさ: 144.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_38695.map.gz / 形式: CCP4 / 大きさ: 144.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | CryoEM structure of ATP-DNA-MuB Filaments in Bacteriophage Mu DNA Transposition | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.82 Å | ||||||||||||||||||||||||||||||||||||
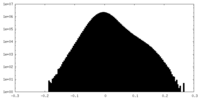
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: CryoEM structure of ATP-DNA-MuB Filaments in Bacteriophage Mu...
| ファイル | emd_38695_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | CryoEM structure of ATP-DNA-MuB Filaments in Bacteriophage Mu DNA Transposition | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
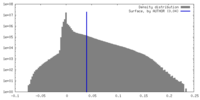
| 密度ヒストグラム |
-ハーフマップ: CryoEM structure of ATP-DNA-MuB Filaments in Bacteriophage Mu...
| ファイル | emd_38695_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | CryoEM structure of ATP-DNA-MuB Filaments in Bacteriophage Mu DNA Transposition | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
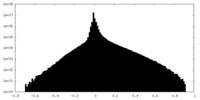
| 密度ヒストグラム |
-ハーフマップ: CryoEM structure of ATP-DNA-MuB Filaments in Bacteriophage Mu...
| ファイル | emd_38695_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | CryoEM structure of ATP-DNA-MuB Filaments in Bacteriophage Mu DNA Transposition | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
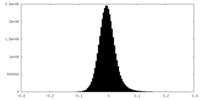
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : ATP-DNA-MuB Filaments in Bacteriophage Mu DNA Transposition
| 全体 | 名称: ATP-DNA-MuB Filaments in Bacteriophage Mu DNA Transposition |
|---|---|
| 要素 |
|
-超分子 #1: ATP-DNA-MuB Filaments in Bacteriophage Mu DNA Transposition
| 超分子 | 名称: ATP-DNA-MuB Filaments in Bacteriophage Mu DNA Transposition タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#3 |
|---|---|
| 由来(天然) | 生物種:  Escherichia phage Mu (ファージ) Escherichia phage Mu (ファージ) |
-分子 #1: DNA (5'-D(P*AP*AP*AP*AP*AP*AP*AP*AP*AP*AP*AP*AP*AP*AP*AP*AP*AP*AP...
| 分子 | 名称: DNA (5'-D(P*AP*AP*AP*AP*AP*AP*AP*AP*AP*AP*AP*AP*AP*AP*AP*AP*AP*AP*AP*AP*AP*AP*AP*A)-3') タイプ: dna / ID: 1 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:  Escherichia phage Mu (ファージ) Escherichia phage Mu (ファージ) |
| 分子量 | 理論値: 7.472007 KDa |
| 配列 | 文字列: (DA)(DA)(DA)(DA)(DA)(DA)(DA)(DA)(DA)(DA) (DA)(DA)(DA)(DA)(DA)(DA)(DA)(DA)(DA)(DA) (DA)(DA)(DA)(DA) |
-分子 #3: DNA (5'-D(P*TP*TP*TP*TP*TP*TP*TP*TP*TP*TP*TP*TP*TP*TP*TP*TP*TP*TP...
| 分子 | 名称: DNA (5'-D(P*TP*TP*TP*TP*TP*TP*TP*TP*TP*TP*TP*TP*TP*TP*TP*TP*TP*TP*TP*TP*TP*TP*TP*T)-3') タイプ: dna / ID: 3 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:  Escherichia phage Mu (ファージ) Escherichia phage Mu (ファージ) |
| 分子量 | 理論値: 7.25567 KDa |
| 配列 | 文字列: (DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT) (DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT) (DT)(DT)(DT)(DT) |
-分子 #2: ATP-dependent target DNA activator B
| 分子 | 名称: ATP-dependent target DNA activator B / タイプ: protein_or_peptide / ID: 2 / コピー数: 8 / 光学異性体: LEVO EC番号: 加水分解酵素; 酸無水物に作用; リン含有酸無水物に作用 |
|---|---|
| 由来(天然) | 生物種:  Escherichia phage Mu (ファージ) Escherichia phage Mu (ファージ) |
| 分子量 | 理論値: 35.153133 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MNISDIRAGL RTLVENEETT FKQIALESGL STGTISSFIN DKYNGDNERV SQMLQRWLEK YHAVAELPEP PRFVETQTVK QIWTSMRFA SLTESIAVVC GNPGVGKTEA AREYRRTNNN VWMITITPSC ASVLECLTEL AFELGMNDAP RRKGPLSRAL R RRLEGTQG ...文字列: MNISDIRAGL RTLVENEETT FKQIALESGL STGTISSFIN DKYNGDNERV SQMLQRWLEK YHAVAELPEP PRFVETQTVK QIWTSMRFA SLTESIAVVC GNPGVGKTEA AREYRRTNNN VWMITITPSC ASVLECLTEL AFELGMNDAP RRKGPLSRAL R RRLEGTQG LVIIDEADHL GAEVLEELRL LQESTRIGLV LMGNHRVYSN MTGGNRTVEF ARLFSRIAKR TAINKTKKAD VK AIADAWQ INGEKELELL QQIAQKPGAL RILNHSLRLA AMTAHGKGER VNEDYLRQAF RELDLDVDIS TLLRN UniProtKB: ATP-dependent target DNA activator B |
-分子 #4: ADENOSINE-5'-TRIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-TRIPHOSPHATE / タイプ: ligand / ID: 4 / コピー数: 8 / 式: ATP |
|---|---|
| 分子量 | 理論値: 507.181 Da |
| Chemical component information |  ChemComp-ATP: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | filament |
- 試料調製
試料調製
| 緩衝液 | pH: 7.8 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 実像数: 6298 / 平均電子線量: 60.9 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 2.0 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 最終 再構成 | 想定した対称性 - らせんパラメータ - Δz: 6.91 Å 想定した対称性 - らせんパラメータ - ΔΦ: 56.16 ° 想定した対称性 - らせんパラメータ - 軸対称性: C1 (非対称) 解像度のタイプ: BY AUTHOR / 解像度: 3.4 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 377689 |
|---|---|
| CTF補正 | タイプ: NONE |
| 初期モデル | モデルのタイプ: NONE |
| 最終 角度割当 | タイプ: NOT APPLICABLE |
 ムービー
ムービー コントローラー
コントローラー











 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)