+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | MCM in complex with dsDNA in presence of ATP. | |||||||||
 マップデータ マップデータ | MCM_ATP_dsDNA | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Helicase / Replication / HYDROLASE | |||||||||
| 生物種 |   Thermococcus kodakarensis (古細菌) Thermococcus kodakarensis (古細菌) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.57 Å | |||||||||
 データ登録者 データ登録者 | Ma J / Yi G / Ye M / MacGregor-Chatwin C / Sheng Y / Lu Y / Li M / Gilbert RJC / Zhang P | |||||||||
| 資金援助 |  英国, 2件 英国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2024 ジャーナル: Nat Commun / 年: 2024タイトル: Open architecture of archaea MCM and dsDNA complexes resolved using monodispersed streptavidin affinity CryoEM. 著者: Jianbing Ma / Gangshun Yi / Mingda Ye / Craig MacGregor-Chatwin / Yuewen Sheng / Ying Lu / Ming Li / Qingrong Li / Dong Wang / Robert J C Gilbert / Peijun Zhang /    要旨: The cryo-electron microscopy (cryoEM) method has enabled high-resolution structure determination of numerous biomolecules and complexes. Nevertheless, cryoEM sample preparation of challenging ...The cryo-electron microscopy (cryoEM) method has enabled high-resolution structure determination of numerous biomolecules and complexes. Nevertheless, cryoEM sample preparation of challenging proteins and complexes, especially those with low abundance or with preferential orientation, remains a major hurdle. We developed an affinity-grid method employing monodispersed single particle streptavidin on a lipid monolayer to enhance particle absorption on the grid surface and alleviate sample exposure to the air-water interface. Using this approach, we successfully enriched the Thermococcus kodakarensis mini-chromosome maintenance complex 3 (MCM3) on cryoEM grids through biotinylation and resolved its structure. We further utilized this affinity method to tether the biotin-tagged dsDNA to selectively enrich a stable MCM3-ATP-dsDNA complex for cryoEM structure determination. Intriguingly, both MCM3 apo and dsDNA bound structures exhibit left-handed open spiral conformations, distinct from other reported MCM structures. The large open gate is sufficient to accommodate a dsDNA which could potentially be melted. The value of mspSA affinity method was further demonstrated by mitigating the issue of preferential angular distribution of HIV-1 capsid protein hexamer and RNA polymerase II elongation complex from Saccharomyces cerevisiae. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
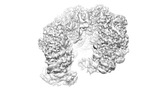
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_38111.map.gz emd_38111.map.gz | 11 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-38111-v30.xml emd-38111-v30.xml emd-38111.xml emd-38111.xml | 13.4 KB 13.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_38111.png emd_38111.png | 53.5 KB | ||
| Filedesc metadata |  emd-38111.cif.gz emd-38111.cif.gz | 5.9 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-38111 http://ftp.pdbj.org/pub/emdb/structures/EMD-38111 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-38111 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-38111 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_38111_validation.pdf.gz emd_38111_validation.pdf.gz | 372.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_38111_full_validation.pdf.gz emd_38111_full_validation.pdf.gz | 372.5 KB | 表示 | |
| XML形式データ |  emd_38111_validation.xml.gz emd_38111_validation.xml.gz | 7 KB | 表示 | |
| CIF形式データ |  emd_38111_validation.cif.gz emd_38111_validation.cif.gz | 8.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-38111 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-38111 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-38111 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-38111 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_38111.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_38111.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | MCM_ATP_dsDNA | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.932 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : MCM homohexamer in complex with dsDNA in presence of ATP
| 全体 | 名称: MCM homohexamer in complex with dsDNA in presence of ATP |
|---|---|
| 要素 |
|
-超分子 #1: MCM homohexamer in complex with dsDNA in presence of ATP
| 超分子 | 名称: MCM homohexamer in complex with dsDNA in presence of ATP タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:   Thermococcus kodakarensis (古細菌) Thermococcus kodakarensis (古細菌) |
-分子 #1: mini-chromosome maintenance complex 3
| 分子 | 名称: mini-chromosome maintenance complex 3 / タイプ: protein_or_peptide / ID: 1 / コピー数: 6 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Thermococcus kodakarensis (古細菌) Thermococcus kodakarensis (古細菌) |
| 分子量 | 理論値: 76.631242 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MDREEMIARF AKFLREYVDD EGNEVYINRL KDLLTVTPKR SLAIDWAHLN SFDPELADEL LNNPEEAIAS AEDAIQIVLR EPPLLVERE FKVHARFYNL PKTLLVKELG SEHINKLIQV EGIITRVSEV KPFVEKAVFV CRDCGNEMVR LQRPYENLVK P AKCDACGS ...文字列: MDREEMIARF AKFLREYVDD EGNEVYINRL KDLLTVTPKR SLAIDWAHLN SFDPELADEL LNNPEEAIAS AEDAIQIVLR EPPLLVERE FKVHARFYNL PKTLLVKELG SEHINKLIQV EGIITRVSEV KPFVEKAVFV CRDCGNEMVR LQRPYENLVK P AKCDACGS RNIELDVDKS RFLNFQSFRL QDRPESLKGG QMPRFVDAIL LDDLVDAALP GDRVLVTGVL RVILEQREKR PI FKKILEV NHIEQLSKEI EELEISPEDE QKIRELAKRK DIVDAIVDSI APAIWGHRIV KKGIALALFG GVQRTLPDGT KLR GESHVL LVGDPGVAKS QLLRYVANLA PRAIYTSGKS SSAAGLTAAA VRDEFTGSWV LEAGVLVLAD GGFALIDEFD KMSD RDRSA IHEALEQQTI SISKAGITAT LNSRTTVIAA ANPKFGRFNR HKSLPEQLDL PPTLLSRFDL IFLLLDEPDE KVDAS IAEH ILKVRRGEAE AVTPKIPYDL LKKYIAYARK NVHPVLSREA MEEIKRYYVK MRKGLRRGDE DGVQPIPITA RQLEAL IRL SEAHARMRLS ETVTREDARA AIEIIEAMMK TIAVDEEGNL DVSILEVGKS SKKINKIEKL VDIIKSLESE GEFGAPE EK VIEAAKQAGI GTKADIEKLL NELKSDGRVY EPRAGFYRVI |
-分子 #2: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 2 / コピー数: 3 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-分子 #3: ADENOSINE-5'-TRIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-TRIPHOSPHATE / タイプ: ligand / ID: 3 / コピー数: 2 / 式: ATP |
|---|---|
| 分子量 | 理論値: 507.181 Da |
| Chemical component information |  ChemComp-ATP: |
-分子 #4: ADENOSINE-5'-DIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-DIPHOSPHATE / タイプ: ligand / ID: 4 / コピー数: 3 / 式: ADP |
|---|---|
| 分子量 | 理論値: 427.201 Da |
| Chemical component information |  ChemComp-ADP: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.8 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) 平均電子線量: 39.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 0.5 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: OTHER 詳細: Generated using ab-initio reconstruction routine in cryoSPARC |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 3.57 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 126808 |
| 初期 角度割当 | タイプ: RANDOM ASSIGNMENT |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
 ムービー
ムービー コントローラー
コントローラー



























 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)




















