+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of VP9 in Banna virus | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | receptor binding / spike / reovirus / VIRAL PROTEIN | |||||||||
| 機能・相同性 | Outer capsid protein VP9/VP10/VP11 / Outer capsid protein VP9, N-terminal / Reoviridae VP9 / identical protein binding / VP9 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Banna virus (ウイルス) Banna virus (ウイルス) | |||||||||
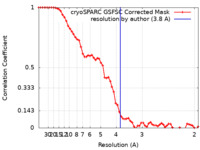
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.8 Å | |||||||||
 データ登録者 データ登録者 | Li Z / Cao S | |||||||||
| 資金援助 |  中国, 1件 中国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2024 ジャーナル: Nat Commun / 年: 2024タイトル: Cryo-EM structures of Banna virus in multiple states reveal stepwise detachment of viral spikes. 著者: Zhiqiang Li / Han Xia / Guibo Rao / Yan Fu / Tingting Chong / Kexing Tian / Zhiming Yuan / Sheng Cao /  要旨: Banna virus (BAV) is the prototype Seadornavirus, a class of reoviruses for which there has been little structural study. Here, we report atomic cryo-EM structures of three states of BAV virions- ...Banna virus (BAV) is the prototype Seadornavirus, a class of reoviruses for which there has been little structural study. Here, we report atomic cryo-EM structures of three states of BAV virions-surrounded by 120 spikes (full virions), 60 spikes (partial virions), or no spikes (cores). BAV cores are double-layered particles similar to the cores of other non-turreted reoviruses, except for an additional protein component in the outer capsid shell, VP10. VP10 was identified to be a cementing protein that plays a pivotal role in the assembly of BAV virions by directly interacting with VP2 (inner capsid), VP8 (outer capsid), and VP4 (spike). Viral spikes (VP4/VP9 heterohexamers) are situated on top of VP10 molecules in full or partial virions. Asymmetrical electrostatic interactions between VP10 monomers and VP4 trimers are disrupted by high pH treatment, which is thus a simple way to produce BAV cores. Low pH treatment of BAV virions removes only the flexible receptor binding protein VP9 and triggers significant conformational changes in the membrane penetration protein VP4. BAV virions adopt distinct spatial organization of their surface proteins compared with other well-studied reoviruses, suggesting that BAV may have a unique mechanism of penetration of cellular endomembranes. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_36872.map.gz emd_36872.map.gz | 20.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-36872-v30.xml emd-36872-v30.xml emd-36872.xml emd-36872.xml | 16.2 KB 16.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_36872_fsc.xml emd_36872_fsc.xml | 5.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_36872.png emd_36872.png | 82.9 KB | ||
| Filedesc metadata |  emd-36872.cif.gz emd-36872.cif.gz | 5.6 KB | ||
| その他 |  emd_36872_half_map_1.map.gz emd_36872_half_map_1.map.gz emd_36872_half_map_2.map.gz emd_36872_half_map_2.map.gz | 20.6 MB 20.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-36872 http://ftp.pdbj.org/pub/emdb/structures/EMD-36872 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-36872 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-36872 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_36872_validation.pdf.gz emd_36872_validation.pdf.gz | 908.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_36872_full_validation.pdf.gz emd_36872_full_validation.pdf.gz | 908.3 KB | 表示 | |
| XML形式データ |  emd_36872_validation.xml.gz emd_36872_validation.xml.gz | 13.2 KB | 表示 | |
| CIF形式データ |  emd_36872_validation.cif.gz emd_36872_validation.cif.gz | 17 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-36872 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-36872 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-36872 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-36872 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8k44MC  8k42C  8k43C  8k49C  8k4aC  8w9pC  8w9qC  8w9rC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_36872.map.gz / 形式: CCP4 / 大きさ: 22.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_36872.map.gz / 形式: CCP4 / 大きさ: 22.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.95 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_36872_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_36872_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Banna virus
| 全体 | 名称:  Banna virus (ウイルス) Banna virus (ウイルス) |
|---|---|
| 要素 |
|
-超分子 #1: Banna virus
| 超分子 | 名称: Banna virus / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: all / NCBI-ID: 77763 / 生物種: Banna virus / Sci species strain: YN15-126-01 / ウイルスタイプ: VIRION / ウイルス・単離状態: SPECIES / ウイルス・エンベロープ: No / ウイルス・中空状態: No |
|---|
-分子 #1: VP9
| 分子 | 名称: VP9 / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Banna virus (ウイルス) Banna virus (ウイルス) |
| 分子量 | 理論値: 30.581178 KDa |
| 配列 | 文字列: MLSETELRAL KKLSTTTSRV VGDSTLALPS NVKLSKGEVE KIAVTKKEMF DELAQCNLPT IELITREHTF NGDVIRFAAW LFLMNGQKL MIANNVAVRM GMQYATNLAG NNVKITYVTS NNVVKLGHIA AGVLANPYSN KGSGLFITYE YNLISNLIET G KVCVLFIT ...文字列: MLSETELRAL KKLSTTTSRV VGDSTLALPS NVKLSKGEVE KIAVTKKEMF DELAQCNLPT IELITREHTF NGDVIRFAAW LFLMNGQKL MIANNVAVRM GMQYATNLAG NNVKITYVTS NNVVKLGHIA AGVLANPYSN KGSGLFITYE YNLISNLIET G KVCVLFIT SLSTTASSTN SFAYSTCSVP IENWDFNMIK LTAETSCASL TAMTNLVNSL VPGERTRPVG LYVDIPGVTV TT SASSGSL PLTTIPAVTP LIFSAYTKQV EEVGVINTLY ALSYLP UniProtKB: VP9 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 289 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | JEOL CRYO ARM 300 |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 40.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 0.5 µm |
 ムービー
ムービー コントローラー
コントローラー











 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)